从0开始小花教你利用ggplot2绘制PCA图!!!
点击蓝字 关注我们
小伙伴在整理数据时候,由于分组较多,不知道怎么样去说明分组的差异,这个时候就会用到主成分分析(PCA).
小花先带大家了解一下PCA,其实PCA是最常见的多元数据分析类型之一。当基因表达定量后获得了各样本中所有基因的表达值信息,我们通常会期望比较样本之间在基因表达值的整体相似性或者差异程度。基因数量成千上万,肯定不能对每个基因的表达都作个比较,这时候就要用到“降维”算法,PCA分析因此派上用场。PCA设法将N维(N=基因数量)的表达矩阵降维成只有少数几个主成分的形式,这几个主成分就可以代表所有基因的整体表达格局,进而据此描述样本差异。
小花这里利用的R语言中ggolot2包,带小伙伴绘制可发表级别的主成分分析图。
我们先载入数据集和R包library(ggplot2)#小花这里使用经典iris数据集df <- iris[c(1, 2, 3, 4)]head(df)
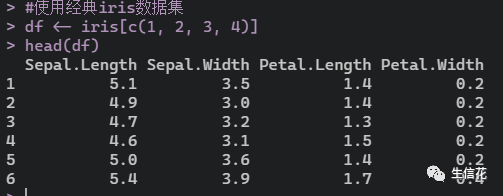
小伙伴们可以根据这种数据集形式,自行准备。
接下来进行主成分分析
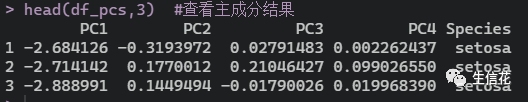
df_pca <- prcomp(df) #利用prcomp函数计算主成分df_pcs <-data.frame(df_pca$x, Species = iris$Species)head(df_pcs,3) #查看主成分结果
我们先使用基础的函数去绘制PCA图:

这样结果就展示出来了,但是图片没有颜色等其它的可以区分差异的特点,接下来我们使用ggplot2去美化PCA图。
#使用ggplot2 绘制PCA图#Species分颜色ggplot(df_pcs,aes(x=PC1,y=PC2,color=Species))+ geom_point()
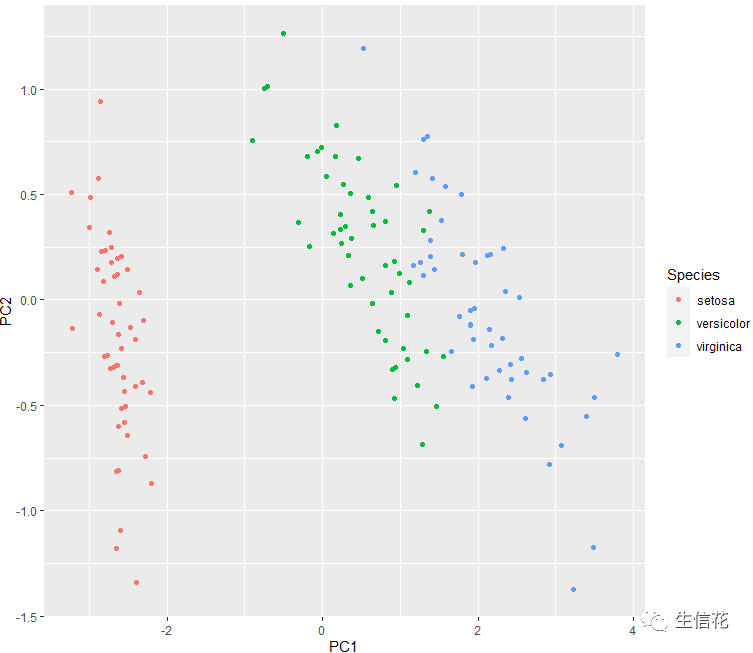
这样PCA图就绘制出来了,小伙伴发现,横坐标PC1以及纵坐标PC2,颜色都有变化,这样就可以清晰的看出三组数据有无明显差异。接下里我们继续调整一下细节。
#去掉背景及网格线ggplot(df_pcs,aes(x=PC1,y=PC2,color=Species))+geom_point()+theme_bw() +theme(panel.border=element_blank(),panel.grid.major=element_blank(),panel.grid.minor=element_blank(),axis.line= element_line(colour = "black"))
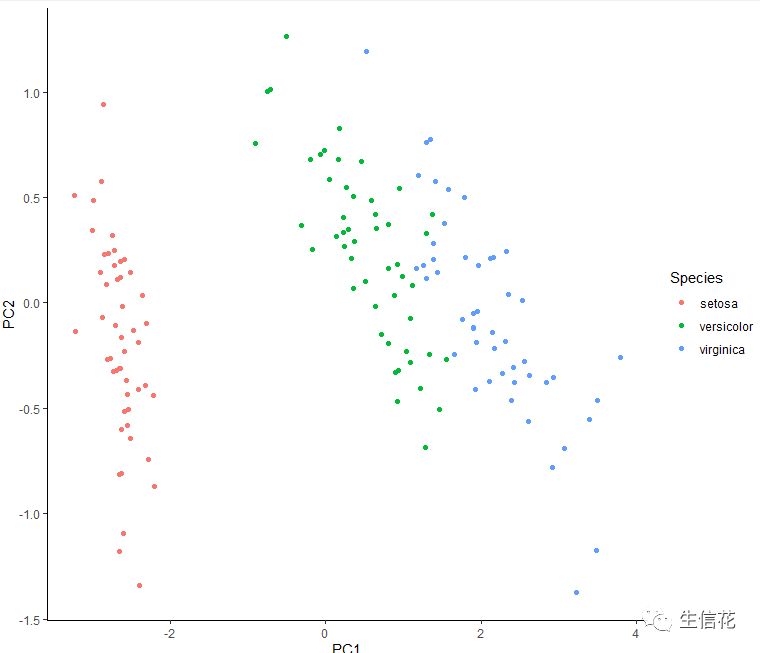
我们把背景网格去掉变得更清晰。
接下来我们添加
我们把背景网格去掉变得更清晰。接下来我们添加# 添加PC1 PC2的百分比percentage<-round(df_pca$sdev / sum(df_pca$sdev) * 100,2)percentage<-paste(colnames(df_pcs),"(", paste(as.character(percentage), "%", ")", sep=""))ggplot(df_pcs,aes(x=PC1,y=PC2,color=Species))+geom_point()+xlab(percentage[1]) +ylab(percentage[2])
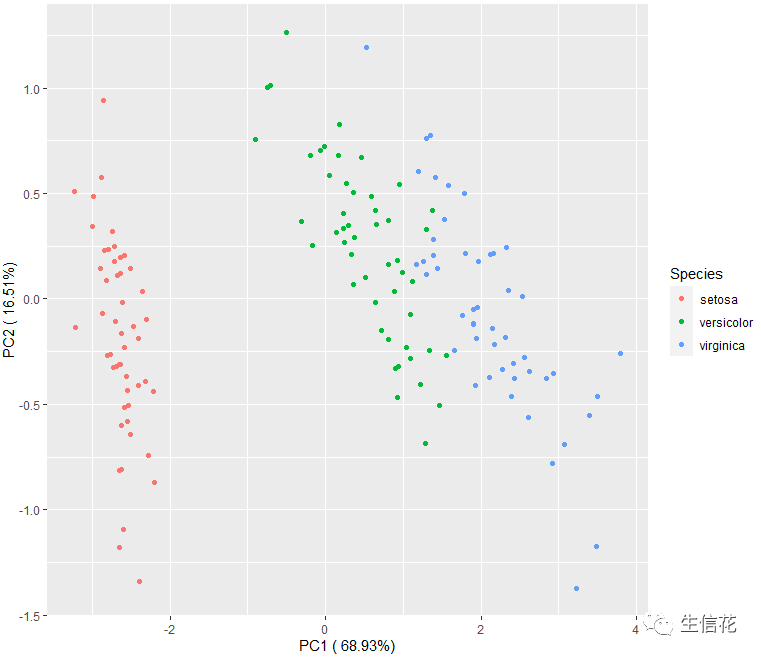
往往有数值的显示才能展现出图片的意义,所以我们把PC1和PC2的百分比,
下面我们添加# 添加置信椭圆ggplot(df_pcs,aes(x=PC1,y=PC2,color = Species))+ geom_point()+stat_ellipse(level = 0.95, show.legend = F) +annotate('text', label = 'setosa', x = -2, y = -1.25, size = 5, colour = '#f8766d') +annotate('text', label = 'versicolor', x = 0, y = - 0.5, size = 5, colour = '#00ba38') +annotate('text', label = 'virginica', x = 3, y = 0.5, size = 5, colour = '#619cff')
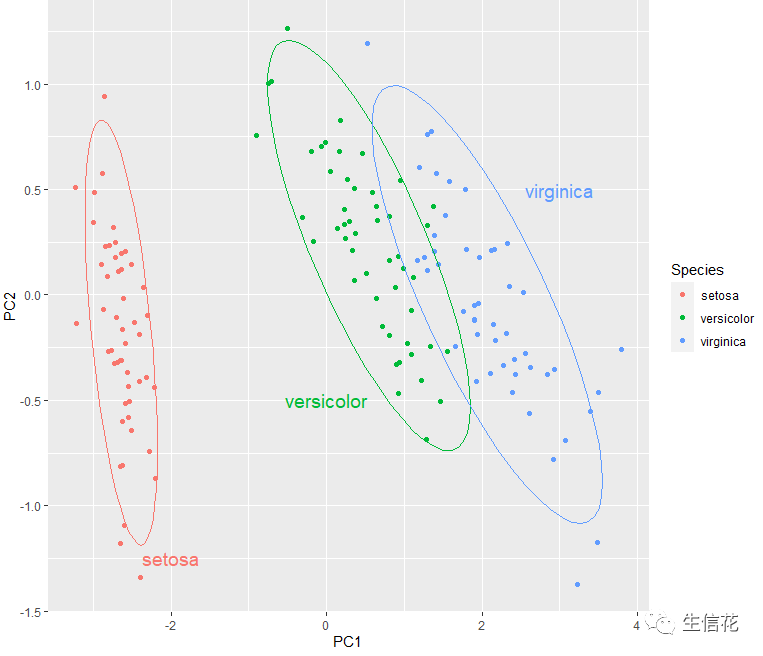
添加置信区间让分组的区别更明显。
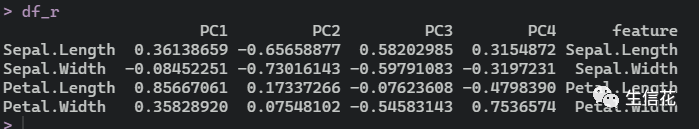
但是我们还需要计算各个变量对于PCA的贡献值,
#查看各变量对于PCA的贡献df_r <- as.data.frame(df_pca$rotation)df_r$feature <- row.names(df_r)df_r
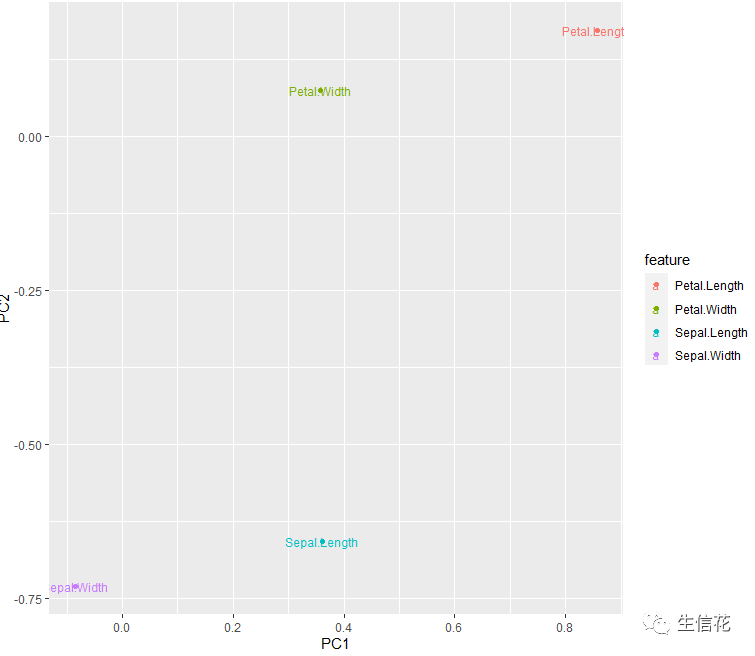
我们将贡献值以图片形式展示出来:
#贡献度绘图ggplot(df_r,aes(x=PC1,y=PC2,label=feature,color=feature )) + geom_point()+ geom_text(size=3)
最后我们PCA绘图进行汇总展示,把上述的细节都放在一起,进行绘制:
#PCA绘图汇总展示ggplot(df_pcs,aes(x=PC1,y=PC2,color=Species )) + geom_point()+xlab(percentage[1]) + ylab(percentage[2]) + stat_ellipse(level = 0.95, show.legend = F) +annotate('text', label = 'setosa', x = -2, y = -1.25, size = 5, colour = '#f8766d') +annotate('text', label = 'versicolor', x = 0, y = - 0.5, size = 5, colour = '#00ba38') +annotate('text', label = 'virginica', x = 3, y = 0.5, size = 5, colour = '#619cff') + labs(title="Iris PCA Clustering",subtitle=" PC1 and PC2 principal components ", caption="Source: Iris") + theme_classic()#这些参数小伙伴可以根据自己需求去修改
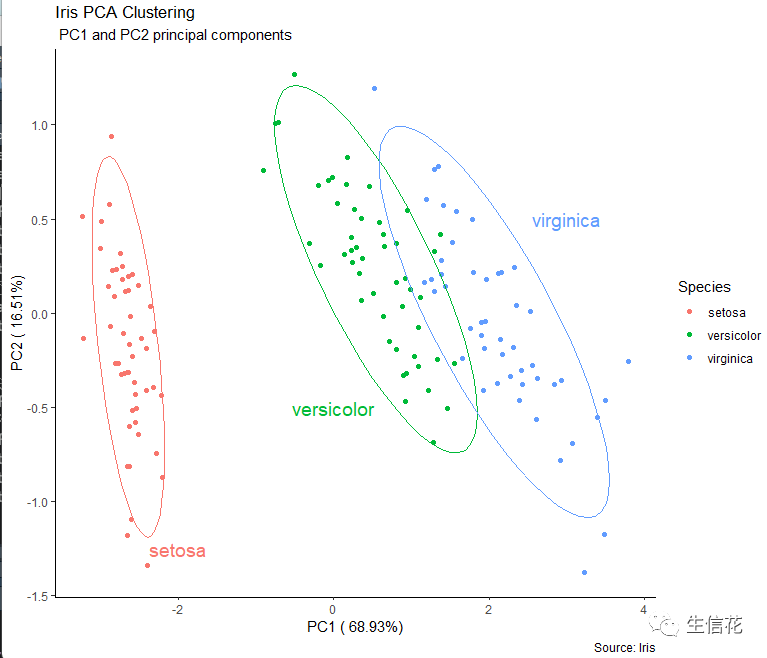
这样就大功告成了,有没有心动呢,小伙伴接下来就可以了更改数据集动手绘制PCA了。小花提醒小伙伴一下,要多多理解代码的含义,这样就可以根据自己喜好更改参数绘制好看的图片。
欢迎使用:云生信 – 学生物信息学 (biocloudservice.com)
如果想用服务器可以联系微信:18502195490(快来联系我们使用吧!)

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

