保姆级教程-小图教你如何使用ggrisk高效的绘制风险因子联动图!!!
点击蓝字
关注小图
小伙伴在做预后分析的时候,肯定都做过预后生存分析,那就很多时候避免不了构建预后生存风险模型,比如COX回归风险模型,lasso风险模型等等。而我们平时使用比较多的当属COX单因素或者多因素的风险模型,以往我们都已森林图或者三线表的形式去可视化COX生存风险模型,其实我们还可以用风险得分关联图去可视化风险模型的结果。小图今天在这里就教小伙伴绘制风险因子联动图,主要的展示是风险得分的散点图,还有高低风险的生存时间以及生存状态的散点图,当然还有重点基因的表达热图。
小图今天主要介绍R包ggrisk去绘制,并且教小伙伴常用的参数去调整,快跟着小图去学习吧:
library(ggrisk)library(survival)library(rms)
因为涉及到生存时间和状态的计算,这里主要用到这三个R包,用到其中的函数。
这里我们用到内置的数据集去展示LIRI
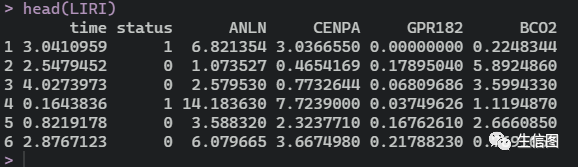
就是常规的含有生存时间和状态的基因表达谱,小伙伴可以按照这个格式去自行设置。
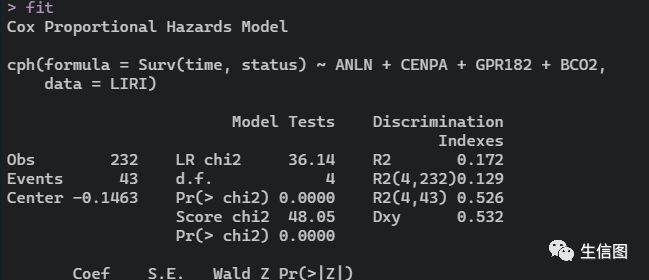
接下来我们先构建COX模型,这里我们使用ANLN、CENPA、GPR182和BCO2四个基因的表达值取构建
fit <- cph(Surv(time,status)~ANLN+CENPA+GPR182+BCO2,LIRI)
接下里就是可视化我们的风险因子联动图了,
先构建默认的图形:
ggrisk(fit)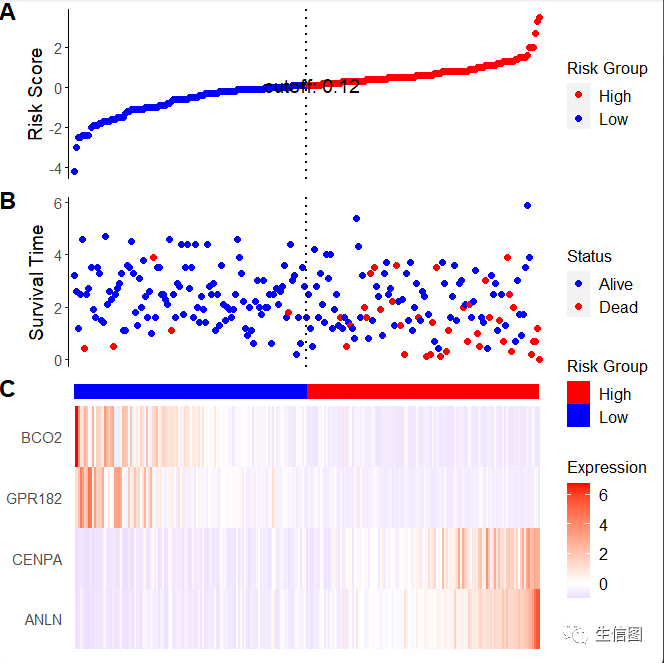
小伙伴发现没,图中有三个图,我们来解释一下,图A为风险得分按照从小到大顺序排列,小图这里使用的中值取分组,当然我们还可以使用计算好的风险评分取分组。图B为风险得分与生存时间的散点图,然后按照结果将散点图分为红色和蓝色,也就是生存状态,图C就很常规了是我们基因的表达量热图。
接下里我们可以用参数调整风险得分的cutoff还有位置。
ggrisk(fit,cutoff.value='cutoff', #可选‘median’, ’roc’ or ’cutoff’cutoff.x = 150, #“cutoff”文本的水平位置cutoff.y = -1 #“cutoff”文本的垂直位置)
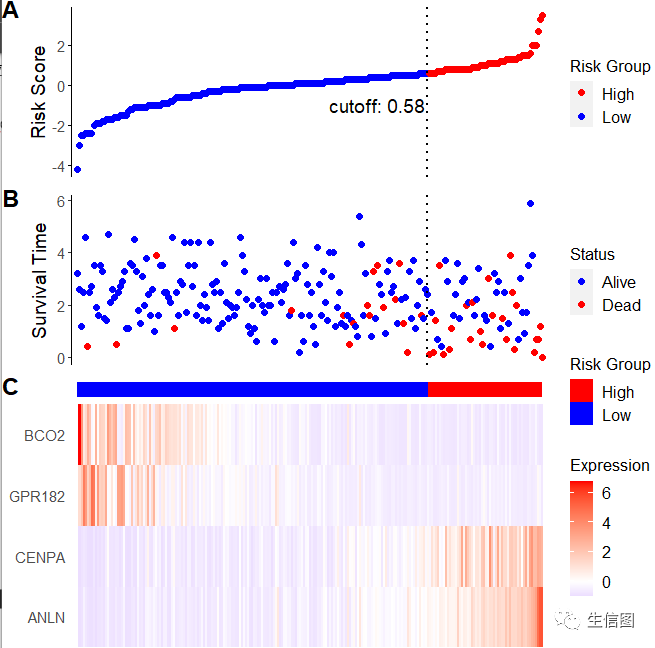
是不是明显不一样:
参数代码中呢,cutoff.value 是为划分风险值cutoff的方式,有以下几种:
-
cutoff.value = “median”:默认方式,使用风险得分的中位数作为切点值;
-
cutoff.value = “roc”:将风险得分和结局时间进行roc分析,将约登点作为最佳切点值;
-
cutoff.value = “cutoff”:将会使用cutoff包,通过最小p值法计算出最佳切点。
-
cutoff.value = 赋值数值:根据切点值将风险得分分为高危组和低危组。
小伙伴可以自行去设置
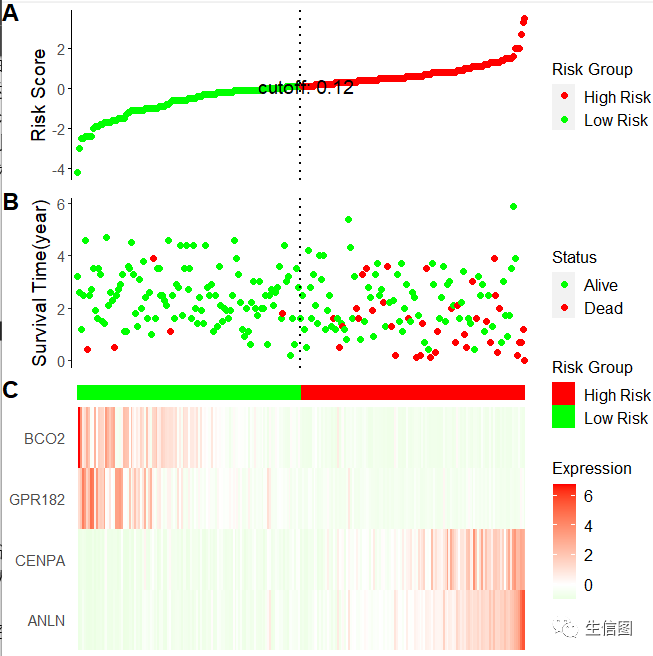
我们接下里去调整一下细节,还有颜色
ggrisk(fit,code.highrisk = 'High Risk',#高风险标签,默认为 ’High’code.lowrisk = 'Low Risk', #低风险标签,默认为 ’Low’title.A.ylab='Risk Score', #A图 y轴名称title.B.ylab='Survival Time(year)', #B图 y轴名称,注意区分year month daytitle.A.legend='Risk Group', #A图图例名称title.B.legend='Status', #B图图例名称title.C.legend='Expression', #C图图例名称relative_heights=c(0.1,0.1,0.01,0.15), #A、B、热图注释和热图C的相对高度color.A=c(low='green',high='red'),#A图中点的颜色color.B=c(code.0='green',code.1='red'), #B图中点的颜色color.C=c(low='green',median='white',high='red'), #C图中热图颜色vjust.A.ylab=1, #A图中y轴标签到y坐标轴的距离,默认是1vjust.B.ylab=2 #B图中y轴标签到y坐标轴的距离,默认是2)
小图这里就用绿色和红色去演示一下,小伙伴有喜欢演示自行去设置哦
参数含义,小图也在上面标注了
小伙伴还可以展示自己想要的基因热图:
ggrisk(fit, heatmap.genes=c('GPR182','CENPA','BCO2')) 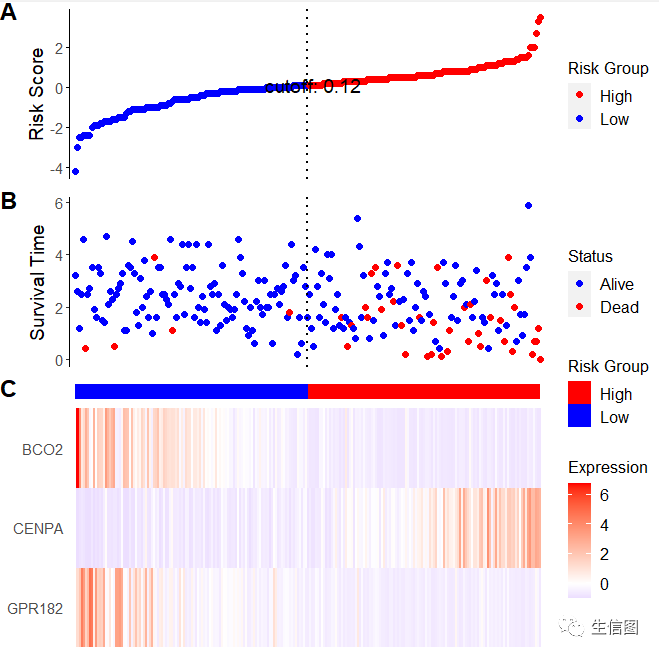
其他参数小伙伴可以按照上面的代码去设置。或者我们不要热图:
two_scatter(fit,cutoff.value = 'median',cutoff.x = 142,cutoff.y = -0.5)
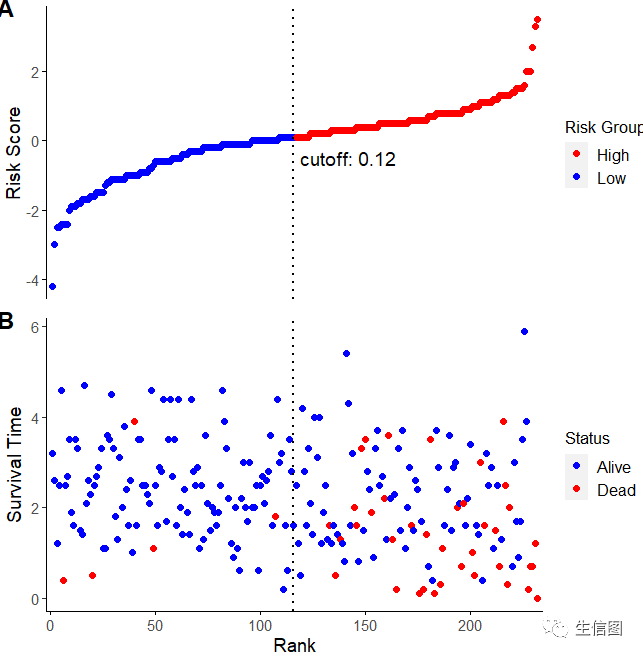

如果遇到不懂的也可以借助线上的云平台哦~http://www.biocloudservice.com/home.html
好了今天的峰峦图就讲到这里,欢迎大家有问题与小图一起讨论哦~
欢迎使用:云生信平台 ( http://www.biocloudservice.com/home.html)

|
往期推荐 |
|
|
|
|
|
|
👇点击阅读原文进入网址