玩转maftools:探索TCGA的基因世界!
hello,你知道如何使用maftools处理TCGA数据库下载的maf文件吗?今天小师妹就来教你轻松实现!带你玩转maftools包,感兴趣的话就和小师妹一起看下去吧!

什么是maftools?
maftools是一个在R语言环境中使用的强大的工具包,用于分析和可视化MAF(Mutation Annotation Format)文件。MAF文件是一种常用的存储肿瘤基因变异信息的格式。maftools提供了一系列函数和图形化工具,可以帮助研究人员更好地理解和解释肿瘤基因组数据。
以下是maftools包的一些主要功能和特点:
1.读取MAF文件:maftools提供了read.maf()函数,方便用户读取和解析MAF文件。它能够自动处理不同版本的MAF文件,并将其转换为易于处理的数据结构。
2.数据摘要和统计:maftools允许用户对MAF文件进行基本的摘要和统计分析。通过函数如mafSummary()和mafStats(),可以获取变异位点的分布、突变类型的频率、基因级别的变异统计等信息。
3.可视化工具:maftools提供了多种绘图函数,可以帮助用户直观地展示和可视化MAF数据。例如,oncoplot()函数可以生成基于基因变异频率的癌症类型条形图;oncostrip()函数可以绘制某些关注基因的突变概况;plotTiTv()函数可以绘制碱基转换(Transition)和碱基转换(Transversion)的分布比例。
4.突变负荷和突变特征分析:maftools提供了一系列函数,用于评估肿瘤样本的突变负荷(Mutation Burden),包括突变数目、突变频率等统计。此外,也可以通过signatures()函数来进行突变特征分析,识别潜在的突变模式和变异特征。
5.交互式可视化和报告生成:maftools支持创建交互式的HTML报告,其中包含了MAF数据的摘要信息、绘图结果和统计分析结果。这使得用户可以方便地共享和展示分析结果。
总而言之,maftools是一个功能强大且易于使用的R包,为研究人员提供了一套全面的工具,帮助他们分析和理解MAF文件中的肿瘤基因组数据。
下载TCGA LIHC MAF 数据
好啦,接下来进入我们今天的正题,首先我们需要下载TCGA LUAD MAF文件。我们今天通过TCGAbiolinks来下载所需要的数据,所以在下载之前大家要确保已经安装了TCGAbiolinks和maftools这两个R包哦。
library(TCGAbiolinks)query <- GDCquery( project = "TCGA-LIHC", data.category = "Simple Nucleotide Variation", data.type = "Masked Somatic Mutation", access = "open")# 下载GDCdownload(query)#保存 这里要设置为自己的工作目录以上代码可以帮助我们直接通过R包下载好所需的数据,从而免于从官方数据库下载,当然同学们也可以通过GDC数据库来下载哦,详细的下载方法参见小师妹之前的教程哦!
maftools处理TCGA数据
读取MAF数据
使用read.maf()函数读取MAF数据文件,并将结果存储在变量laml中。在这里,我们只读取组学数据,不添加临床数据。
#读取maf文件library(maftools)laml <- read.maf(maf.code)#本次只展示maf的一些统计绘图,只读入组学数据,不添加临床数据查看数据基本情况
使用laml来查看读取的MAF数据的基本情况,包括数据摘要和统计信息。
绘制MAF文件的整体结果图
plotmafSummary(maf = maf1, rmOutlier = TRUE, addStat = 'median', dashboard = TRUE)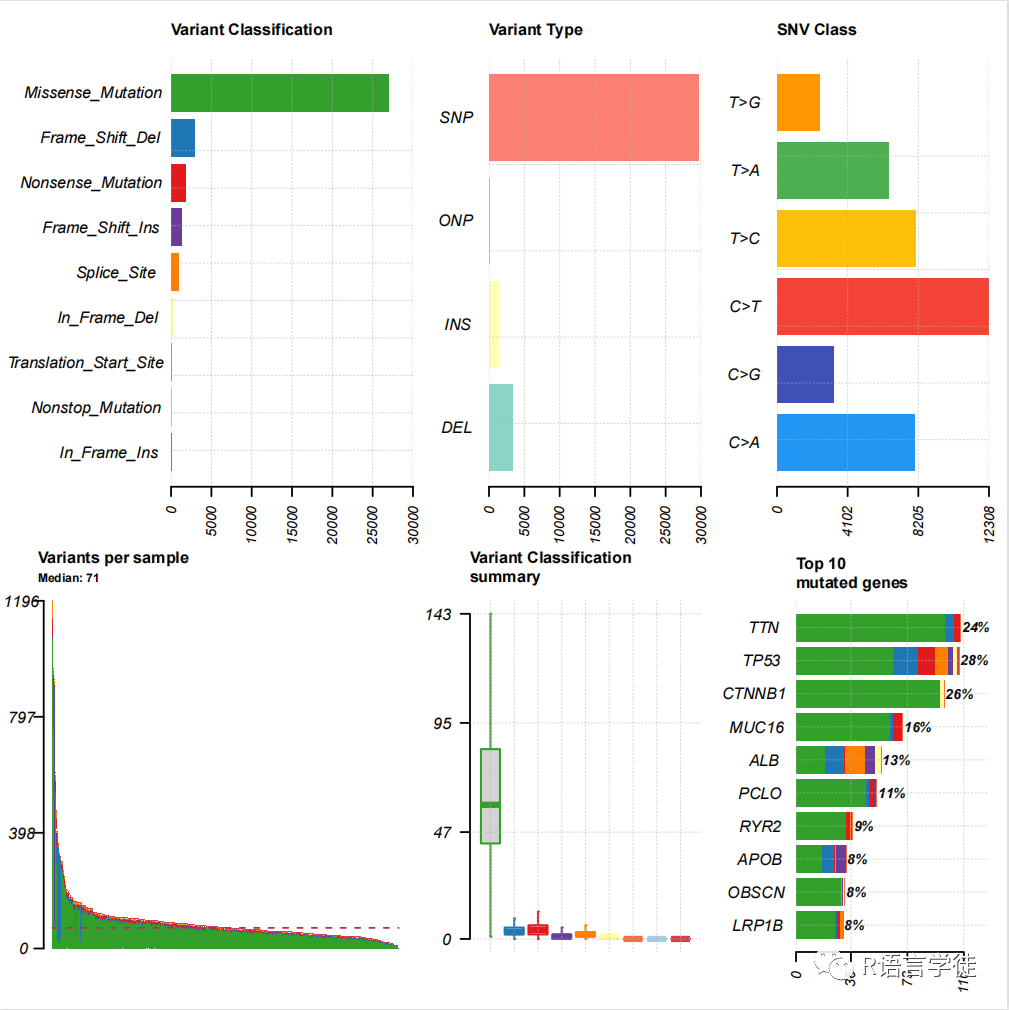
绘制oncoplot图
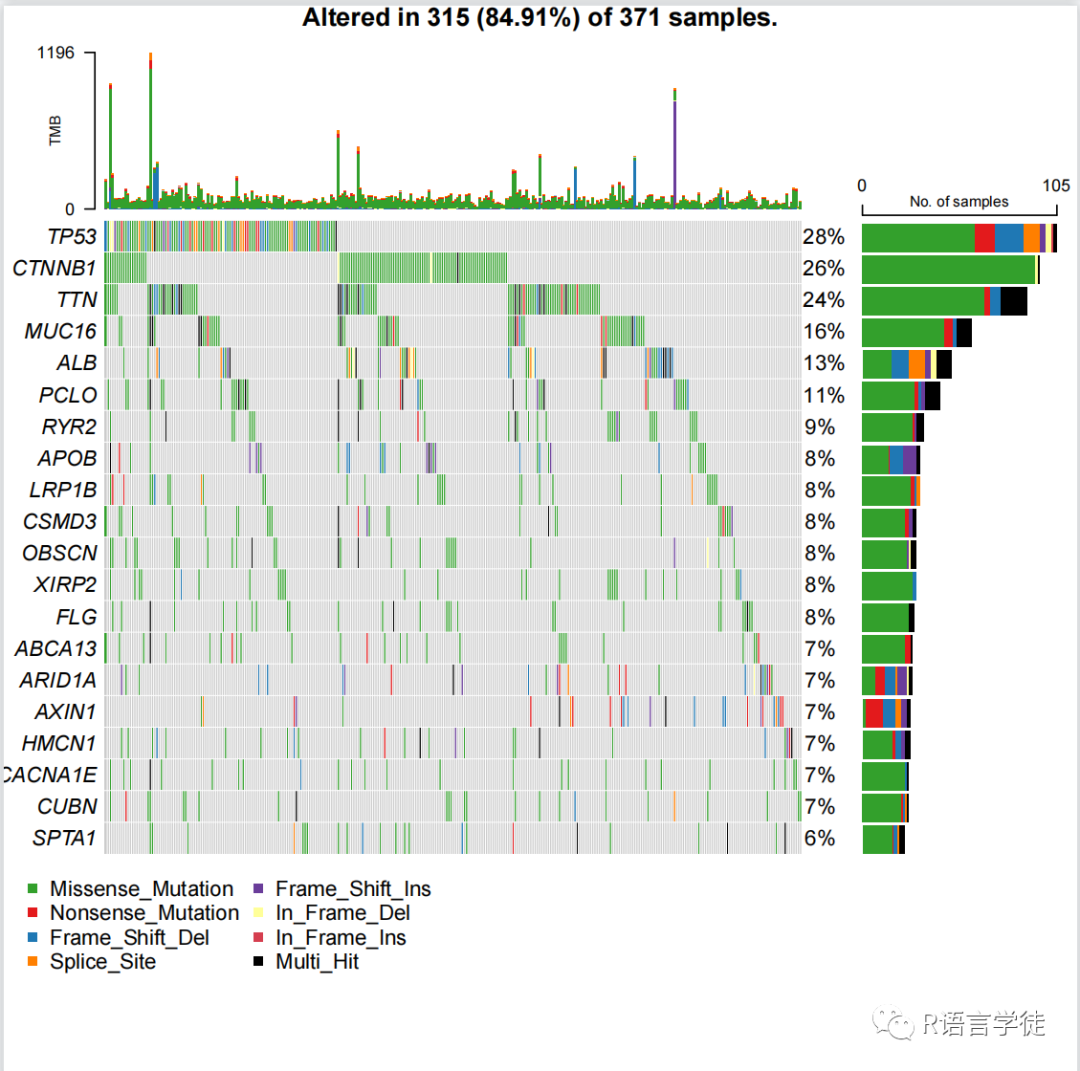
绘制Oncostrip图
可以使用 oncostrip 函数展示特定基因在样本中的突变情况,此处查看肝癌中关注较多的’TP53′,’CTNNB1′, ‘ARID1A’三个基因,如下:
oncostrip(maf = laml, genes = c('TP53','CTNNB1', 'ARID1A'))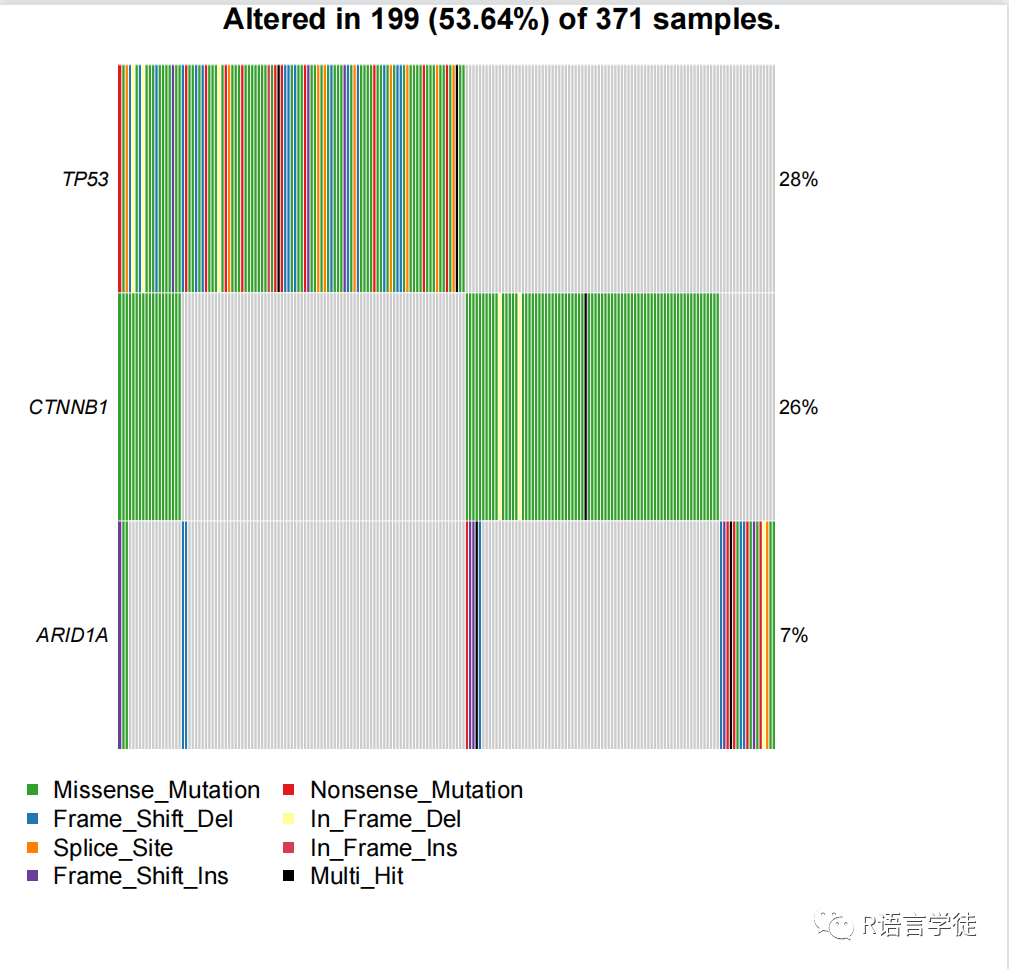
Transition vs Transversions
titv函数将SNP分类为Transitions_vs_Transversions,并以各种方式返回汇总表的列表。汇总数据也可以显示为一个箱线图堆积条形图显示每个样本中的转换比例
laml.titv = titv(maf = laml, plot = FALSE, useSyn = TRUE)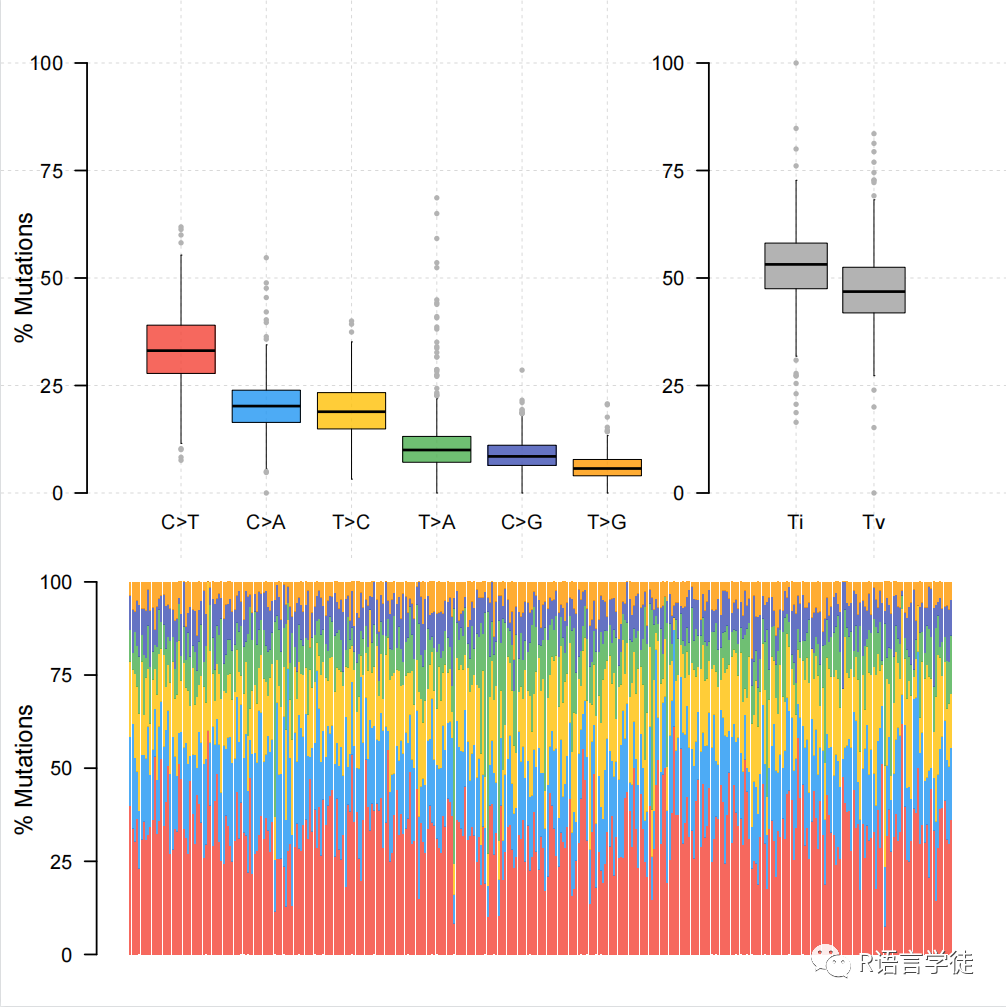
Rainfall plots
使用rainfallPlot参数绘制rainfall plots,展示超突变的基因组区域。detectChangePoints设置为TRUE,rainfall plots可以突出显示潜在变化的区域。
rainfallPlot(maf = laml, detectChangePoints = TRUE, pointSize = 0.6)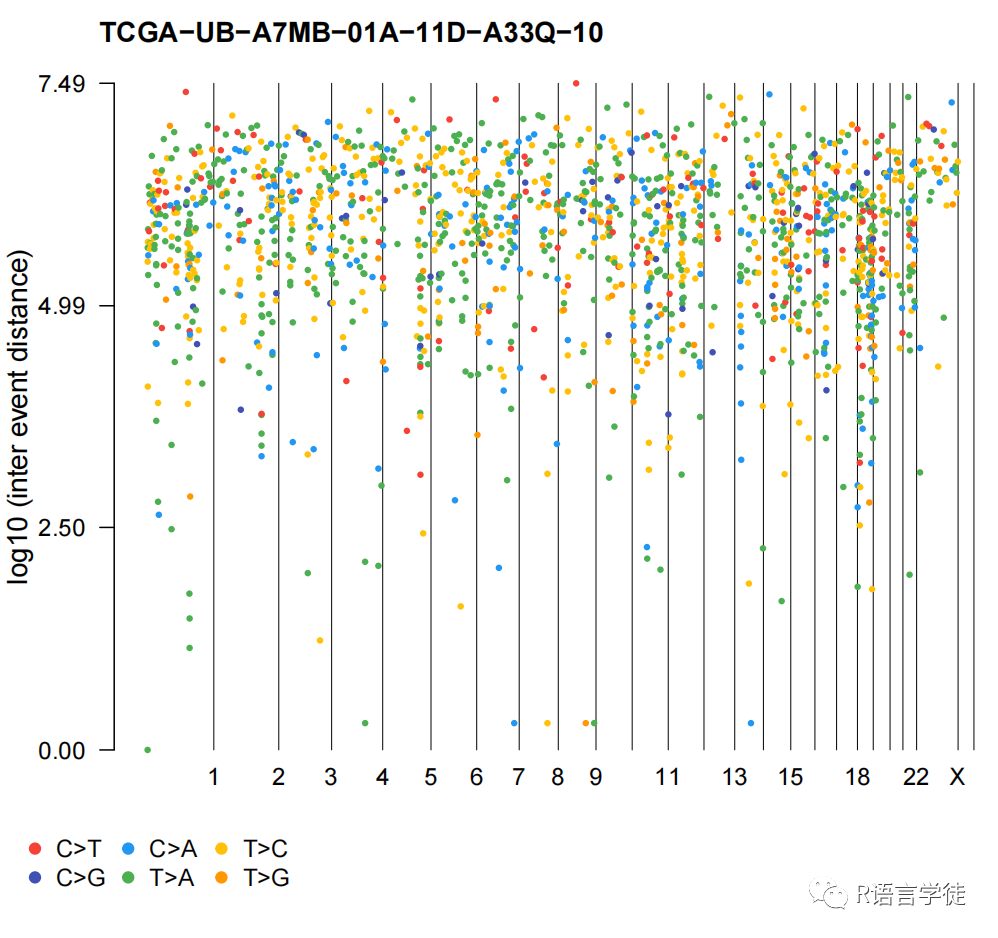
通过以上步骤,你可以使用给定的代码下载TCGA的MAF数据并进行基本的数据整理和分析。小师妹温馨提示:一定要确保在运行代码之前,确保正确安装了所需的R包,并根据需要修改文件路径和其他参数哦。
怎么样,今天的教程你学会了吗?是不是很简单呢!更多学习干货请继续关注小师妹哦!

