10行代码搞定随机森林算法筛选重要基因特征
点击蓝字 关注我们
小伙伴们大家好,我是大海哥,今天大海哥打算给大家介绍一个实现起来简单,而且在生信分析种非常实用的一种用于基因筛选的机器学习方法,那就是咱们熟悉的随机森林,只不过不是用于预测,而是用于特征提取!一起来学习一下吧!
首先,我们来了解一下为什么要进行基因特征提取呢?
基因特征提取是生物信息学和生物数据分析领域的重要任务之一,它的背景可以追溯到分子生物学、遗传学和计算机科学的交叉领域:
在基因研究和生物信息学中,特征提取是一个关键的任务。通过从大规模的基因数据中提取出有意义的特征,研究人员可以识别基因与疾病、表型和其他生物学特征之间的关联。这些特征可以用于分类、聚类、预测和其他生物信息学任务,有助于深入理解生物学过程和疾病机制。
同时近年来,机器学习和人工智能技术已经在生物信息学中得到广泛应用。这些技术可以用于自动化基因特征提取的过程,帮助科研人员发现隐藏在生物学数据中的模式和关联。
所以使用机器学习手段来提取潜在的重要基因特征在这个大数据时代显得格外重要。
那么介绍了这么多,让我们一起来看看究竟需要怎样才能10行代码就实现这一流程呢?让我们开始吧!
#首先我们需要加载随机森林的关键实现包library(randomForest)#然后导入我们的基因表达数据df2<-read.csv(“data.csv”,header=T,row.names=1,check.names=F)#然后们看看数据的基本格式是什么样子的
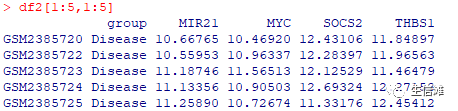
#可以看到,行为样本,列为基因,同时包含一列分类特征,这里大海哥选择是否患病为分类特征,大家也可以根据需求自行修改哦!#然后我们就需要指定我们的训练数据和结果变量x<-df2[,-1]y<-factor(df2$group)#然后就可以开始构建随机森林了rf<-randomForest(x, y,data=df2,importance=TRUE,mtry=3, proximity=TRUE)#这里我给大家介绍一下具体的参数,拓展一下知识面。subset以向量的形式确定样本数据集;na.action指定数据集中缺失值的处理方法,默认为na.fail,即不允许出现缺失 值,也可以指定为na.omit,即删除缺失样本;x指定模型的解释变量,可以是矩阵,也可以是数据框;y指定模型的因变量,可以是离散的因子,也可以是连续的数值,分别对应于随机森林的分类模型和预测模型。这里需要说明的是,如果不指定y值,则随机森林将是一个无监督的模型;ntree指定随机森林所包含的决策树数目,默认为500;mtry指定节点中用于二叉树的变量个数,默认情况下数据集变量个数的二次方根(分类模型)或三分之一(预测模型)。一般是需要进行人为的逐次挑选,确定最佳的m值;replace指定Bootstrap随机抽样的方式,默认为有放回的抽样classwt指定分类水平的权重,对于回归模型,该参数无效;strata为因子向量,用于分层抽样;sampsize用于指定样本容量,一般与参数strata联合使用,指定分层抽样中层的样本量;nodesize指定决策树节点的最小个数,默认情况下,判别模型为1,回归模型为5;maxnodes指定决策树节点的最大个数;importance逻辑参数,是否计算各个变量在模型中的重要性,默认不计算,该参数主要结合importance()函数使用;proximity逻辑参数,是否计算模型的临近矩阵,主要结合MDSplot()函数使用;norm.votes显示投票格式,默认以百分比的形式展示投票结果,也可以采用绝对数的形式;#上面就是一些主要的参数,还有更多可以自行查看官网哦!#查看一下我们构建的模型
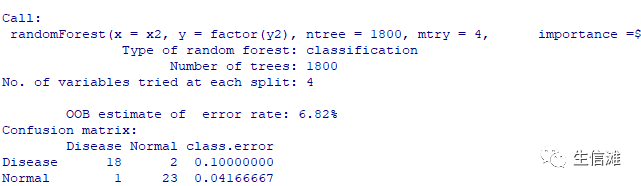
#然后我们就可以看一下重要性评分啦!head(importance(rf), type=2)
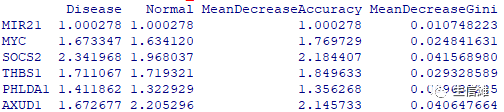
#type=1是以准确率递减方法得到维度重要性值。type=2为基尼系数方法#直方图直观查看系数重要性,下边两个好像没啥区别,大家自行选择,第二种可以选择展示的基因数量varImpPlot(rf, main = "variable importance")varImpPlot(rf, sort=TRUE,n.var=nrow(head(rf$importance,10)),main = "variable importance")
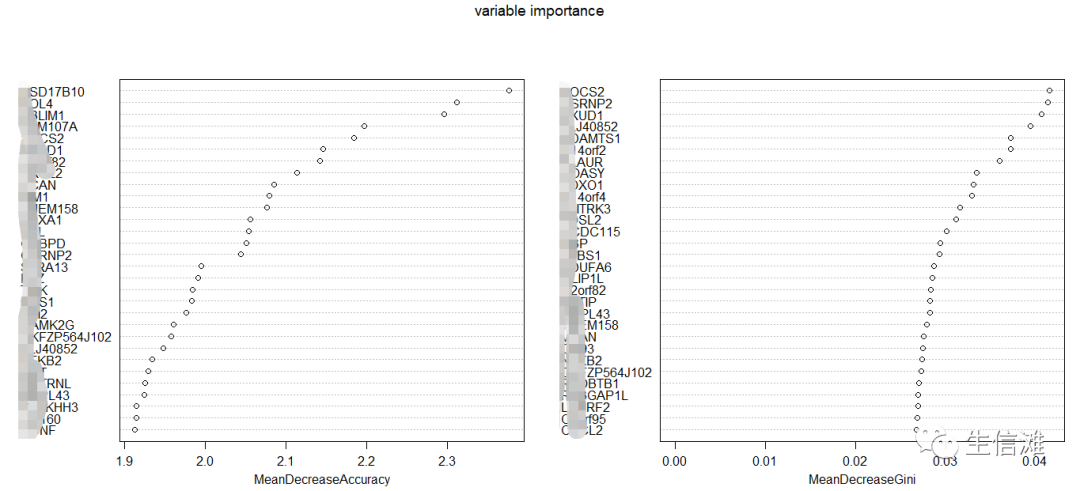
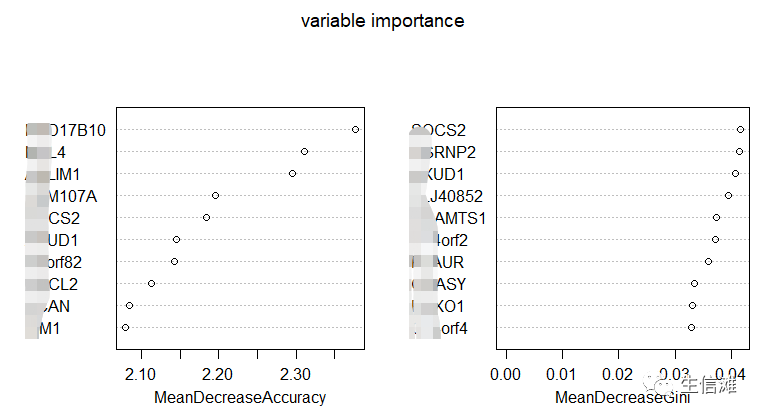
不过排名都是一样的。

生信滩公众号

点击“阅读原文”进入网址
