还在画常规的火山图?小果教你让你的火山图“活”过来!
火山图的含义我们也不过多的介绍,一般的我们常用来展示基因表达差异的分布,横坐标一般为Fold change,越偏离中心差异倍数就越大;横坐标就为P value也就是我们常见到P值,值越大呢就越显著,接下来就跟随小果去学习吧!
这里我们使用的是我们的老朋友ggplot2
公众号后台回复“111″,领取代码,代码编号:231006
library(ggplot2)#调用一下,顺便导入我们的数据data <- read.csv("火山data.csv",header=TRUE,row.names = 1)head(data)
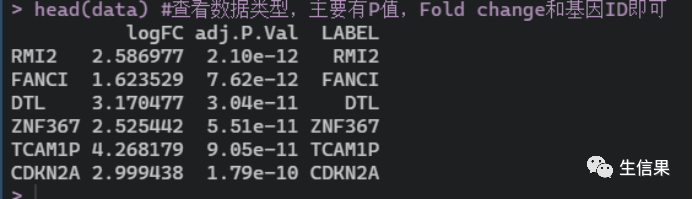
#查看数据类型,主要有P值,Fold change和基因ID即可,后面有一个LABEL,可能会有小伙伴疑惑,后面小果会介绍到,不要担心,小伙伴自行把其他数据设置好就可以跟着小果继续学习
先绘制简单的火山图点图
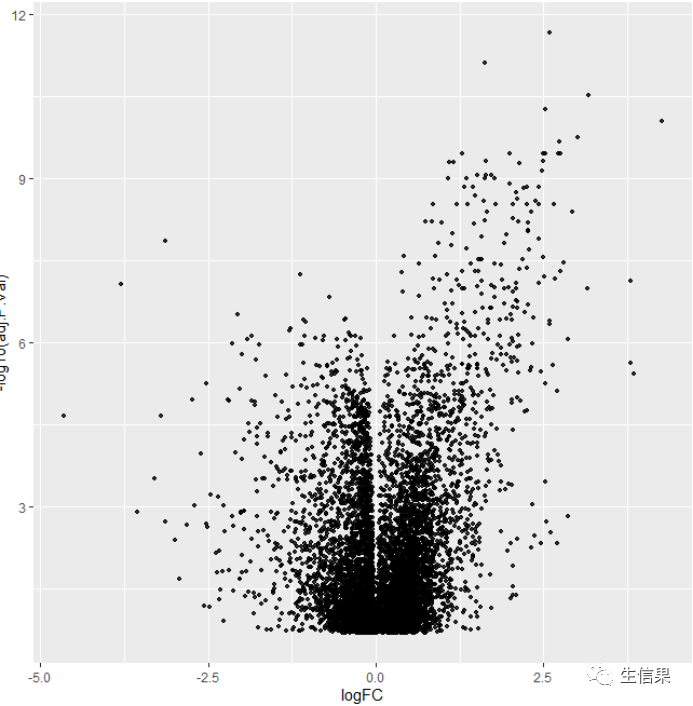
是不是单调,没有任何信息让我们去分析,我们接下来把细节去优化一下比如:最重要的上下调基因的区分,横纵轴的阈值线,重点基因的标注,我们一步一步来
先根据阈值设定上下调的基因 这里我们用代码新增change列,然后利用ifelse函数添加基因的上下调情况,然后color进行区分,最后使用geom_hline和geon_vline()的参数去设置阈值线。
data$change <- as.factor(ifelse(data$adj.P.Val < 0.01 & abs(data$logFC) > 1,ifelse(data$logFC > 1,'UP','DOWN'),'NOT'))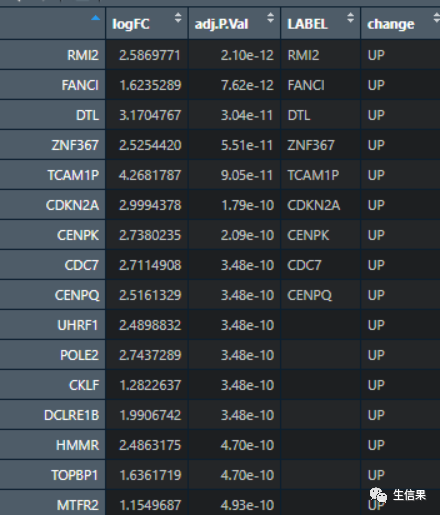
是不是增加了一列新的
然后添加阈值线还有颜色区别
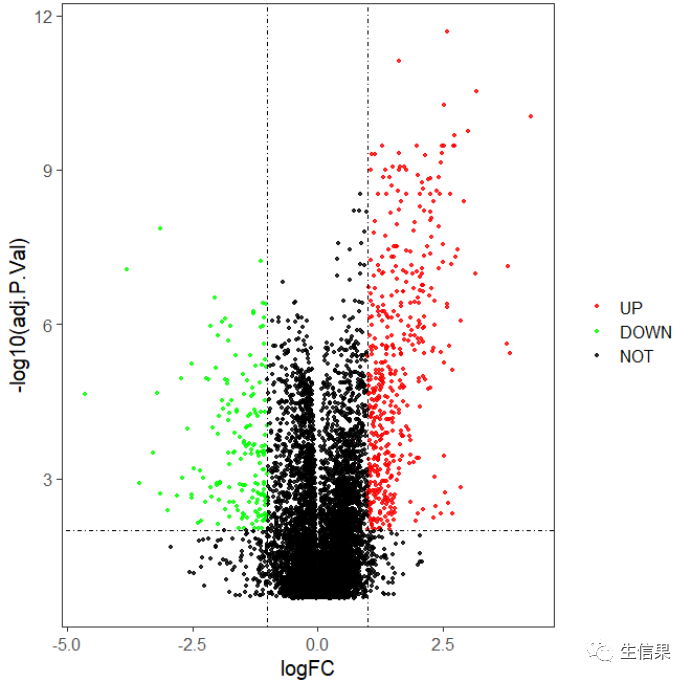
这样一来就明显多了,小伙伴往往绘制到这里就不往下绘制了这样一来我们显著的基因也没办法在图片中寻找到,我们去标注重点显著的差异基因:
这里我们新增sign列,利用ifelse函数添加重点显著差异基因,然后使用geom_text参数添加到图上
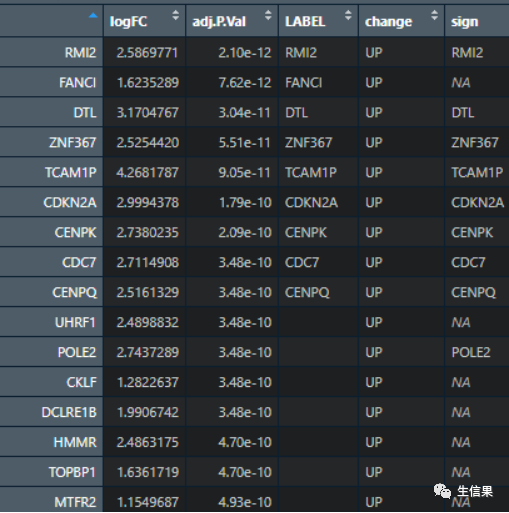
然后绘制图片
ggplot(data = data, aes(x = logFC, y = -log10(adj.P.Val), color = change)) +geom_point(alpha=0.8, size = 1) +theme_bw(base_size = 15) +theme(panel.grid.minor = element_blank(),panel.grid.major = element_blank()) +geom_hline(yintercept=2 ,linetype=4) +geom_vline(xintercept=c(-1,1) ,linetype=4 ) +scale_color_manual(name = "", values = c("red", "green", "black"), limits = c("UP", "DOWN", "NOT")) +geom_text(aes(label = sign), size = 3)
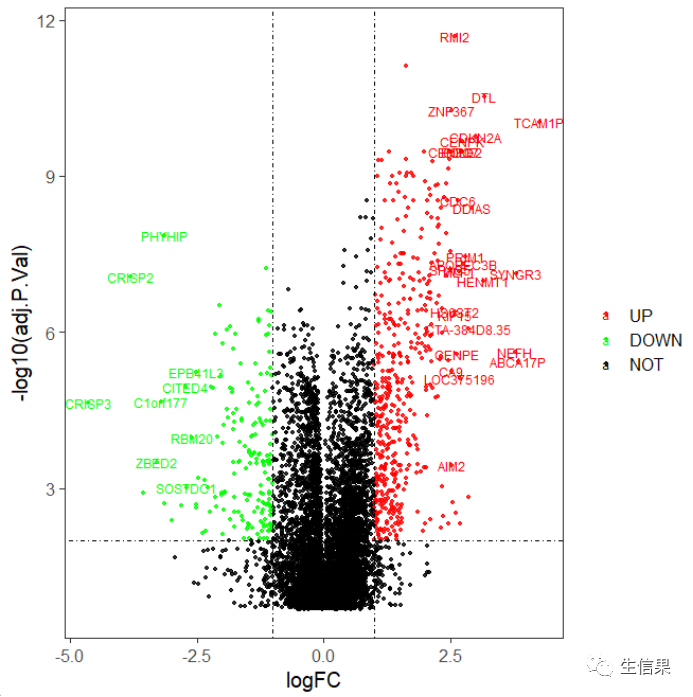
结果就出来了,不过小伙伴发现很多的基因都是重叠的,没办法分清楚
这个时候就用到ggplot2包中的ggrepel我们的老朋友啦
这个包可以帮我们解决重叠的问题:
library(ggrepel)ggplot(data = data, aes(x = logFC, y = -log10(adj.P.Val), color = change)) +geom_point(alpha=0.8, size = 1) +theme_bw(base_size = 15) +theme(panel.grid.minor = element_blank(),panel.grid.major = element_blank()) +scale_color_manual(name = "", values = c("red", "green", "black"), limits = c("UP", "DOWN", "NOT")) +geom_label_repel(aes(label=sign), fontface="bold", color="grey50", box.padding=unit(0.35, "lines"), point.padding=unit(0.5, "lines"), segment.colour = "grey50")
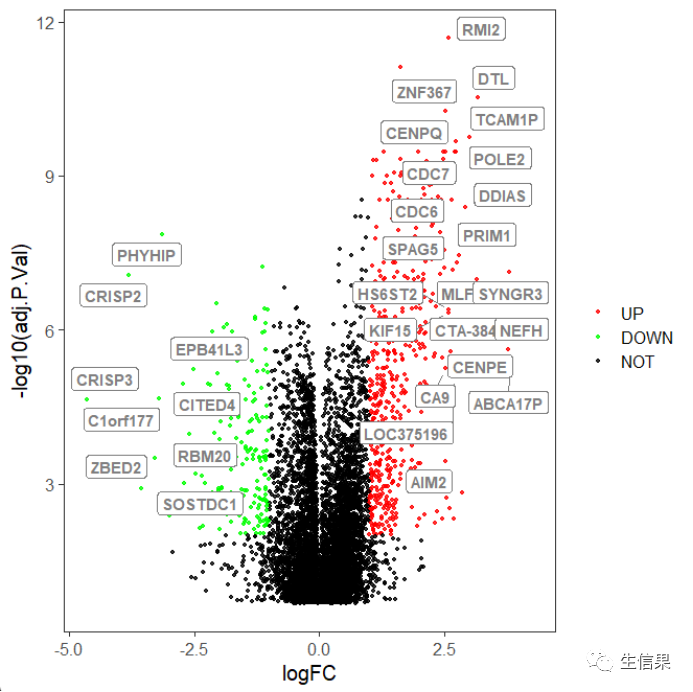
是不是明显不一样了 每一个基因都很清楚的展示出来,
那小伙伴想展示自己想展示的基因怎么办,不要困惑,小果继续教大家:
我们把感兴趣的基因添加在LABEL列,就是一开始小果导入数据的卖的官司,这里小果自己随便写了几个基因给小伙伴演示一下:
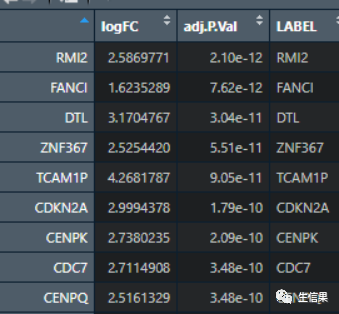
下面我们去绘制图片:
ggplot(data = data, aes(x = logFC, y = -log10(adj.P.Val), color = change)) +geom_point(alpha=0.8, size = 1) +theme_bw(base_size = 15) +theme(panel.grid.minor = element_blank(),panel.grid.major = element_blank() ) +scale_color_manual(name = "", values = c("red", "green", "black"), limits = c("UP", "DOWN", "NOT")) +geom_label_repel(aes(label=LABEL), fontface="bold", color="grey50", box.padding=unit(0.35, "lines"), point.padding=unit(0.5, "lines"), segment.colour = "grey50")
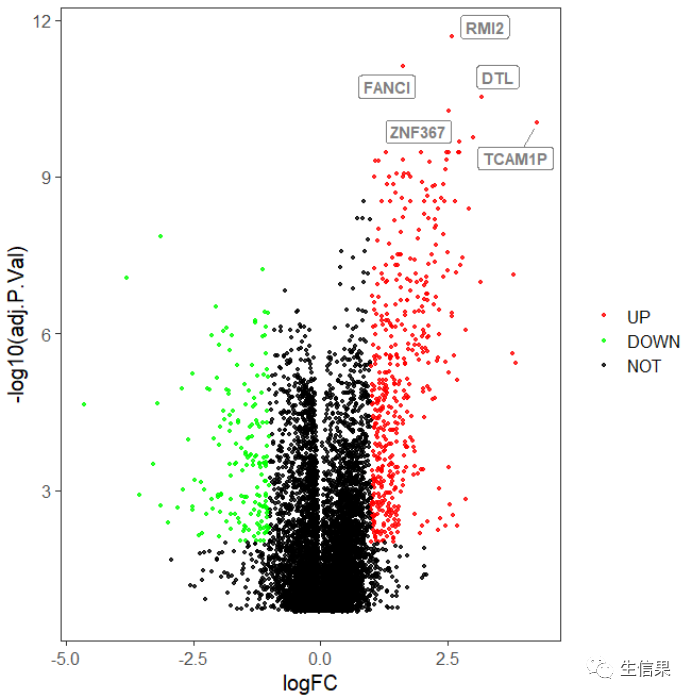
是不是就出现了呢,小伙伴是否学会呢,一定要亲手去实验一下,才能学的更精,
不过小果在这里做个小插曲
我们可以制作交互式的火山图,虽然不能放在小伙伴的文章中,不过平时我们汇报其他用途时候还是很有意思的
来看一下:用plot_ly函数绘制散点图
library(plotly)plot_ly(data,x = ~logFC, y = ~-log10(adj.P.Val),text = ~sign, type = 'scatter', mode = 'markers')
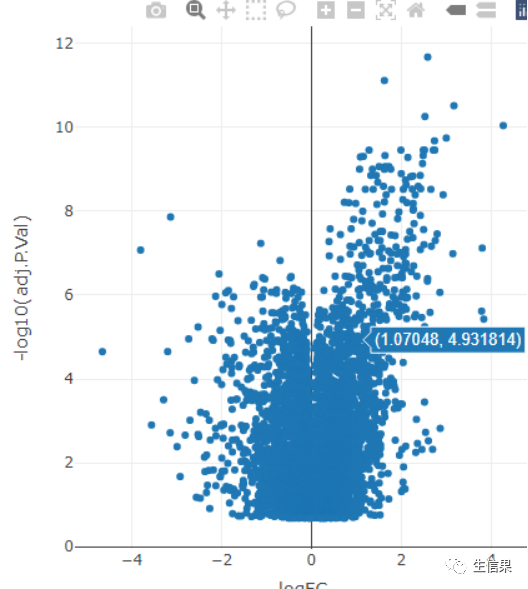
会出现一个页面,当我们鼠标放上去时候就会显示那个点的信息,是不是很有意思,
上述“活”过来的火山图是否学会了呢,小伙伴要多多理解代码的意思,才能绘制出自己想要的图片!
往期推荐

