CNV究竟是细胞的福音还是噩梦?单细胞转录组数据到底有多复杂?今天,用 infercnv包为你答疑解惑!

公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240423
需要租赁服务器的小伙伴可以扫码添加小果,此外小果还提供生信分析,思路设计,文献复现等,有需要的小伙伴欢迎来撩~

if (!require("BiocManager", quietly = TRUE))install.packages("BiocManager ")BiocManager::install("infercnv") # 在BiocManager环境下安装infercnv

packageVersion("infercnv") # 查看infercnv版本
install.packages("tibble") # 安装tibble包install.packages("devtools") # 安装devtools包devtools::install_github("bmbroom/tsvio") # 安装tsviodevtools::install_github("bmbroom/NGCHMR", ref="stable") # 安装 NGCHMRdevtools::install_github("broadinstitute/inferCNV_NGCHM") #安装inferCNV_NGCHM
library(infercnv) # 载入infercnv包setwd("/R-4.3.3/library/infercnv/extdata") # 需要更换为自己R的安装位置infercnv_obj <- CreateInfercnvObject(raw_counts_matrix = "./oligodendroglioma_expression_downsampled.counts.matrix.gz", # 载入原始数据annotations_file = "./oligodendroglioma_annotations_downsampled.txt", # 载入注释文件delim = "t",gene_order_file = "./gencode_downsampled.EXAMPLE_ONLY_DONT_REUSE.txt", # 载入基因目录文件ref_group_names = c("Microglia/Macrophage", "Oligodendrocytes (non-malignant)"))使用infercnv包进行基因拷贝突变数分析:class(infercnv_obj) # 查看infercnv_obj对象类型

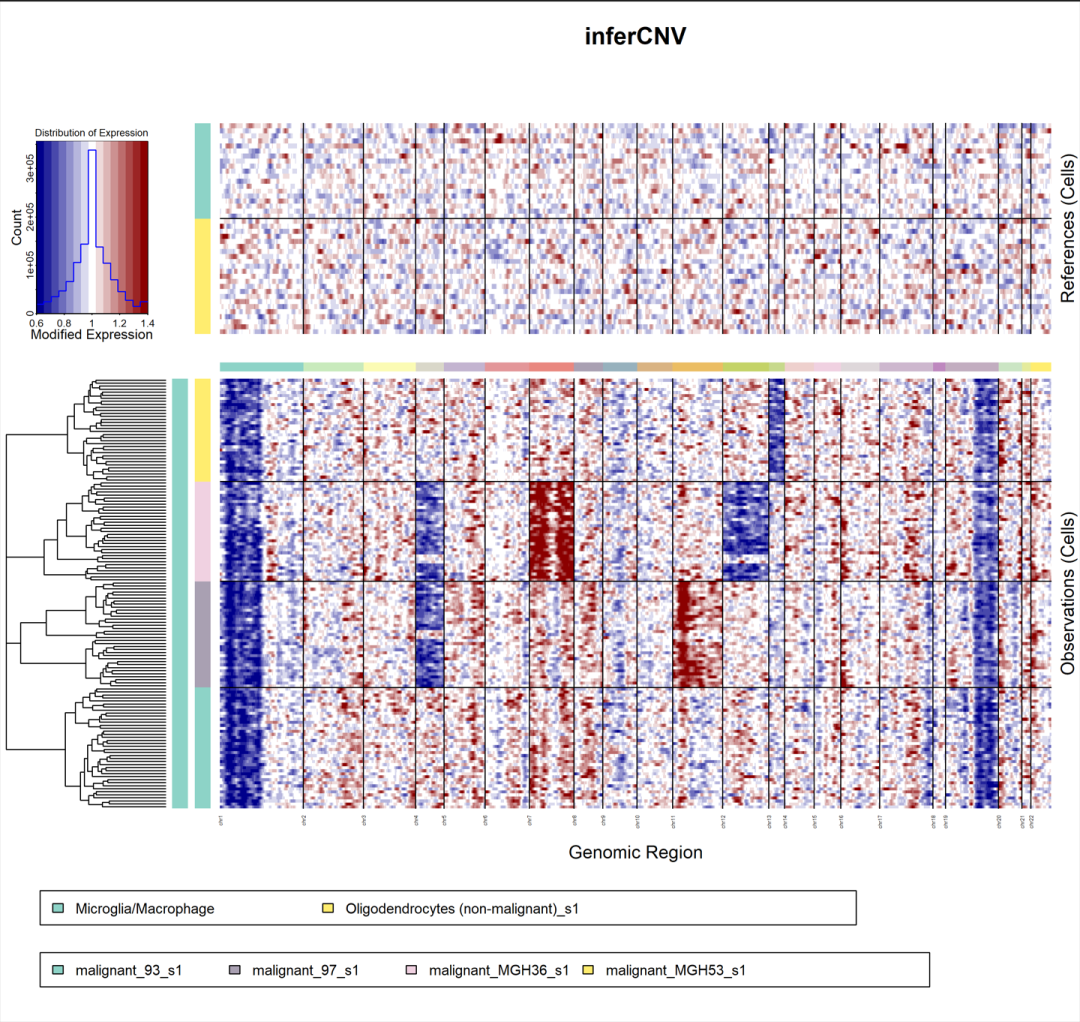
out_dir = getwd()infercnv_obj_default = infercnv::run(infercnv_obj, # 使用之前创建的infercnv对象infercnv_obj作为输入cutoff=1, # 设置用于过滤细胞的表达量阈值,cutoff=1适用于Smart-seq2数据,cutoff=0.1适用于10x Genomics数据out_dir=out_dir, # 指定输出结果的文件夹路径,out_dir是您之前定义的输出目录变量cluster_by_groups=TRUE, # 根据指定的参考组进行细胞群集分析plot_steps=FALSE, # 是否绘制分析步骤的图表,设置为FALSE表示不绘制denoise=TRUE, # 是否对数据进行去噪处理,设置为TRUE表示进行去噪HMM=FALSE, # 是否使用隐马尔可夫模型(Hidden Markov Model)进行分析,设置为FALSE表示不使用no_prelim_plot=TRUE, # 是否绘制预处理后的细胞图谱,设置为TRUE表示不绘制png_res=60 # 设置输出PNG图像的分辨率为60dpi)

小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果

往期回顾
|
01 |
|
02 |
|
03 |
|
04 |