做蛋白组的同学看过来!DEP保姆级教程!!
近年来随着质谱仪器的发展,蛋白质组学取得了长足进步,在蛋白质组学愈加火热的今天,还不赶紧学习蛋白质组的分析方法?好的,今天就让小果手把手教你如何使用蛋白质组分析软件包DEP吧!
1.分析概述
软件包DEP提供了一个集成的分析工作流程,用于对差异蛋白质表达或差异富集的质谱法蛋白质组学数据进行可靠和可重复的分析。
公众号后台回复“111″,领取代码,代码编号:231011
2.安装以及加载R包
if (!requireNamespace("BiocManager", quietly=TRUE))install.packages("BiocManager")BiocManager::install("DEP")library("DEP")
3.交互式使用(可选)
该软件包包含shiny的应用程序,它允许下面描述的整个工作流程的交互式分析。这些应用程序特别适用于有限或没有 R 经验的用户。目前,有两种不同的应用程序。一个用于基于无标签定量(LFQ)的分析(来自 MaxQuant 的输出) ,另一个用于基于串联质量标签(TMT)的分析(来自 IsobarQuant 的输出)。要运行这些应用程序,只需运行以下命令:
# 用于 LFQ 分析run_app("LFQ")# 用于 TMT 分析run_app("TMT")
4.差异表达分析
我们分析了泛素-蛋白质相互作用的蛋白质组学数据集(Zhang et al. Mol Cell 2017)。原始质谱数据首先使用 MaxQuant (Cox and Mann, Nat Biotech 2007)进行分析,经过分析之后得到的“ proteininGroups.txt”文件用作下游分析的输入。
4.1 样品数据集:Ubiquitin interactors
4.2 导入数据
# 导入用于数据处理的软件包。library("dplyr")# 这里我们使用软件包自带的数据。data <- UbiLength# 我们筛选污染蛋白质和诱饵数据库匹配结果,它们分别由“Potential.contaminants”和“Reverse”列中的“ +”表示data <- filter(data, Reverse != "+", Potential.contaminant != "+")The data.frame has the following dimensions:dim(data)## [1] 2941 23The data.frame has the following column names:colnames(data)

4.3数据准备
我们确认数据集是否具有唯一的 Uniprot 标识符
# 先判断数据集是否有重复的基因名?data$Gene.names %>% duplicated() %>% any()

# 制作一个重复基因名称的表格。data %>% group_by(Gene.names) %>% summarize(frequency = n()) %>%arrange(desc(frequency)) %>% filter(frequency > 1)

# 为了进一步分析,这些蛋白质必须得到唯一的名称。此外,一些蛋白质没有注释的基因名称,对于这些,我们将使用 Uniprot IDdata_unique <- make_unique(data, "Gene.names", "Protein.IDs", delim = ";")# Are there any duplicated names?data$name %>% duplicated() %>% any()

4.4 生成一个SummarizedExperiment 对象
许多Bioconductor使用 SummarizedLaboratory 对象作为输入和/或输出。这类对象包含并协调实际(化验)数据、关于样品的信息以及特征注释。我们可以使用两种不同的方法从数据中生成 SummarizedLaboratory 对象。我们可以直接从列名中提取样本信息,或者使用实验设计模板添加样本信息。示例数据集的包中包含了一个实验性设计。
实验设计必须包含“label”、“condition”和“replicate”列。“标签”列包含不同样品的标识符,它们应该与包含化验数据的列名相对应。“条件”和“复制”列包含用户定义的这些示例的注释。
# 根据实验设计生成一个SummarizedExperiment对象。LFQ_columns <- grep("LFQ.", colnames(data_unique))experimental_design <- UbiLength_ExpDesigndata_se <- make_se(data_unique, LFQ_columns, experimental_design)LFQ_columns <- grep("LFQ.", colnames(data_unique)) # get LFQ column numbersdata_se_parsed <- make_se_parse(data_unique, LFQ_columns)# 现在让我们看一下生成的 SummarizedExperiment 对象data_se
4.5 SummarizedExperiment的先决条件
Make _ se 和 make _ se _ parse 函数生成一个 SummarizedLaboratory 对象,该对象具有两个规范。测定数据是 log2转换的,其行名描述蛋白质名称。RowData 包含 make _ only 生成的“ name”和“ ID”列。ColData 包含实验设计,从而包含示例注释。因此,colData 包括“ label”、“condition”和“replicate”列以及一个新生成的“ ID”列。Log2转换的测定数据和指定的 rowData 和 colData 列是后续分析步骤的先决条件。
4.6 对缺失值进行筛选
数据集包含的蛋白质并非在所有重复中都被量化。有些蛋白质甚至只在一次重复中被量化。
# 绘制样品间蛋白质鉴定重叠的条形图。plot_frequency(data_se)

这使得我们的数据集包含很多需要被填充的缺失值。然而,对于含有太多缺失值的蛋白质不应该这样做。因此,我们首先筛选出含有太多缺失值的蛋白质。这是通过在 filter _ missval 函数中为每个条件允许的缺失值数量设置阈值来完成的。
# 过滤出来至少在一个条件的所有重复中被鉴定的蛋白质data_filt <- filter_missval(data_se, thr = 0)data_filt2 <- filter_missval(data_se, thr = 1)# 绘制每个样品中鉴定出的蛋白质数量的条形图plot_numbers(data_filt)

# 绘制样品间蛋白质鉴定重叠的条形图plot_coverage(data_filt)

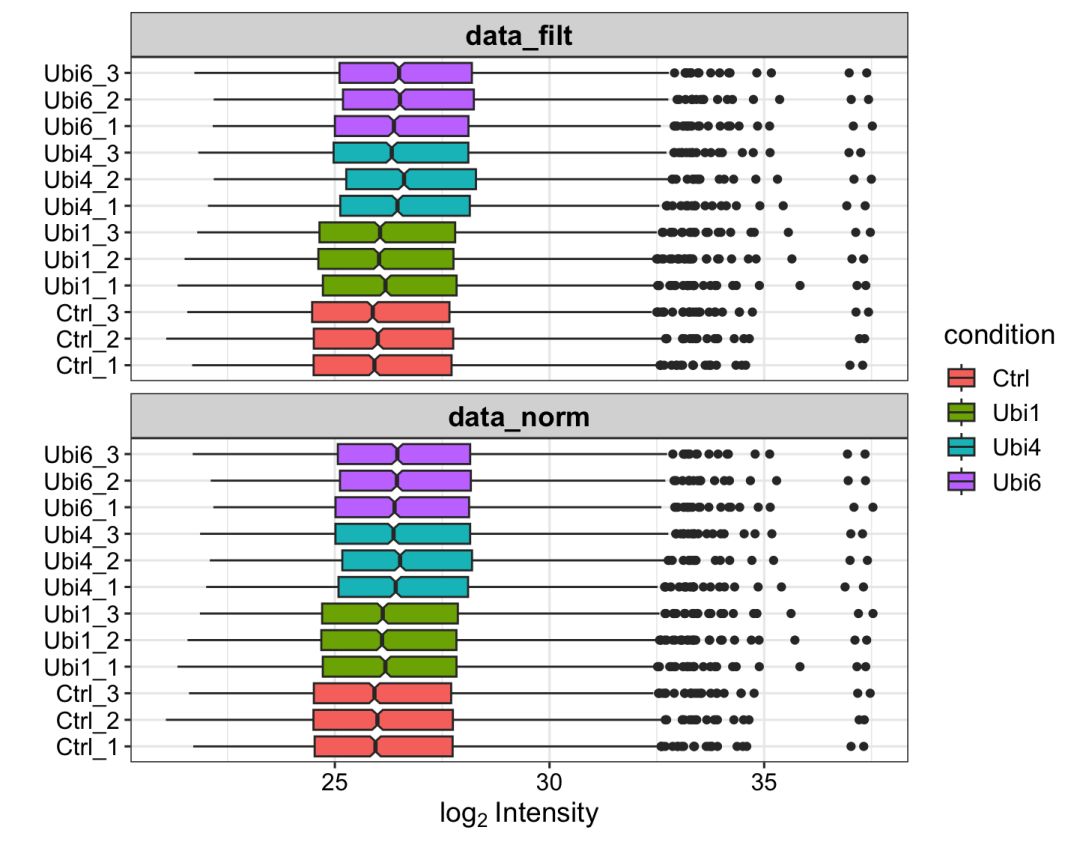
4.7 归一化
通过方差稳定化变换(vsn)对数据进行背景校正和归一化。
# 归一化数据。data_norm <- normalize_vsn(data_filt)# 通过箱形图展示所有样品在归一化前后的区别。plot_normalization(data_filt, data_norm)
 4.8 填充缺失值
4.8 填充缺失值
需要估算数据集中其余缺少的值。数据可能随机丢失(MAR) ,例如,如果蛋白质在某些重复中被量化,而在其他重复中不被量化。数据也可能不是随机丢失(MNAR) ,例如,如果蛋白质没有在特定条件下定量(例如在对照样品中)。MNAR 可以表明蛋白质在特定样品中低于检测限,这在蛋白质组学实验中可能是非常好的情况。
# 绘制包含缺失值的蛋白质热图。plot_missval(data_filt)

这个热图表明,缺失的值高度偏向于特定的样本。示例数据集是泛素相互作用的亲和力富集数据集,它可能在特定样品中具有低于检测限的蛋白质。这些可能是在泛素纯化中特异性富集的蛋白质,但在对照样品中不富集,反之亦然。为了检查缺失值是否偏向于较低强度的蛋白质,密度和累积分数绘制了有或没有缺失值的蛋白质。
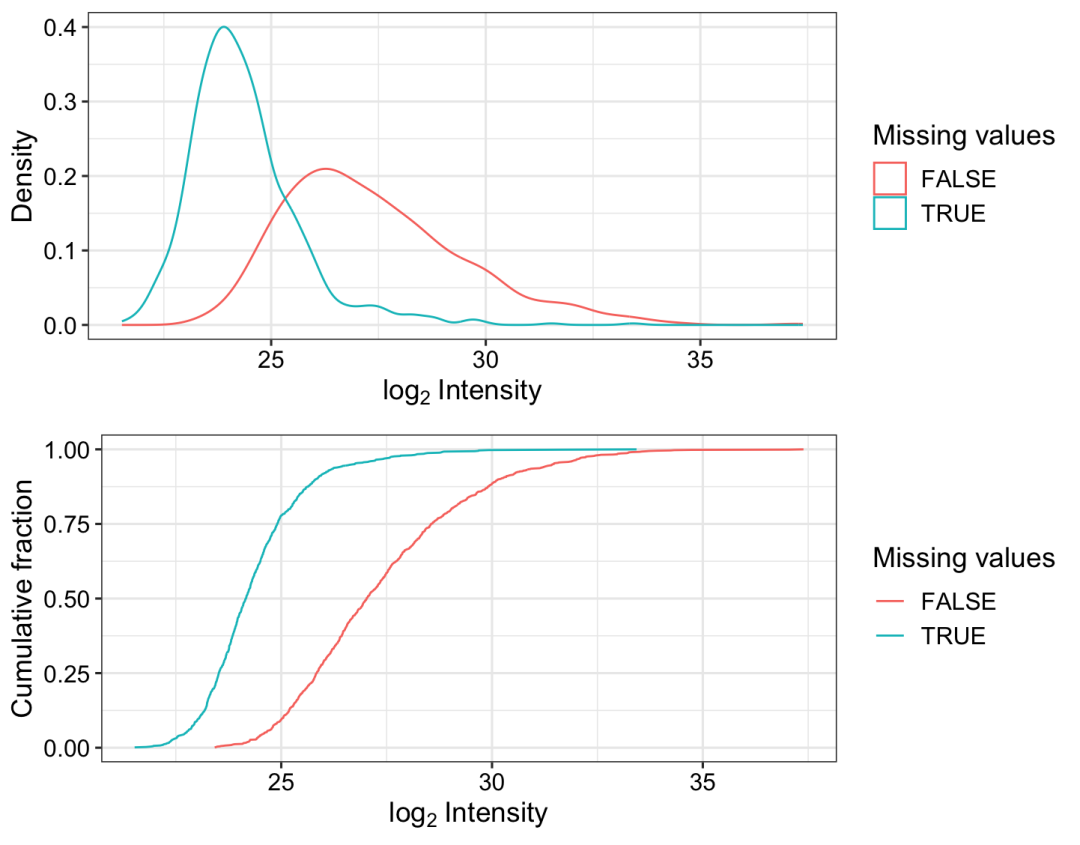
# 使用以最小值为中心的随机抽取的正态分布填充缺失的数据。data_imp <- impute(data_norm, fun = "MinProb", q = 0.01)# 填充前后的强度分布图plot_imputation(data_norm, data_imp)
 4.9 差异富集分析
4.9 差异富集分析
# 基于线性模型和贝叶斯统计的富集分析。data_diff <- test_diff(data_imp, type = "control", control = "Ctrl")data_diff_all_contrasts <- test_diff(data_imp, type = "all")data_diff_manual <- test_diff(data_imp, type = "manual",test = c("Ubi4_vs_Ctrl", "Ubi6_vs_Ctrl"))dep <- add_rejections(data_diff, alpha = 0.05, lfc = log2(1.5))
4.10 结果的可视化
前面分析的结果可以通过许多函数很容易地显示出来。这些可视化有助于确定使用的最佳cutoff值,突出最有趣的样品和对比度,并精确定位差异富集/表达的蛋白质。
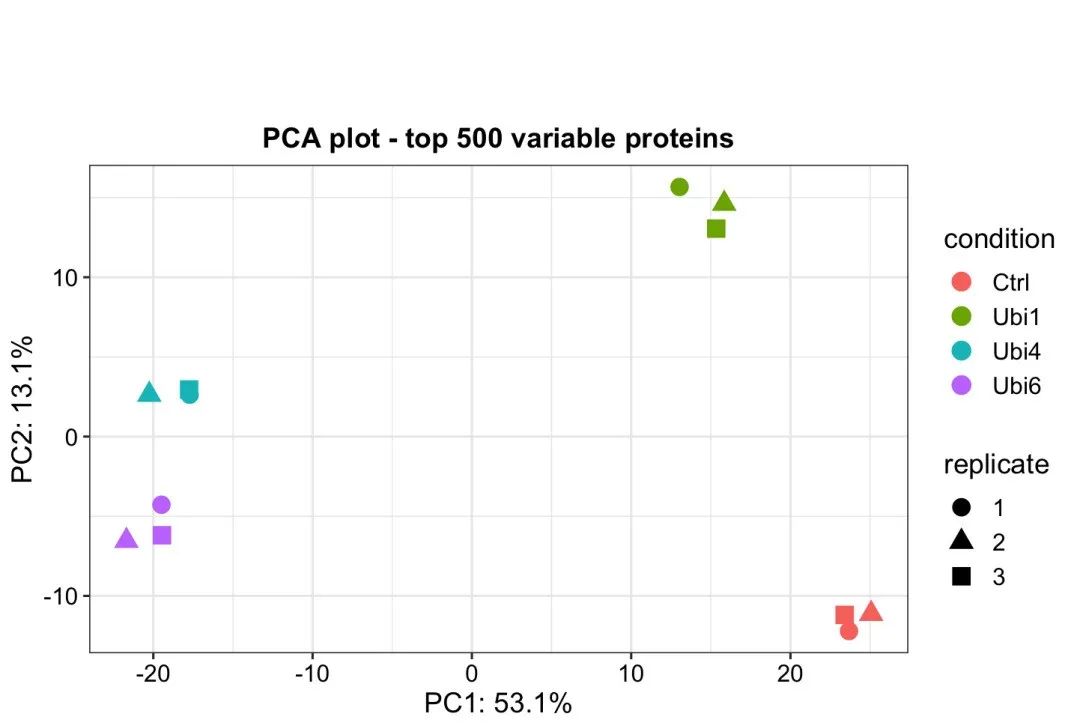
4.10.1 PCA.
# 绘制第一和第二主成分。plot_pca(dep, x = 1, y = 2, n = 500, point_size = 4)
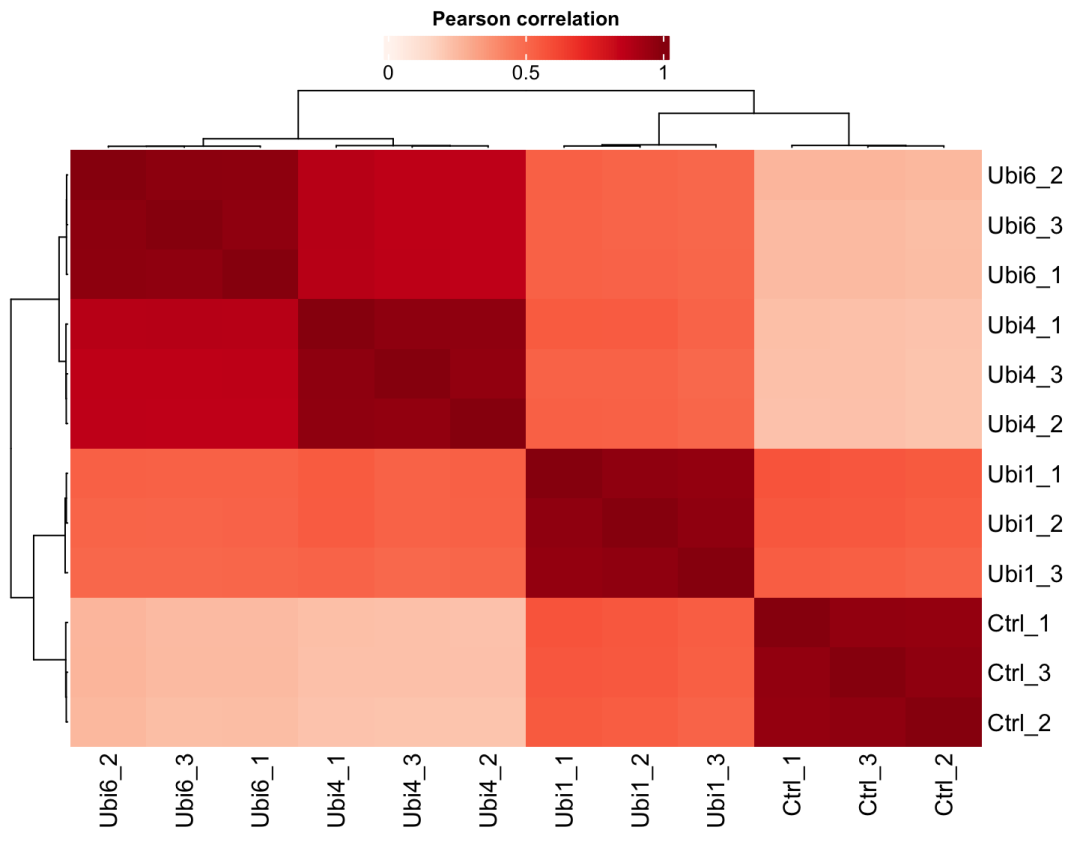
4.10.2 相关性系数矩阵
plot_cor(dep, significant = TRUE, lower = 0, upper = 1, pal = "Reds")
4.10.3 所有重要蛋白质的热图
热图表示给出了所有样品(列)中所有重要蛋白质(行)的概述。这样可以看到一般趋势,例如一个样本或复制是否与其他样本真的不同。此外,样品(列)的聚类可以表明更密切的相关样品和蛋白质(行)的聚类表明类似的行为蛋白质。
# 绘制所有重要蛋白质的热图。plot_heatmap(dep, type = "centered", kmeans = TRUE,k = 6, col_limit = 4, show_row_names = FALSE,indicate = c("condition", "replicate"))

4.10.4 绘制差异蛋白火山图
# 绘制 "Ubi6 vs Ctrl""的火山图。plot_volcano(dep, contrast = "Ubi6_vs_Ctrl", label_size = 2, add_names = TRUE)

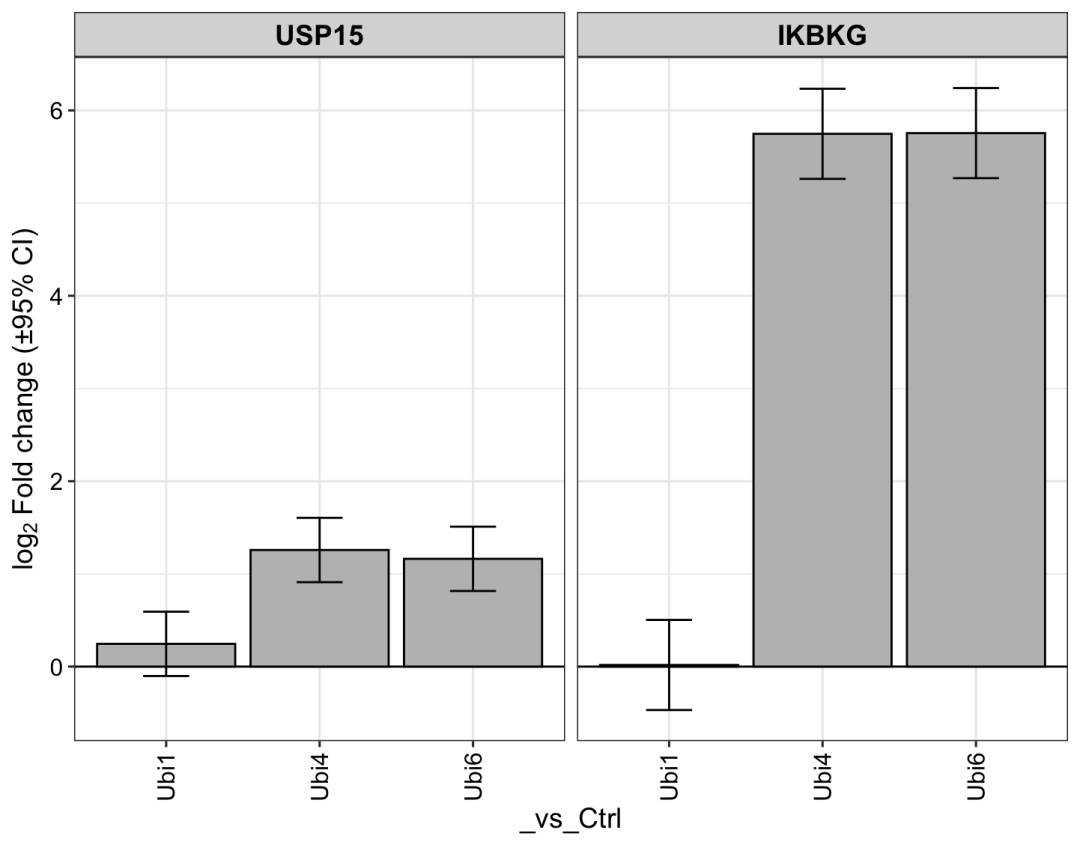
4.10.5 感兴趣蛋白质的条形图
# 绘制 USP15 和 IKBKG 的条形图。plot_single(dep, proteins = c("USP15", "IKBKG"))
4.10.6 重要蛋白的频率图
# 绘制不同条件下重要蛋白的频率图

好啦,以上就是蛋白质组下游分析软件包DEP的使用教程,包括下游数据处理以及差异富集分析等,希望小果可以帮助到你!
往期推荐



