解锁生物数据的无限奥秘:IMIFA方法的探索与应用
收录于话题
大家好,小果又和大家见面了。大家都知道因子分析模型(FA:Factor Analysers)可以用来对数据进行探索性分析,探索数据的潜在结构,也可以用来做聚类等有趣的无监督学习任务。但是因子分析模型有个潜在的问题,那就是我们要事先确定潜在因子的个数,这往往是非常困难的,所以研究者们开发出了一种针对该问题的高效解决方法——IMIFA。
IMIFA(Infinite Mixtures of Infinite Factor Analysers)方法是一种用于数据聚类和降维的贝叶斯非参数模型,它结合了因子分析模型和聚类分析模型。这个方法的核心思想是在不需要预先确定聚类数量和潜在因子数量的情况下,自动地从数据中学习这些信息。这就为我们的建模提供了一种简便自动化的高效方法。
R包中的IMIFA提供了灵活高效的函数,用于拟合无限混合的无限因子分析器(IMIFA)及其相关模型。主要模型IMIFA本身通过潜在高斯模型进行贝叶斯非参数聚类。虽然该包提供了因子分析(FA)和因子分析器混合(MFA)模型的贝叶斯实现,但这些模型需要预先确定潜在因子的数量和/或成分的数量,这些数量必须保持不变。IMIFA模型的主要优势在于a) 模型搜索大幅减少,b) 这些数量会自动估计,c) 潜在因子的数量允许是特定于聚类的,d) 可以量化聚类数量和聚类特定因子数量的不确定性。
通常情况下,我们会在不同的聚类数量和因子数量范围内运行FA或MFA模型,并选择最优化某些模型选择标准的组合。IMIFA则通过自动选择聚类数量和/或特定于聚类的潜在因子,实现了基于贝叶斯非参数模型的聚类,使用因子分析协方差结构,无需使用模型选择标准。
IMIFA模型的主要特点包括因子载荷的乘法Gamma过程缩小先验,允许理论上无限多个因子(这也可以在MIFA环境中使用,例如,其中聚类数量固定但特定于聚类的因子被估计),自适应Gibbs采样器动态截断无限载荷矩阵,以及使用切片高效抽样的Dirichlet过程先验,允许理论上无限多个聚类。还提供了工具,用于获取这些先验的合理超参数。
在生物信息学中,IMIFA方法可以用于处理复杂的生物数据,例如基因表达数据、蛋白质互作网络、基因组学数据等。以下是将IMIFA方法应用于生物信息学的一些关键点:
-
基因表达数据的聚类: 对于基因表达数据,IMIFA可以用于识别不同的基因表达模式,而无需预先设定基因簇的数量。这对于发现不同的生物学子群(例如,在肿瘤中识别不同的分子亚型)非常有用。
-
功能注释和通路分析: IMIFA可以用于将基因组学数据(如基因-基因互作网络或基因表达谱)中的基因分组到具有相似功能或参与相似生物学通路的簇中。这有助于揭示生物学过程中的关键调控模块和通路。
-
蛋白质互作网络分析: 在研究蛋白质互作网络时,IMIFA可以帮助识别具有相似结构或功能的蛋白质簇。这有助于理解蛋白质相互作用网络的模块化结构。
-
降维和可视化: IMIFA还可以用于将高维生物数据降维到更低维度,以便于可视化和解释。通过自动确定潜在因子数量,可以更好地捕获数据的结构。
-
不确定性建模: IMIFA方法还允许在聚类和因子分析中建模不确定性,这在生物信息学中是非常重要的,因为生物学数据通常具有噪声和不确定性。
-
……
IMIFA方法在生物信息学中具有广泛的应用潜力,特别是在处理复杂的高维生物数据时,它可以帮助我们自动地发现数据中的潜在结构和模式,而无需事先的假设或参数设置。这使得它成为生物信息学研究中的有力工具,有助于更好地理解生物学系统的复杂性。那么下面,就让小果带大家揭下IMIFA方法的面纱。
公众号后台回复“111″,领取代码,代码编号:231018
下载并导入R包
IMIFA方法的核心思想是结合因子分析和聚类分析,以发现数据中的隐藏模式和结构。它不仅能够自动地确定聚类数量,还可以在每个聚类中自动估计潜在因子的数量。这使得IMIFA方法特别适用于处理复杂的高维数据,如基因表达数据、蛋白质互作网络等。要使用IMIFA方法,我们需要首先下载并导入R包IMIFA。
install.packages("devtools")devtools::install_github('Keefe-Murphy/IMIFA')library(IMIFA)
数据准备和模型拟合
要使用IMIFA方法,我们首先需要准备数据,并使用mcmc_IMIFA()函数拟合模型。这个函数提供了灵活的MCMC采样器,用于估计模型的参数和隐变量。这个函数是IMIFA方法的核心,提供了自适应的Gibbs/Metropolis-within-Gibbs采样器,用于非参数模型的基于模型的聚类。它允许使用IMIFA系列模型进行模型基于聚类,其中包括了因子分析、有限混合、过拟合混合以及使用多项式Gamma过程(MGP)缩小先验的无限因子模型等。该函数创建了一个’IMIFA’类的原始对象,用户可以使用get_IMIFA_results函数从中提取最优/模态模型。在模型拟合过程中,可以指定各种参数,如聚类数量、潜在因子数量等。
在这个示例中,我们首先使用Mixture of Factor Analysers模型(MFA)拟合了一个以单位尺度的橄榄油数据。可以根据需要指定聚类数量(range.G)、潜在因子数量(range.Q)等参数。(当然,在IMIFA方法中,通常可以忽略)还可以选择是否对唯一性进行约束,以及是否存储潜在因子得分等。n.iters指定了最大的迭代次数,verbose指定了是否需要打印进度。
# 准备数据(橄榄油数据)data(olive)?mcmc_IMIFA# 拟合IMIFA模型simMFA <- mcmc_IMIFA(olive, method="MFA", n.iters=10000, range.G=3:6, range.Q=0:3, centering=FALSE,scaling="unit", uni.type="isotropic", score.switch=FALSE)#MFA方法,需要指定很多参数simMIFA <- mcmc_IMIFA(olive, method="MIFA", n.iters=10000, centering=TRUE,range.G=1:3, z.init="kmeans")#MIFA方法,指定的参数较MFA略少simOMIFA <- mcmc_IMIFA(olive, method="OMIFA", n.iters=10000, range.G=10, learn.alpha=TRUE,nu=3, alpha.d1=3.5, alpha.d2=7, prop=0.8, epsilon=0.01)#OMIFA方法,效果较好,但参数较多simIMIFA <- mcmc_IMIFA(olive, method="IMIFA", n.iters=50000, verbose=TRUE) #IMIFA方法,推荐使用
根据小果的运行结果可以发现,IMIFA模型的运行时间较长,可以通过改变迭代次数来进行运行时间的缩短,但是相对应的,更大的迭代次数意味着更准确的结果。
提取并分析结果
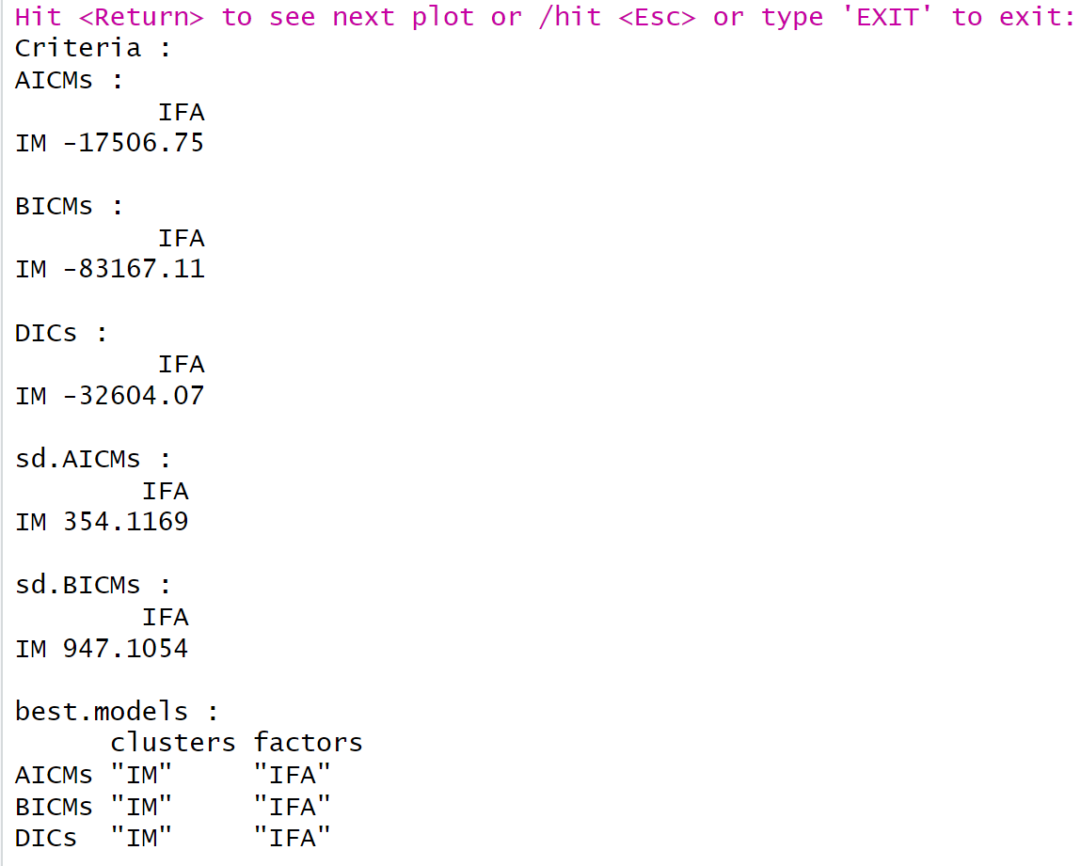
在提取IMIFA模型家族中的模型结果、进行后验推断和计算性能指标时,可以将mcmc_IMIFA函数的输出传递给get_IMIFA_results()函数。该函数可以根据我们选择的模型选择标准,从MCMC样本中找到最优的G(聚类数量)和Q(每个聚类的潜在因子数量)的组合,并准备相应的结果。如果上述示例中提供了simMFA模型,该函数将找到最优的G和Q值,并准备该模型的结果。如果提供了simIMIFA模型,该函数将找到G和每个qg(每个聚类的潜在因子数量)的模态估计,并相应地准备结果。
这个函数可以以较小的计算成本重新运行,以提取由mcmc_IMIFA使用的采样器探索的不同模型,而无需重新运行模型本身。可以轻松生成使用不同聚类数量和不同潜在因子数量(如果模型访问了这些数量)或使用不同模型选择标准(如果需要)的新结果对象。该函数还执行标签切换的后处理校正,以及后处理的Procrustes旋转,以确保合理的后验参数估计,计算误差指标,并构建可信区间、平均相似矩阵和后验混淆矩阵。欲了解有关各种函数参数的更多帮助,请键入?get_IMIFA_results以查看函数的帮助手册。
如果我们想选择最佳的MFA模型,只需键入以下命令:
resMFA <- get_IMIFA_results(simMFA)如果我们想要探索3个聚类的解决方案,并希望通过其他标准选择潜在因子数量,可以尝试以下命令:
resMFA2 <- get_IMIFA_results(simMFA, G=3, criterion="aic.mcmc")现在,让我们从上面的IMIFA运行中提取结果,以便后续进行详细的可视化分析。尽管IMIFA模型不需要模型选择标准,但提取结果的语法与前面的示例完全相同。不过,这次还通过计算N*N相似度矩阵的平均值来总结聚类情况,以便稍后进行可视化。
#提取结果resIMIFA <- get_IMIFA_results(simIMIFA, z.avgsim=TRUE)summary(resIMIFA, MAP=TRUE)

结果可视化
在这一部分,小果将带大家学习如何使用Results_IMIFA类的对象(例如resIMIFA)来可视化IMIFA方法和相关模型的输出和参数。我们可以调用plot函数来访问专门用于可视化IMIFA模型输出和感兴趣参数的函数。在这里,我们将着重介绍plot.meth和param这两个关键参数,它们分别指定要生成的图表类型和感兴趣的参数。

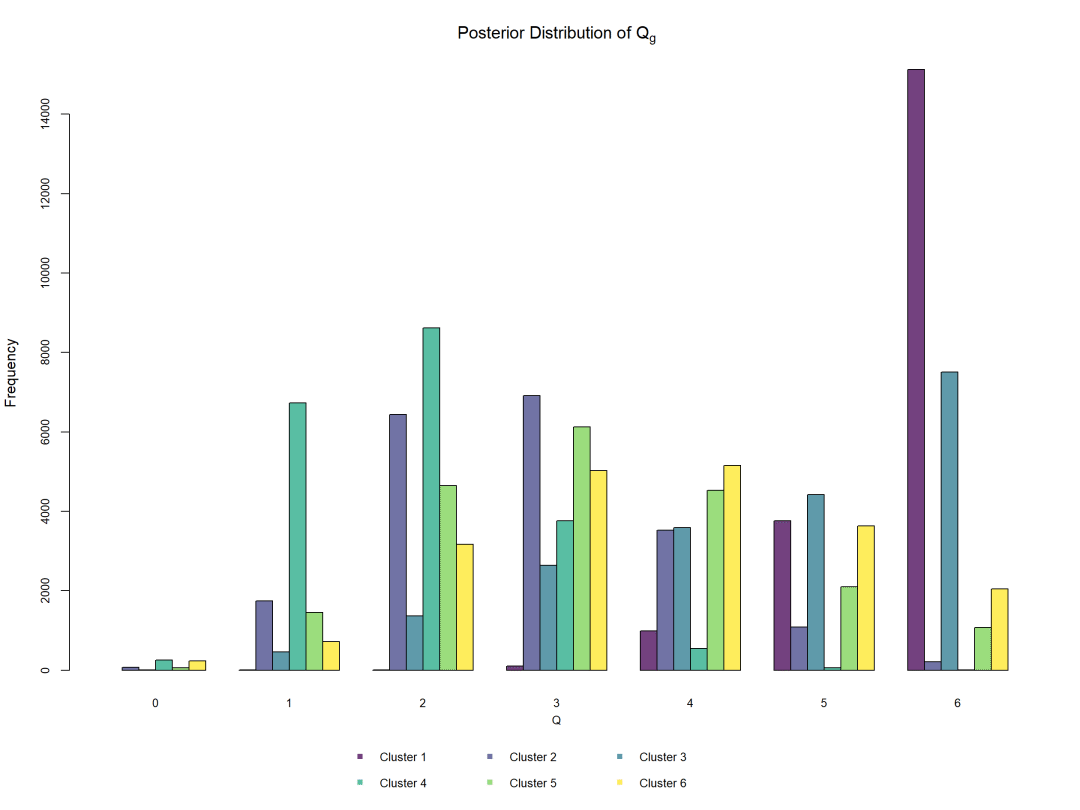

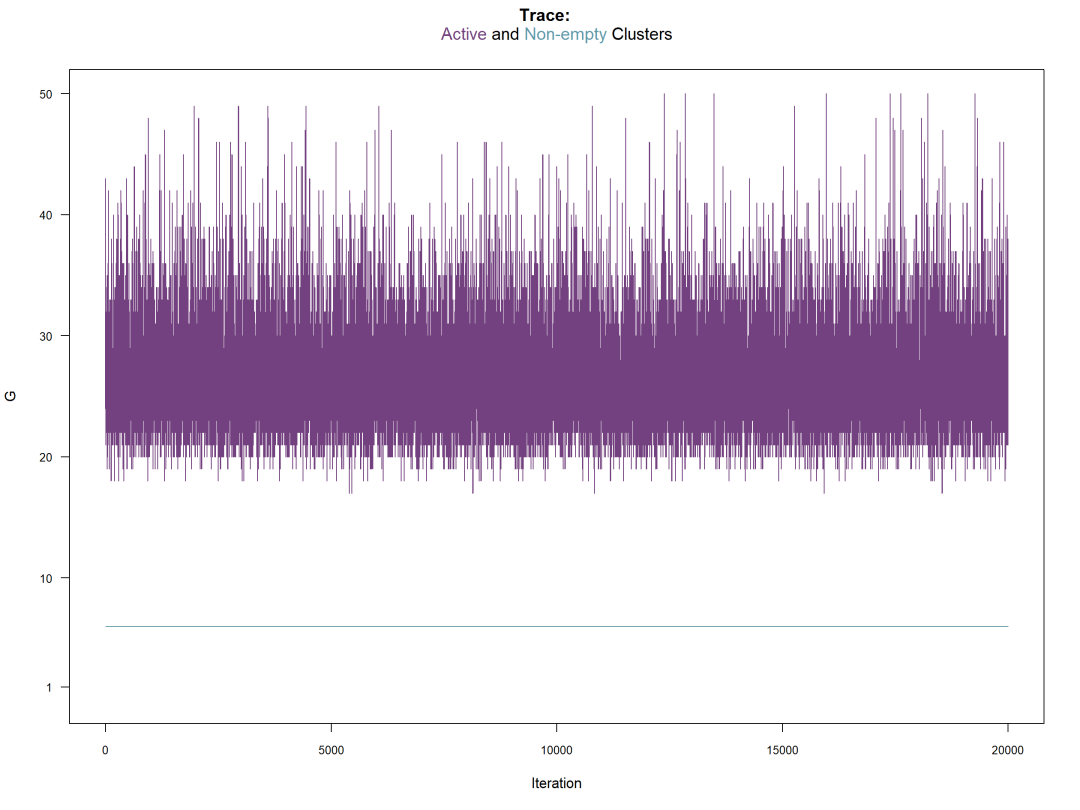
我们可以根据已知的集群标签检查集群性能。大家注意,聚类标签也可能已经提供 get_IMIFA_results,但绘图允许根据新标签评估聚类性能,而无需再次提取完整的结果对象。更具体地说,下面的代码允许我们可视化聚类不确定性(提供或不提供标签;事实上,当提供标签时,错误分类的观察结果会突出显示,否则不确定性超过聚类数量倒数的观察结果会突出显示) 。
plot(resIMIFA, plot.meth="zlabels", zlabels=olive$area, g=1)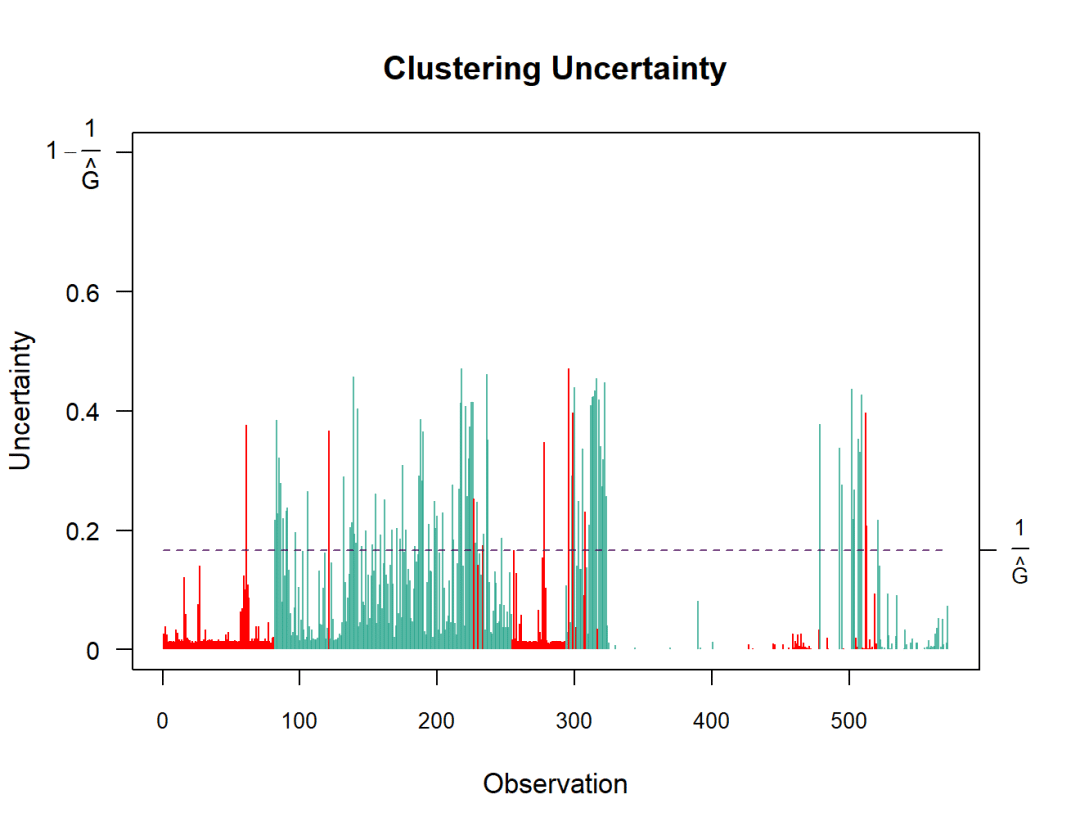
还有一些其他可视化方法,限于篇幅,小果就不向大家一一展示了,这里先向大家列出来,感兴趣的小伙伴记得自己动手试一试哦!
#其他可视化方法plot(resIMIFA, plot.meth="zlabels", zlabels=olive$area, g=2)plot(resIMIFA, plot.meth="zlabels", g=4)plot(resIMIFA, plot.meth="zlabels", g=5)plot(resIMIFA, plot.meth="means", param="means", mat=TRUE, g=1)plot(resIMIFA, plot.meth="trace", param="scores", mat=TRUE, ind=1)plot(resIMIFA, plot.meth="trace", param="scores", mat=TRUE, by.fac=TRUE, fac=2)plot(resIMIFA, plot.meth="means", param="loadings", heat.map=TRUE, g=1)ot(resIMIFA, plot.meth="parallel.coords", param="uniquenesses")plot(resIMIFA, plot.meth="errors", g=1)plot(resIMIFA, plot.meth="all", param="alpha")plot(resIMIFA, plot.meth="all", param="discount", partial=TRUE)
那么IMIFA方法的学习就到这里结束了,小果要和大家说再见咯,一定要自己练习一下哦,同时如果大家想要继续了解更多有关R语言内容可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~
往期推荐


