CCPlotR:可视化细胞间相互作用(scRNA-seq)
 今天小果要分享的新鲜好包是2023年9月20日发表于Bioinformatics Advances上用于可视化细胞间相互作用的CCplotR!
今天小果要分享的新鲜好包是2023年9月20日发表于Bioinformatics Advances上用于可视化细胞间相互作用的CCplotR!

在生物信息学领域,研究细胞间的相互作用对于理解复杂的生物学过程至关重要。为了帮助小伙伴们更好地分析和可视化这些相互作用,小果向大家介绍这一款名为CCPlotR的R包。它专为处理和可视化从单细胞基因表达数据预测出的细胞间相互作用而设计,只需输入预测的交互作用表即可生成一系列完整的、可用于发表的图形,如热图、点图、环形图和网络图等。对于从事细胞间相互作用研究的小伙伴们来说,CCPlotR无疑是一款非常实用的工具。
CCPlotR可以画出多种类型的图,用于可视化来自Liana、CellPhoneDB、NATMI等多个工具的结果。所需输入仅为一个包含“source”、“target”、“ligand”、“receptor”和“score”这些列的DataFrame。应类似于以下形式:
|
source |
|
B |
|
B |
|
CD8 T |
|
B |
|
NK |
|
B |
对于某些图,还可以选择显示每种细胞类型中的配体和受体的表达。对于这些图,需要第二个数据框,它包含每种细胞类型中每个基因的平均表达值,看起来应该像这样:
|
cell_type |
|
B |
|
B |
|
B |
|
B |
|
B |
|
B |
该包附带示例数据集(‘toy_data’, ‘toy_exp’),大家可以看看~
安装
可以通过以下命令来安装R包:
# install development versiondevtools::install_github("Sarah145/CCPlotR")# or install from Bioconductorif (!requireNamespace("BiocManager", quietly =TRUE)) {install.packages("BiocManager")}BiocManager::install("CCPlotR")
公众号后台回复“111”领取本篇代码、基因集或示例数据等文件;
文件编号:231208
果粉福利:生信人必备神器——服务器
平时生信分析学习中有要的小伙伴可以联系小果租赁,粉丝福利都是市场超低价格,赶快找小果领取免费的试用账号吧!
可视化类型
该包包含用于生成六种类型的图的函数:cc_heatmap,cc_dotplot,cc_network,cc_circos,cc_arrow和cc_sigmoid。下面是每种图形类型的一些示例。
热图(cc_heatmap)
此函数可以生成四种不同样式的热图。选项A仅显示每对细胞类型之间的相互作用总数,而选项B显示参与每个相互作用的配体、受体和细胞类型以及它们的分数。为了避免图形混乱,选项B仅显示一小部分最相关的相互作用。还有一项选项,以CellPhoneDB和Liana等流行的细胞间相互作用预测工具的风格生成热图。
library(CCPlotR)data(toy_data, toy_exp, package ='CCPlotR')cc_heatmap(toy_data)cc_heatmap(toy_data, option ='B', n_top_ints =10)cc_heatmap(toy_data, option ='CellPhoneDB')
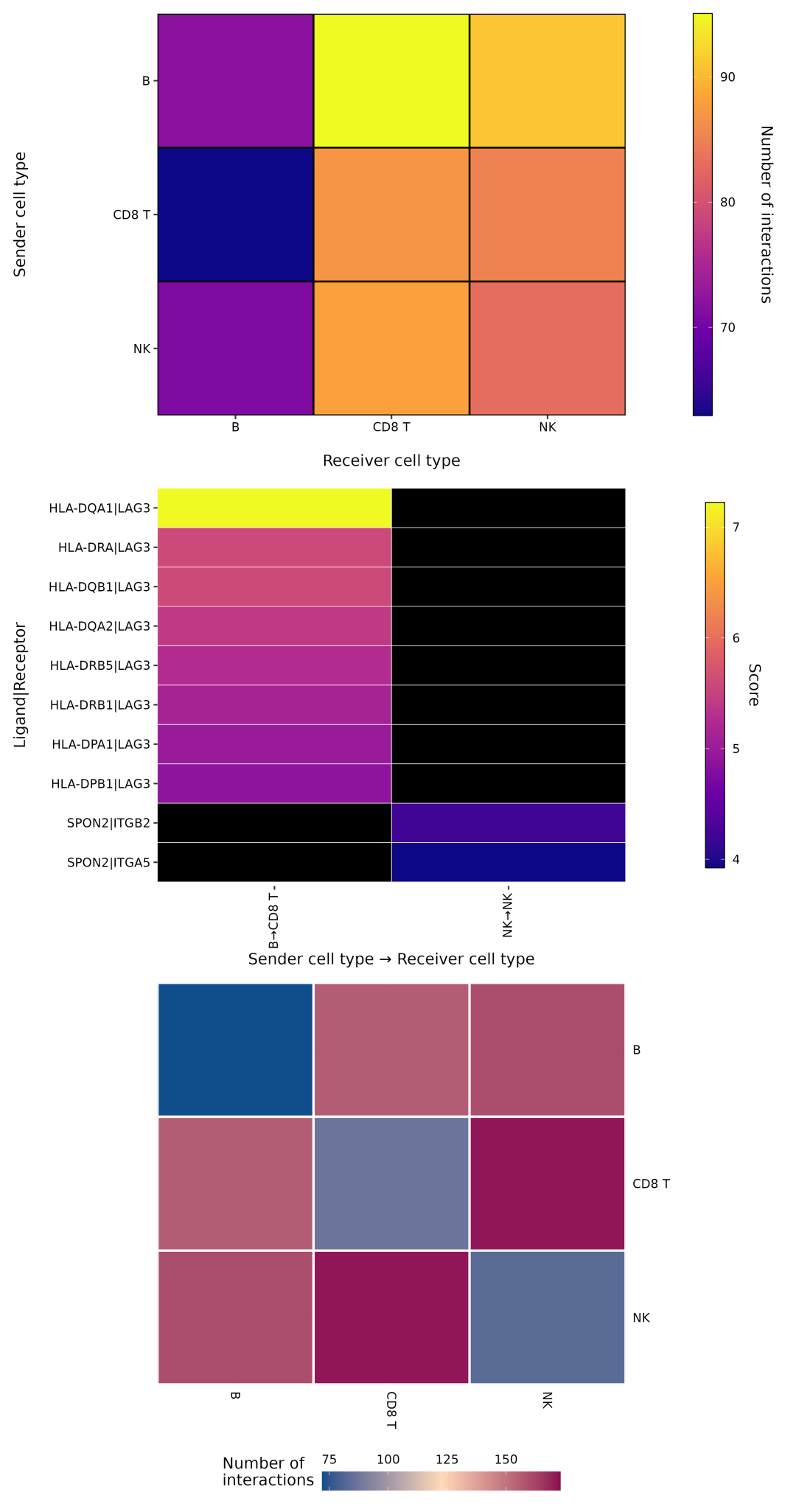
 点图(cc_dotplot)
点图(cc_dotplot)
该函数可以生成四种不同样式的点图。选项A仅显示每种细胞类型对之间的相互作用总数,而选项B显示参与每种相互作用的配体、受体和细胞类型以及它们的相互作用分数。为避免图形混乱,在选项B中仅显示一小部分高相互作用。此外,还有一种选项可以生成类似于广受欢迎的细胞间相互作用预测工具CellPhoneDB和Liana的点图样式。
cc_dotplot(toy_data)cc_dotplot(toy_data, option ='B', n_top_ints =10)cc_dotplot(toy_data, option ='Liana', n_top_ints =15)
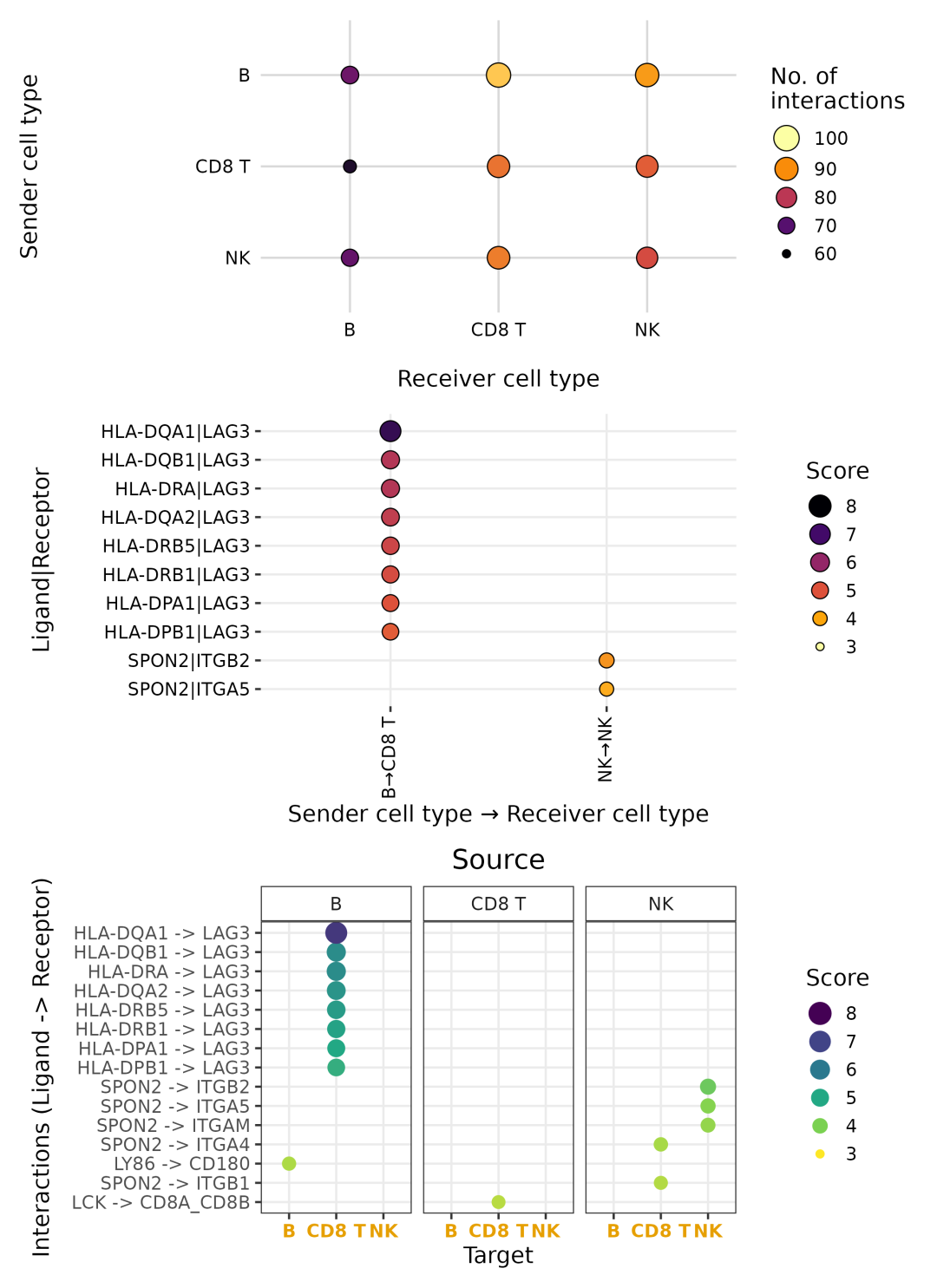
网络(cc_network)
此函数可以生成两种不同类型网络图。在选项A中,节点代表细胞类型,边权重对应于给定细胞类型对之间的总交互数。在选项B中,节点是配体和受体基因,依据表达它们的细胞类型进行着色。对于选项B,仅显示少量最相关的交互作用以避免图形混乱。
cc_network(toy_data)cc_network(toy_data, colours =c('orange', 'cornflowerblue', 'hotpink'), option ='B')
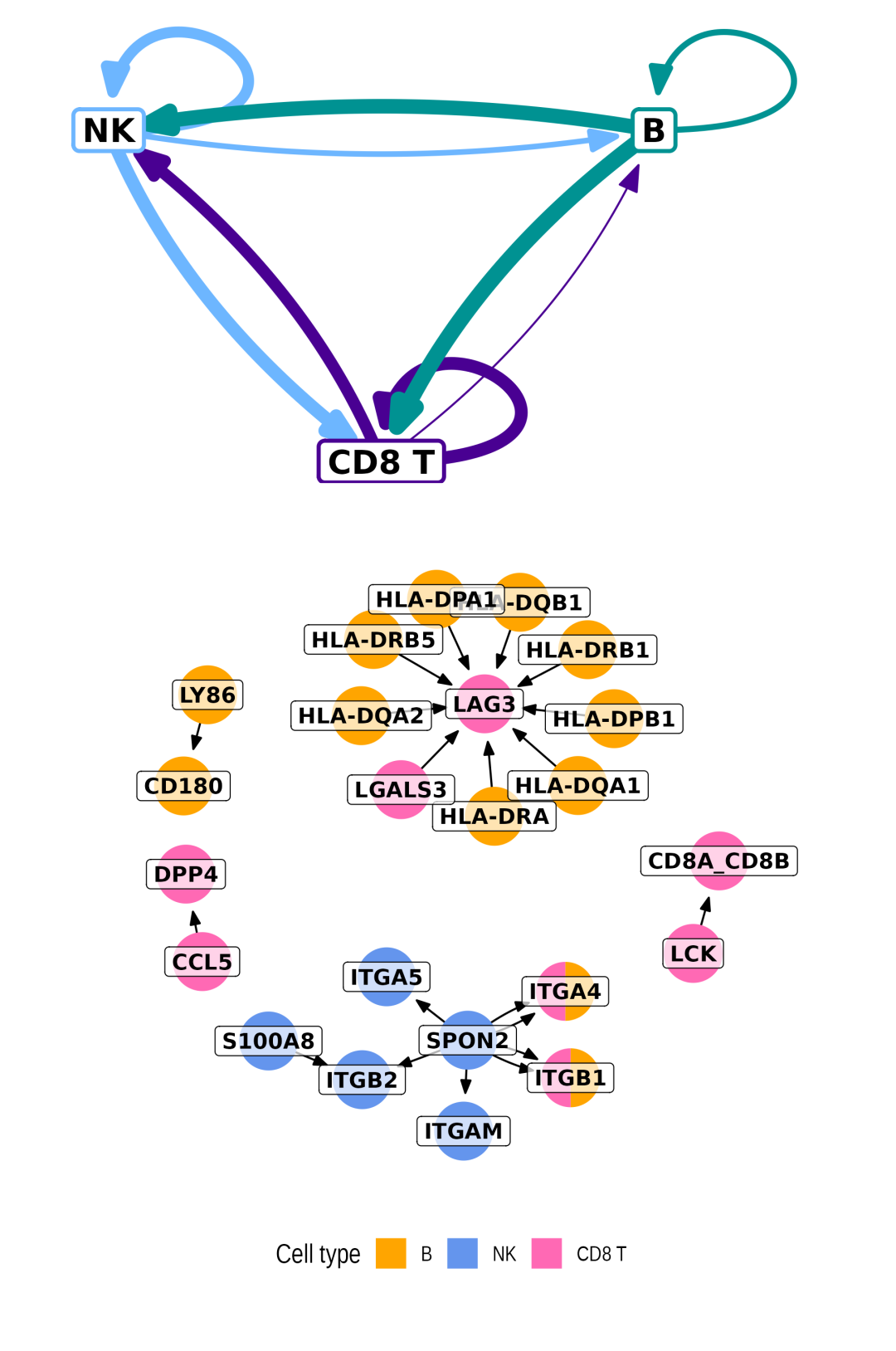
弦图 (cc_circos)
该函数可以生成三种不同类型的弦图。选项A生成一个弦图,其中链接的宽度表示每种细胞类型对之间的总相互作用次数。选项B生成一个弦图,显示了相互作用顶部分涉及的配体、受体和细胞类型。选项C扩大了选项B的内容,还显示了每种细胞类型中配体和受体基因的平均表达水平。在选项B和选项C中,链接的权重表示相互作用的得分。
cc_circos(toy_data)cc_circos(toy_data, option ='B', n_top_ints =10)cc_circos(toy_data, option ='C', n_top_ints =15, exp_df = toy_exp, cell_cols =c(`B`='hotpink', `NK`='orange', `CD8 T`='cornflowerblue'), palette ='PuRd')
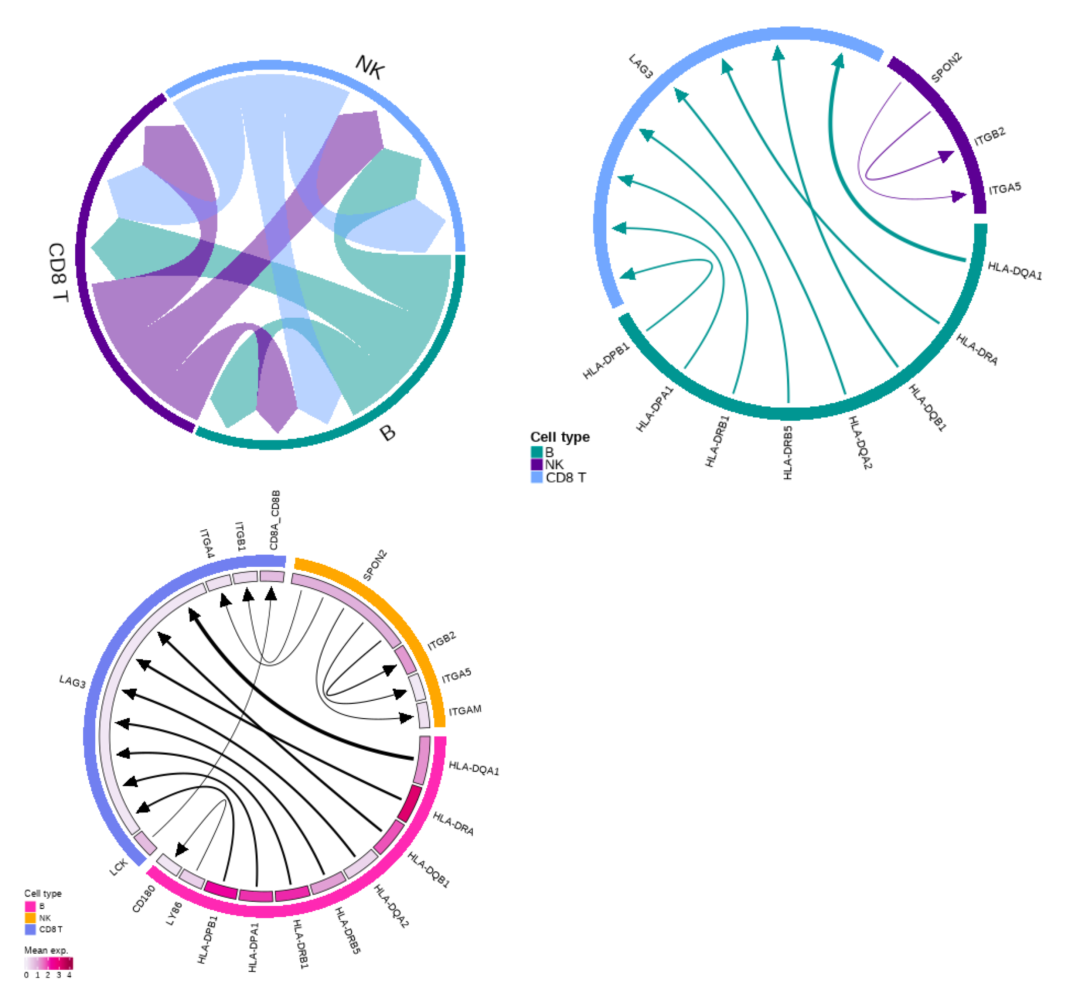
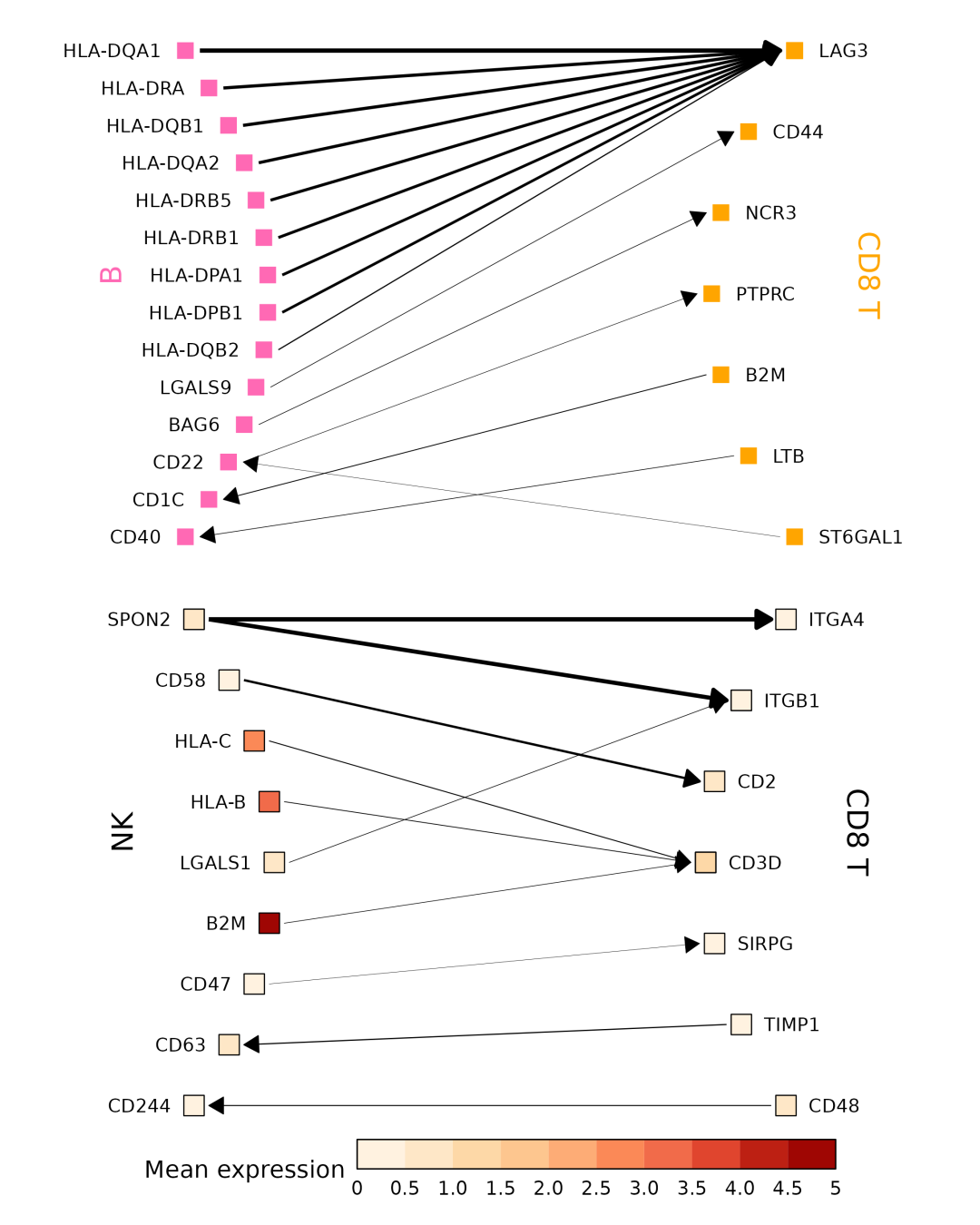
对箭头图(cc_arrow)
此函数生成显示给定细胞类型之间相互作用的图表。选项A仅显示一对细胞类型之间相互作用的配体/受体,而选项B还显示每个细胞类型中配体/受体基因的表达情况。在这两个选项中,箭头的粗细代表相互作用的分数。
cc_arrow(toy_data, cell_types =c('B', 'CD8 T'), colours =c(`B`='hotpink', `CD8 T`='orange'))cc_arrow(toy_data, cell_types =c('NK', 'CD8 T'), option ='B', exp_df = toy_exp, n_top_ints =10, palette ='OrRd')
Sigmoid图(cc_sigmoid)
此函数使用ggbump R包中的geom_sigmoid函数绘制部分相互作用,将发送细胞中的配体与接收细胞中的受体连接起来。
cc_sigmoid(toy_data)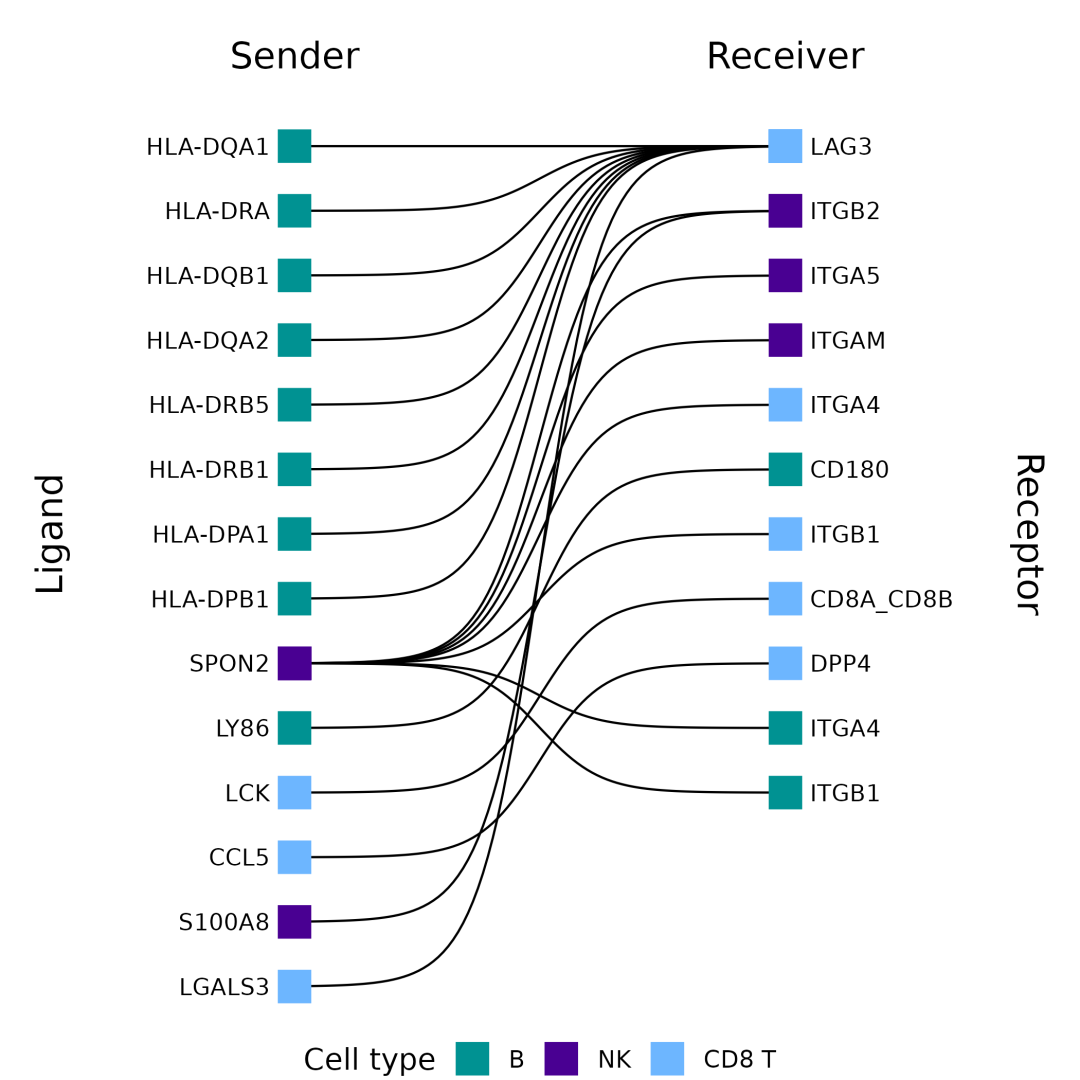
参考文献
[1] Sarah Ennis, Pilib Ó Broin, Eva Szegezdi, CCPlotR: an R package for the visualization of cell–cell interactions, Bioinformatics Advances, Volume 3, Issue 1, 2023, vbad130, https://doi.org/10.1093/bioadv/vbad130


往期推荐