速度看过来!小果教你学习RNA速度分析R包velociraptor!
 简介
简介
首先小果简单介绍下什么是RNA速度(RNA velocity)。RNA丰度是单个细胞状态的有力指标,单细胞 RNA 测序可以以高定量精度、灵敏度和通量揭示 RNA 丰度。然而,这种方法仅捕获某个时间点的静态快照,这对分析胚胎发生或组织再生等时间分辨现象提出了挑战。在2018年La Manno等人引入了RNA速度(RNA velocity)的概念,该项研究发表在了Nature上。通过将测量结果与潜在的mRNA剪接动力学联系起来,探索定向轨迹的推断:特定基因的转录诱导导致(新转录的)前体未剪接mRNA的增加,而相反,转录的抑制或缺失导致未剪接mRNA的减少。因此,通过将未剪接的mRNA与成熟的剪接mRNA进行区分,可以近似地得到mRNA丰度的变化,也就是mRNA的时间导数,即RNA速度。
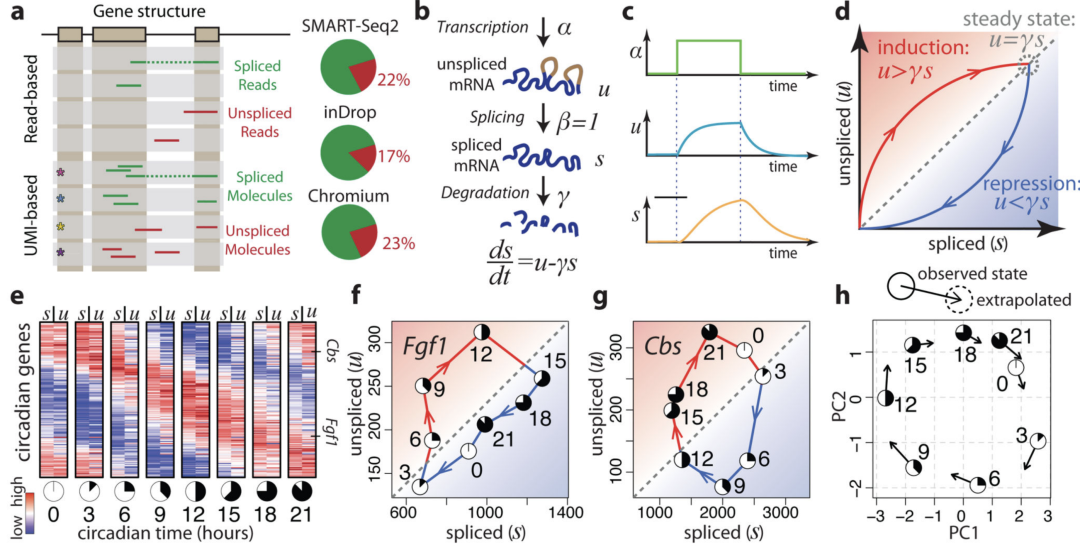
未剪接和剪接 mRNA 之间的平衡可预测细胞状态进展
RNA 速度是一种高维向量,可以预测单个细胞在数小时的时间尺度上的未来状态。那RNA速度有什么用呢?RNA 速度将极大地帮助分析发育谱系和细胞动力学,RNA 速度分析使我们能够使用转录动力学的数学模型来推断 scRNA-seq 实验中未直接观察到的转录动力学。 我们可以使用 RNA 速度来确定某个靶基因在给定的靶细胞群中是否被诱导或抑制。 此外,我们可以通过伪时间轨迹推断这些信息来预测细胞命运的决定。
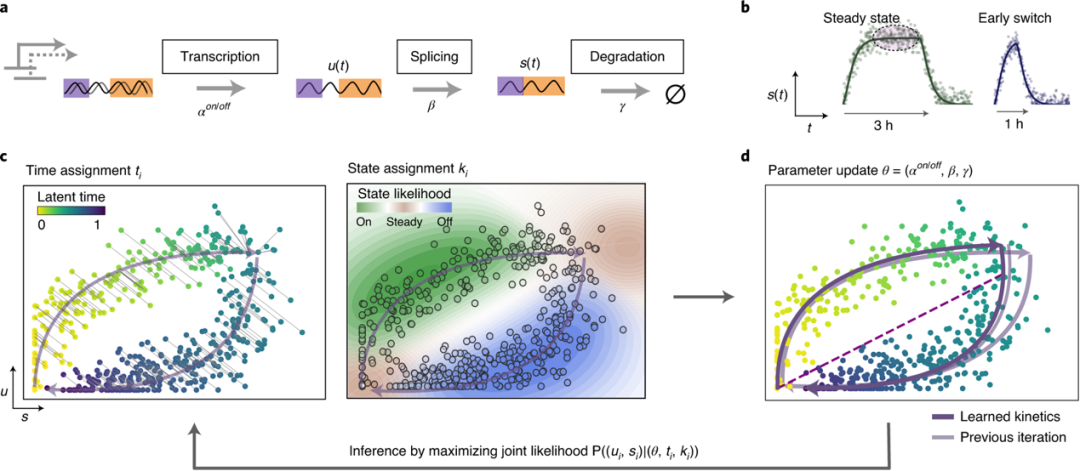
RNA速度动态模型
目前比较常用的用来作为RNA速度分析的是基于python的scVelo, scVelo是基于python的RNA速度分析工具,但是我们很多小伙伴更喜欢使用R语言,所以呢今天我们来介绍下刚发表的基于R语言的RNA速度分析软件包velociraptor,下面跟小果一起学习吧!
该软件包在 Bioconductor SingleCellExperiment 数据结构和 scvelo Python 软件包之间提供了一个轻量级接口,用于 RNA 速度计算。该界面与许多其他 SingleCellExperiment 兼容功能的界面相当,允许用户将 RNA 速度计算插入现有的 Bioconductor 分析框架中。为了进行演示,我们将使用 Hermann 等人的数据集(2018),通过 scRNAseq 包提供。该数据集包含 2,325 个小鼠生精细胞的剪接和未剪接 UMI 计数的基因估计。
导入数据
library(scRNAseq)sce<-HermannSpermatogenesisData()
取样示范
完整的数据集需要高达 12 GB 的内存才能用于此插图中呈现的示例用法。出于演示目的,小果将数据集下采样到前 500 个单元格。如果您有足够的内存,请随意跳过此下采样步骤。
#取样500个细胞sce<-sce[, 1:500]
 基本工作流程
基本工作流程
选择前2000个基因。
library(scuttle)sce<-logNormCounts(sce, assay.type=1)library(scran)dec<-modelGeneVar(sce)top.hvgs<-getTopHVGs(dec, n=2000)
scvelo() 使用稳态方法来估计速度,但也可以通过修改模式参数来使用 scvelo 中实现的随机和动态模型。
library(velociraptor)velo.out<-scvelo(sce, subset.row=top.hvgs, assay.X="spliced")velo.out
在上面的调用中,我们使用”拼接”计数矩阵作为典型外显子计数矩阵的代理。
library(scater)set.seed(100)sce<-runPCA(sce, subset_row=top.hvgs)sce<-runTSNE(sce, dimred="PCA")sce$velocity_pseudotime<-velo.out$velocity_pseudotimeplotTSNE(sce, colour_by="velocity_pseudotime")
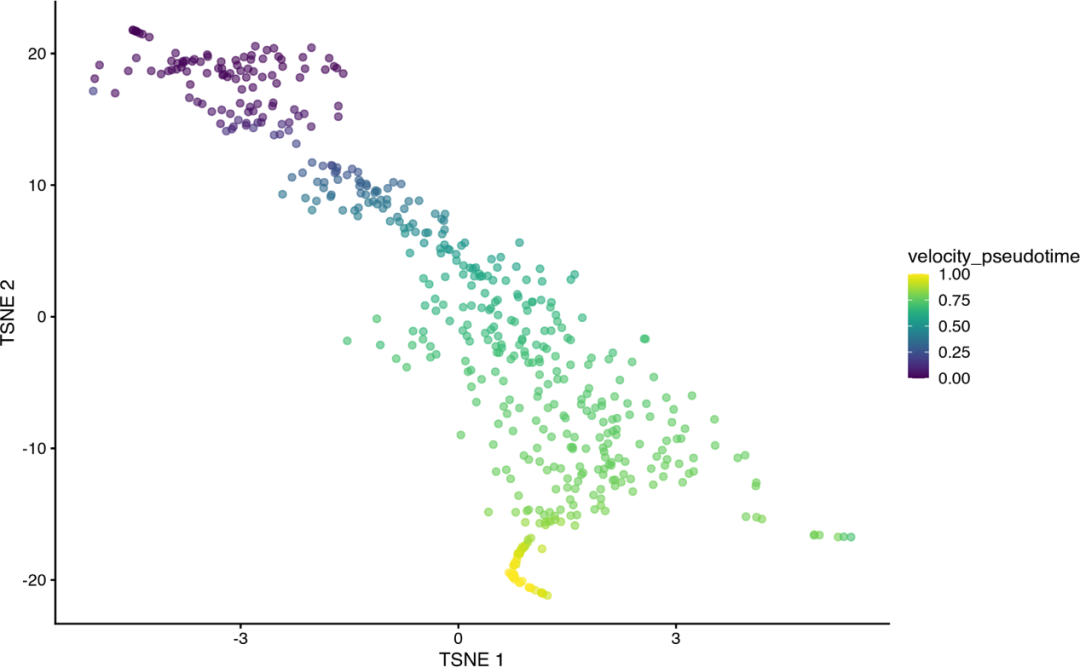
将速度向量嵌入到我们想要的低维空间中也很简单,如下面的 t-SNE 坐标: 这种方法使用基于网格的方法将每个单元的向量汇总为当地代表,以便有效地可视化。
embedded<-embedVelocity(reducedDim(sce, "TSNE"), velo.out)grid.df<-gridVectors(sce, embedded, use.dimred="TSNE")library(ggplot2)plotTSNE(sce, colour_by="velocity_pseudotime") +geom_segment(data=grid.df, mapping=aes(x=start.1, y=start.2,xend=end.1, yend=end.2, colour=NULL), arrow=arrow(length=unit(0.05, "inches")))
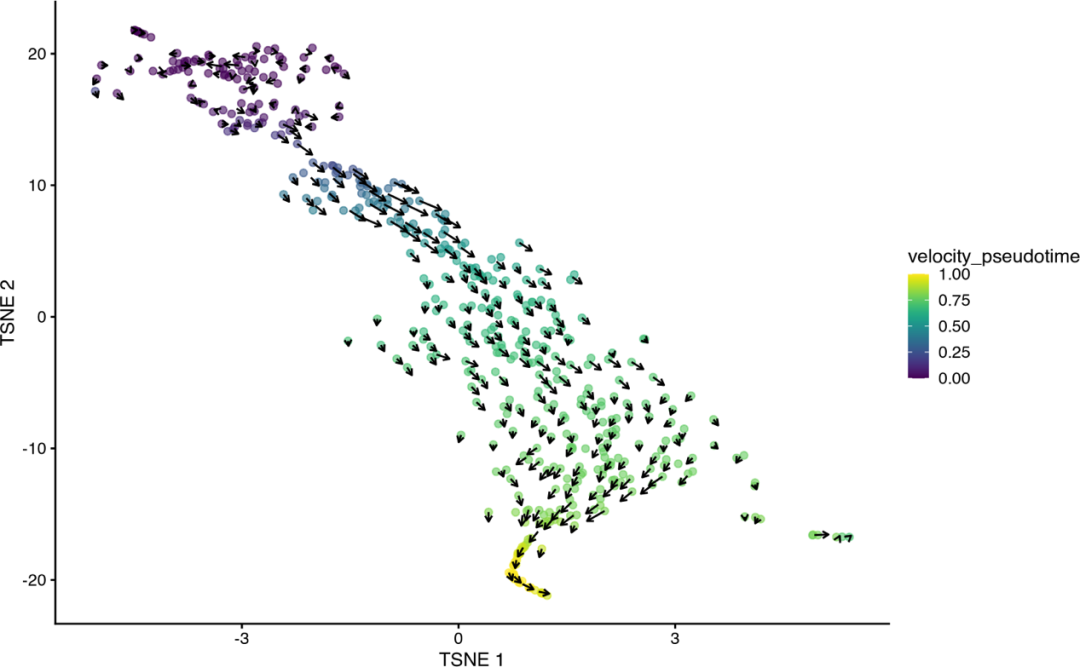
这样,我们轻松得到了RNA速度矢量图。RNA 速度以真实转录动力学为基础的事实有望为我们理解细胞分化过程中基因表达空间的动态提供更坚实的定量基础。我们设想未来的流形学习算法能够基于 RNA 速度同时拟合流形和该流形上的动力学。RNA速度已经能够对整个生物体的动态过程进行详细研究,并将极大地促进谱系分析,特别是在人类胚胎中。小伙伴们还不赶紧来学习?好了,以上就是小果今天分享的内容,希望有帮到大家。想要了解更多生信进展?请关注生信果! 

往期推荐