前言
听到很多小伙伴们抱怨生信分析发文章实在太难了!!卷完工作量卷新方法,卷完新方法卷新算法!实在是苦不堪言。看到别人已经站在了食物链的顶端,自己却还在龟步前行,闷闷不乐、愁眉苦脸!听到大家的苦恼大海哥来帮助大家了。下面看看双注意力机制新型图神经网络与空间多组学碰撞会擦出什么样的火花?不同的组学模式与空间信息相如何进行结合,以获得所研究组织的全面分子视图?SpatialGlue是一种具有双注意力机制的图神经网络模型,它通过空间位置和组学测量的组内整合以及跨组学整合来破译空间域。看看SpatialGlue能不能让你灵光一现呢?
练了十年生信分析的大海哥对于生信分析知识已经如鱼得水,相信有大海哥的加持一定能让大家学会!没有时间学习且有生信分析的小伙伴们快来滴滴大海哥噢!从分析到可视化直到你满意为止!
大海哥在这里给大家送福利了,有需要服务器的小伙伴们,欢迎大家后台私信大海哥哦,保证服务器的性价比最高哦!
代码教程
在本教程中,大海哥给大家演示了如何应用 SpatialGlue 来集成 Stereo-CITE-seq数据以获得细粒度聚类。Stereo-CITE-seq可共同检测免疫器官中的mRNA和蛋白质我们分析了小鼠胸腺数据集。我们从华大基因收集了数据。根据标记基因和蛋白质,我们手动将组织注释为 8 种细胞类型。演示数据请私信大海哥获取哦!
1..安装及加载SpatialGlue包
conda create -n SpatialGlue python=3.8
conda activate SpatialGlue
pip install SpatialGlue
##导入相关的包
import os
import torch
import pandas as pd
import scanpy as sc
import SpatialGlue
# 环境配置。SpatialGlue包可以用CPU或GPU实现。强烈推荐GPU加速以提高效率。
device = torch.device(‘cuda:2’ if torch.cuda.is_available() else ‘cpu’)
# R的位置,这是“mclust”聚类算法所需要的。请将下面的路径替换为本地R安装路径
os.environ[‘R_HOME’] = ‘/scbio4/tools/R/R-4.0.3_openblas/R-4.0.3’
2.加载数据
# 读取数据
file_fold = ‘/home/yahui/anaconda3/work/SpatialGlue_revision/data/Dataset3_Mouse_Thymus1/’ #please replace ‘file_fold’ with the download path
adata_omics1 = sc.read_h5ad(file_fold + ‘adata_RNA.h5ad’)
adata_omics2 = sc.read_h5ad(file_fold + ‘adata_protein.h5ad’)
adata_omics1.var_names_make_unique()
adata_omics2.var_names_make_unique()
#指定数据类型
data_type = ‘Stereo-CITE-seq’
#固定随机种子
from SpatialGlue.preprocess import fix_seed
random_seed = 2022
fix_seed(random_seed)
3. 预处理数据
SpatialGlue 对转录组学和蛋白质数据采用标准的预处理步骤。具体而言,对于转录组学数据,通过SCANPY包对基因表达计数进行对数转换并按文库大小进行归一化。选择前 3,000 个高度可变基因 (HVG) 作为 PCA 的输入,以进行降维。为确保输入维度与蛋白质数据一致,将保留前 k(蛋白质数)主成分并用作模型的输入。这 使用 CLR(中心对数比)对蛋白质表达计数进行规范化。PCA 降维后的所有主成分均用作模型的输入。
from SpatialGlue.preprocess import clr_normalize_each_cell, pca
# RNA
sc.pp.filter_genes(adata_omics1, min_cells=10)
sc.pp.filter_cells(adata_omics1, min_genes=80)
sc.pp.filter_genes(adata_omics2, min_cells=50)
adata_omics2 = adata_omics2[adata_omics1.obs_names].copy()
sc.pp.highly_variable_genes(adata_omics1, flavor=”seurat_v3″, n_top_genes=3000)
sc.pp.normalize_total(adata_omics1, target_sum=1e4)
sc.pp.log1p(adata_omics1)
adata_omics1_high = adata_omics1[:, adata_omics1.var[‘highly_variable’]]
adata_omics1.obsm[‘feat’] = pca(adata_omics1_high, n_comps=adata_omics2.n_vars-1)
# 蛋白质
adata_omics2 = clr_normalize_each_cell(adata_omics2)
adata_omics2.obsm[‘feat’] = pca(adata_omics2, n_comps=adata_omics2.n_vars-1)
4. 构造邻域图
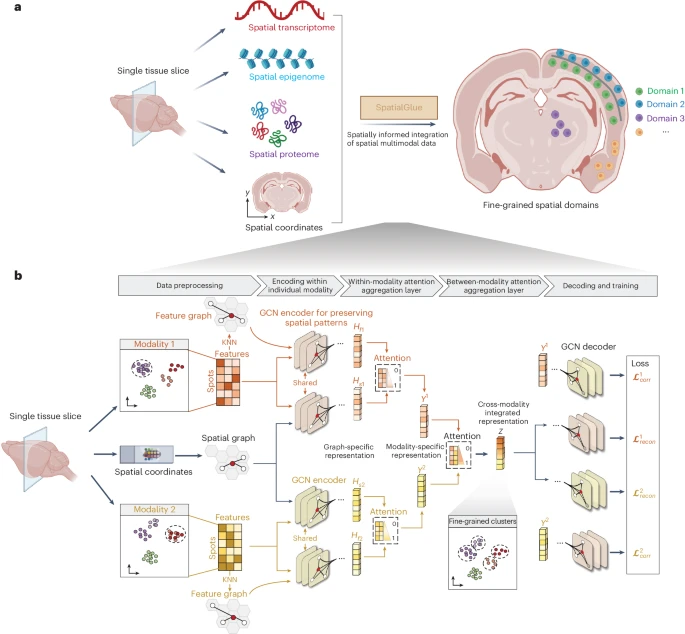
为了保持具有相同细胞类型但在空间上不相邻的斑点之间的物理邻近性和表型邻近性,我们构建了两个相邻图,即空间图和特征图。空间图基于空间信息构建,特征图基于表达式剖面构建。
from SpatialGlue.preprocess import construct_neighbor_graph
data = construct_neighbor_graph(adata_omics1, adata_omics2, datatype=data_type)
5. 训练模型
SpatialGlue 模型旨在通过以空间感知方式自适应地整合不同组学模式的表达谱来学习集成的潜在表征。
SpatialGlue 模型旨在通过以空间感知方式自适应地整合不同组学模式的表达谱来学习集成的潜在表征。
- 潜在表征:
- ’emb_latent_omics1’:第一种组学模式的潜在表示。
- “emb_latent_omics2”:第二组学模式的潜在表示。
- “SpatialGlue”:通过结合表达式数据和空间位置信息来学习联合表示。
联合表示可用于下游分析,例如聚类、可视化或识别差异表达基因 (DEG)。
- 注意权重值:
- “alpha_omics1”:第一种组学模态的模态内注意力权重,解释了每个图对每个簇的贡献。
- ‘alpha_omics2’:第二组学模态的模态内注意力权重,解释每个图对每个簇的贡献。
- “alpha”:模态间注意力权重,解释每种模态对每个集群的贡献。
这些模态内和模态间注意力权重提供了对每个邻域图和模态对每个聚类的重要性的可解释见解。
# 定义模型
from SpatialGlue.SpatialGlue_pyG import Train_SpatialGlue
model = Train_SpatialGlue(data, datatype=data_type, device=device)
# 训练模型
output = model.train()
adata = adata_omics1.copy()
adata.obsm[’emb_latent_omics1′] = output[’emb_latent_omics1′]
adata.obsm[’emb_latent_omics2′] = output[’emb_latent_omics2′]
adata.obsm[‘SpatialGlue’] = output[‘SpatialGlue’]
adata.obsm[‘alpha’] = output[‘alpha’]
adata.obsm[‘alpha_omics1’] = output[‘alpha_omics1’]
adata.obsm[‘alpha_omics2’] = output[‘alpha_omics2’]
6. 跨组学整合分析
集成后,使用mclust进行聚类分析。在这里,我们提供了三种可选的聚类工具,包括 mclust、leiden 和 louvain。在我们的实验中,大海哥发现在大多数情况下,“mclust”算法在空间数据上的表现优于“leiden”和“louvain”。因此,大海哥建议使用“mclust”算法进行聚类。
# 我们将“mclust”默认设置为聚类工具。用户还可以选择“leiden”和“louvain”。
tool = ‘mclust’ # mclust, leiden, and louvain
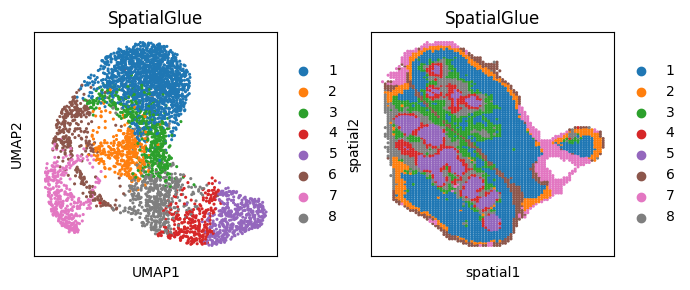
clustering(adata, key=’SpatialGlue’, add_key=’SpatialGlue’, n_clusters=8, method=tool, use_pca=True)
#可视化
import matplotlib.pyplot as plt
adata.obsm[‘spatial’][:,1] = -1*adata.obsm[‘spatial’][:,1]
fig, ax_list = plt.subplots(1, 2, figsize=(7, 3))
sc.pp.neighbors(adata, use_rep=’SpatialGlue’, n_neighbors=30)
sc.tl.umap(adata)
sc.pl.umap(adata, color=’SpatialGlue’, ax=ax_list[0], title=’SpatialGlue’, s=20, show=False)
sc.pl.embedding(adata, basis=’spatial’, color=’SpatialGlue’, ax=ax_list[1], title=’SpatialGlue’, s=20, show=False)
plt.tight_layout(w_pad=0.3)
plt.show()
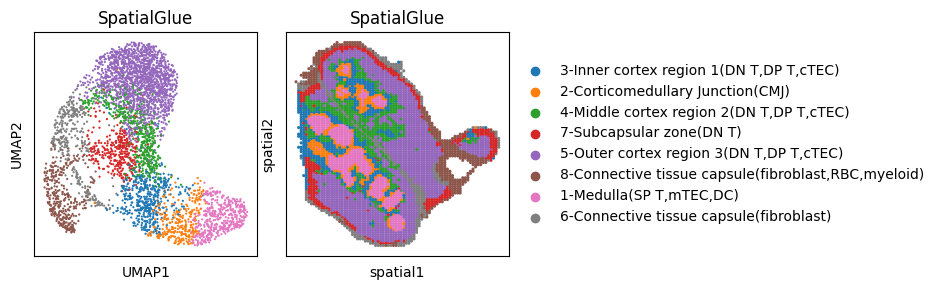
# 注释
adata.obs[‘SpatialGlue_number’] = adata.obs[‘SpatialGlue’].copy()
adata.obs[‘SpatialGlue’].cat.rename_categories({1: ‘5-Outer cortex region 3(DN T,DP T,cTEC)’,
2: ‘7-Subcapsular zone(DN T)’,
3: ‘4-Middle cortex region 2(DN T,DP T,cTEC)’,
4: ‘2-Corticomedullary Junction(CMJ)’,
5: ‘1-Medulla(SP T,mTEC,DC)’,
6: ‘6-Connective tissue capsule(fibroblast)’,
7: ‘8-Connective tissue capsule(fibroblast,RBC,myeloid)’,
8: ‘3-Inner cortex region 1(DN T,DP T,cTEC)’
}, inplace=True)
list_ = [‘3-Inner cortex region 1(DN T,DP T,cTEC)’,’2-Corticomedullary Junction(CMJ)’,’4-Middle cortex region 2(DN T,DP T,cTEC)’,
‘7-Subcapsular zone(DN T)’, ‘5-Outer cortex region 3(DN T,DP T,cTEC)’, ‘8-Connective tissue capsule(fibroblast,RBC,myeloid)’,
‘1-Medulla(SP T,mTEC,DC)’,’6-Connective tissue capsule(fibroblast)’]
adata.obs[‘SpatialGlue’] = pd.Categorical(adata.obs[‘SpatialGlue’],
categories=list_,
ordered=True)
# 绘图
fig, ax_list = plt.subplots(1, 2, figsize=(9.5, 3))
sc.pp.neighbors(adata, use_rep=’SpatialGlue’, n_neighbors=30)
sc.tl.umap(adata)
sc.pl.umap(adata, color=’SpatialGlue’, ax=ax_list[0], title=’SpatialGlue’, s=10, show=False)
sc.pl.embedding(adata, basis=’spatial’, color=’SpatialGlue’, ax=ax_list[1], title=’SpatialGlue’, s=20, show=False)
ax_list[0].get_legend().remove()
plt.tight_layout(w_pad=0.3)
plt.show()
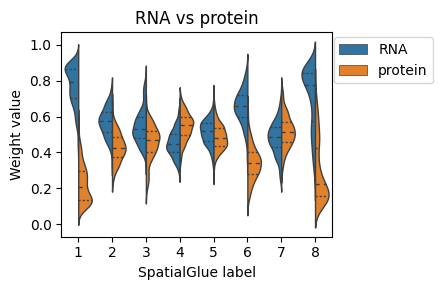
#交换与注释对应的注意权重
list_SpatialGlue = [5,4,8,3,1,6,2,7]
adata.obs[‘SpatialGlue_number’] = pd.Categorical(adata.obs[‘SpatialGlue_number’],
categories=list_SpatialGlue,
ordered=True)
adata.obs[‘SpatialGlue_number’].cat.rename_categories({5:1,
4:2,
8:3,
3:4,
1:5,
6:6,
2:7,
7:8
}, inplace=True)
# 绘制模态权重值
import pandas as pd
import seaborn as sns
plt.rcParams[‘figure.figsize’] = (5,3)
df = pd.DataFrame(columns=[‘RNA’, ‘protein’, ‘label’])
df[‘RNA’], df[‘protein’] = adata.obsm[‘alpha’][:, 0], adata.obsm[‘alpha’][:, 1]
df[‘label’] = adata.obs[‘SpatialGlue_number’].values
df = df.set_index(‘label’).stack().reset_index()
df.columns = [‘label_SpatialGlue’, ‘Modality’, ‘Weight value’]
ax = sns.violinplot(data=df, x=’label_SpatialGlue’, y=’Weight value’, hue=”Modality”,
split=True, inner=”quart”, linewidth=1, show=False)
ax.set_title(‘RNA vs protein’)
ax.set_xlabel(‘SpatialGlue label’)
ax.legend(bbox_to_anchor=(1.4, 1.01), loc=’upper right’)
plt.tight_layout(w_pad=0.05)
#plt.show()
小结
在这里,大海哥给大家介绍了用于空间多组学分析的 SpatialGlue。具体来说,SpatialGlue 是一种空间感知方法,它集成了从同一组织切片获取的多种空间组学数据模态,以更高的空间分辨率破译组织样本的空间域。SpatialGlue 使用图神经网络来学习每种数据模态的低维嵌入,然后进行跨模态的数据集成。为了将空间信息与单个组学数据整合并跨组学整合,我们采用了双注意力聚合机制来自适应地捕捉不同模态的重要性,从而实现更准确的整合。最后大海哥给大家介绍一个云工具!同学们如果觉得自己的代码水平一般,对于很多的参数不知道怎么改,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表,字体大小、标题等也可一键更改。感兴趣的小伙伴去云生信(http://www.biocloudservice.com/home.html)体验一下吧!
