前言
哈喽,朋友们又见面了!今天学习的过程初中被人工智能赋能了吗?小伙伴们都知道深度学习框架最近爆火,像我们熟悉的图神经网络、卷积神经网络等。小伙伴们有没有想过通深度学习框架将空间转录组纳入时空信息会有哪些意想不到的惊喜呢?将伪时间概念与细胞的空间位置相结合,如何来解开细胞的时空格局?带着这样的疑问,跟着大海哥走进今天的主角—— SpaceFlow。 SpaceFlow通过将DGI框架与旨在捕获局部和全局结构模式的空间正则化相结合,提取空间一致的低维嵌入并构建了伪时空图(pSM),它代表了编码细胞之间生物学关系的空间相干伪时间排序,以及区域分割。SpaceFlow 被证明可以产生强大的域分割并识别具有生物学意义的时空模式。
SpaceFlow 是用于从空间转录组学 (ST) 数据中识别时空模式和空间域的 Python 包。SpaceFlow基于深度图网络,提供以下功能:
- 将 ST 数据编码为低维嵌入,以反映 ST 数据中细胞的表达相似性和空间邻近性。
- 通过从嵌入派生的伪时空图 (pSM) 合并 ST 数据中细胞或斑点的时空关系。
- 识别具有空间连贯表达模式的空间域。
有什么生信分析上的问题大家尽管咨询大海哥!没有时间学习的小伙伴们也不要着急哦!有需要生信分析的小伙伴们也可以找大海哥哦!练了十年生信分析的大海哥对于生信分析知识已经如鱼得水从分析到可视化直到你满意为止!
生信数据处理起来占用内存实在太大了,放过自己的电脑吧!大海哥在这里给大家送上福利了,有需要服务器的小伙伴们,欢迎大家联系大海哥,保证服务器的性价比最高哦
代码教程
准备相关环境
要安装 SpaceFlow,我们建议使用 Anaconda Python 发行版并创建一个隔离的环境,以便 SpaceFlow 和依赖项不会与其他包或应用程序发生冲突或干扰。若要创建环境,请在命令行中运行以下脚本:
conda create -n spaceflow_env python=3.7
创建环境后,可以通过以下方式激活环境:spaceflow_env
conda activate spaceflow_env
pip install SpaceFlow
导入 SpaceFlow 和 squidpy 包
import squidpy as sq
import scanpy as sc
from SpaceFlow import SpaceFlow
从 squidpy 包加载 ST 数据
adata = sq.datasets.seqfish()
sc.pp.filter_genes(adata, min_cells=3)
创建 SpaceFlow 对象
我们可以通过任一对象或计数矩阵创建一个 SpaceFlow 对象作为输入:
sf = SpaceFlow.SpaceFlow(adata=adata)
sf = SpaceFlow.SpaceFlow(count_matrix=adata.X, spatial_locs=adata.obsm[‘spatial’], sample_names=adata.obs_names, gene_names=adata.var_names)
参数:
- count_matrix:基因表达的计数矩阵,大小(#个细胞,#个基因)的2Dnumpy数组,类型可选numpy.ndarray
- spatial_locs:细胞(或点)的空间位置与计数矩阵的行匹配,大小为 (n_locations,) 的一维 numpy 数组,类型可选numpy.ndarray
- sample_names:大小为 (n_cells,) 的一维 numpy str 数组中的样本名称列表
- gene_names:大小为 (n_genes,) 的 1D numpy str 数组中的基因名称列表
对ST数据进行预处理
接下来,我们通过运行对 ST 数据进行预处理,预处理包括表达计数矩阵的归一化和对数变换、高变基因的选择以及利用空间坐标构建空间邻近图。这里我们保留前3000个高变基因。
sf.preprocessing_data(n_top_genes=3000)
训练深度图网络模型
然后,我们训练一个空间正则化的深度图网络模型,以学习一个低维嵌入,该嵌入反映了ST数据中细胞的表达相似性和空间邻近性。
sf.train(spatial_regularization_strength=0.1, z_dim=50, lr=1e-3, epochs=1000, max_patience=50, min_stop=100, random_seed=42, gpu=0, regularization_acceleration=True, edge_subset_sz=1000000)
参数:
- spatial_regularization_strength:空间正则化的强度越大,所识别的空间域和时空格局的空间连贯性越大。(默认值:0.1)
- z_dim:学习嵌入的目标大小。(默认值:50)
- lr:优化模型的学习率。(默认值:1E-3)
- epochs:模型训练的最大 epoch 数。(默认值:1000)
- max_patience:等待损失递减的最大纪元数。如果大于此阈值的 epoch 的损失没有减少,则学习将停止,并且具有显示最小损失的参数的模型将保留为最佳模型。(默认值:50)
- min_stop:如果大于 的 epoch 的损失没有减少,则学习可以停止的最早 epoch 。(默认值:100)max_patience
- random_seed:设置为 、 、 包的随机生成器的随机种子。(默认值:42)randomnumpytorch
- gpu:Nvidia GPU 的索引,如果没有 GPU,模型将通过 CPU 进行训练,比 GPU 训练时间慢。(默认值:0)
- regularization_acceleration:是否使用边子集策略加速正则化损失的计算(默认值:True)
- edge_subset_sz:用于正则化加速的边缘子集大小(默认值:1000000)
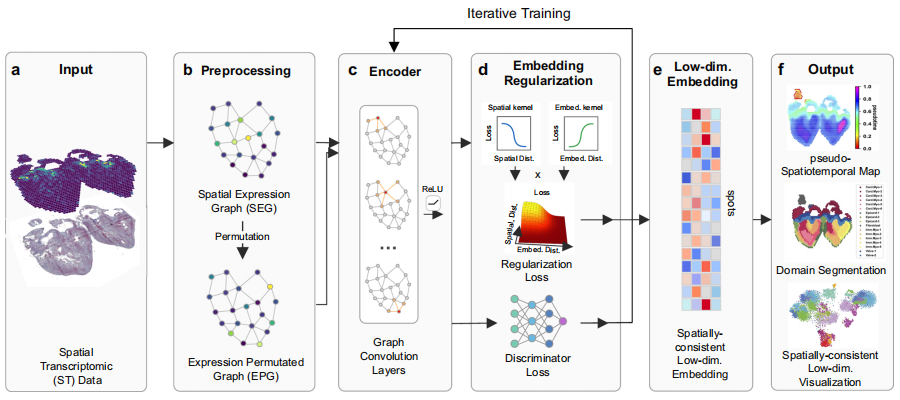
ST数据的领域分割
模型训练完成后,可以通过sf.embedding访问学习到的低维嵌入。SpaceFlow 将使用这种学习嵌入来识别基于 Leiden 算法的空间域。
sf.segmentation(domain_label_save_filepath=”./domains.tsv”, n_neighbors=50, resolution=1.0)
参数:
- domain_label_save_filepath:用于保存已识别域标签的文件路径。(默认值:“./domains.tsv”)
- n_neighbors:使用嵌入作为输入构建莱顿图的每个单元格的最近邻居数。(默认值:50)
- resolution:莱顿聚类的分辨率,域越粗越大。(默认:1.0)
注释和识别空间域的可视化
接下来,我们使用已识别的域标签和细胞的空间坐标绘制空间域。
sf.plot_segmentation(segmentation_figure_save_filepath=”./domain_segmentation.pdf”, colormap=”tab20″, scatter_sz=1., rsz=4., csz=4., wspace=.4, hspace=.5, left=0.125, right=0.9, bottom=0.1, top=0.9)
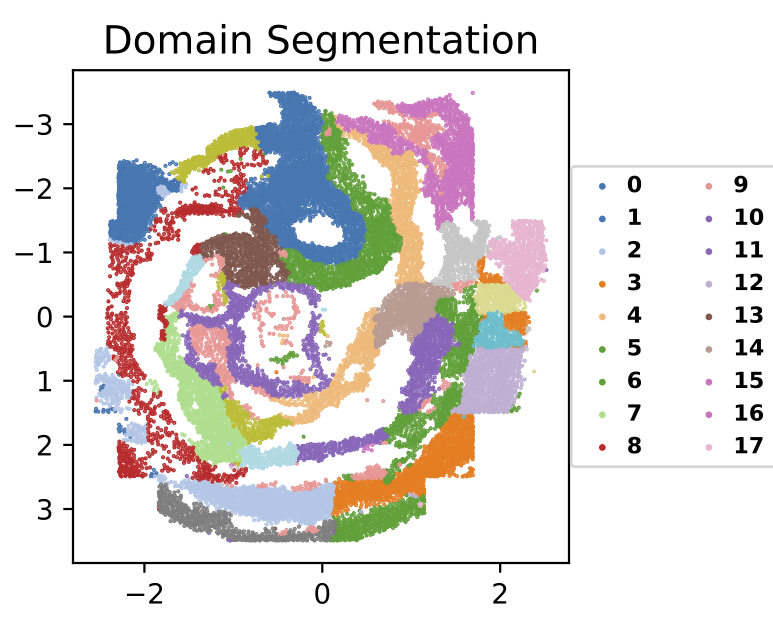
参数:
- segmentation_figure_save_filepath:可选,类型:str,用于保存空间域可视化图的文件路径。(默认值:“./domain_segmentation.pdf”)
- colormap:可选,类型:str,不同域的颜色图,完整的颜色图选项参见 matplotlib
- scatter_sz:可选,类型:float,标记大小(以磅为单位)。(默认:1.0)
- rsz:可选,类型:浮点数,图形的行大小(以英寸为单位),(默认值:4.0)
- csz:可选,类型:float,图形的列大小(以英寸为单位),(默认:4.0)
- wspace: 可选,类型:float,为子图之间的空间保留的宽度量,表示为平均轴宽度的分数(默认值:0.4)
- hspace:可选,类型:float,为子图之间的空间保留的高度量,表示为平均轴高度的分数(默认值:0.4)
- left:可选,类型:float,图的子图的最左边位置(以分数表示)(默认值:0.125)
- right:可选,类型:float,图的子图的最右边位置(以分数表示)(默认值:0.9)
- bottom:可选,类型:float,图的子图的底部位置(以分数表示)(默认值:0.1)
- top:可选,类型:float,以分数表示的图形子图的顶部位置(默认值:0.9)
import scanpy as sc
sc.pl.spatial(adata, color=”celltype_mapped_refined”, spot_size=0.03)

通过伪时空图(pSM)识别ST数据的时空格局
接下来,我们将扩散伪时间(dpt)算法应用于学习到的空间一致性嵌入,以生成伪时空图(pSM)。该 pSM 代表细胞的空间相干伪时间排序,它编码细胞之间的生物学关系,例如发育轨迹和癌症进展。
sf.pseudo_Spatiotemporal_Map(pSM_values_save_filepath=”./pSM_values.tsv”, n_neighbors=20, resolution=1.0)
参数:
- pSM_values_save_filepath:用于保存推断的 pSM 值的文件路径。
- n_neighbors:使用嵌入作为输入构建莱顿图的每个细胞的最近邻居数。(默认值:20)
- resolution:莱顿聚类的分辨率,域越粗越大。(默认:1.0)
识别的伪时空图(pSM)的可视化
sf.plot_pSM(pSM_figure_save_filepath=”./pseudo-Spatiotemporal-Map.pdf”, colormap=”roma”, scatter_sz=1., rsz=4., csz=4., wspace=.4, hspace=.5, left=0.125, right=0.9, bottom=0.1, top=0.9)
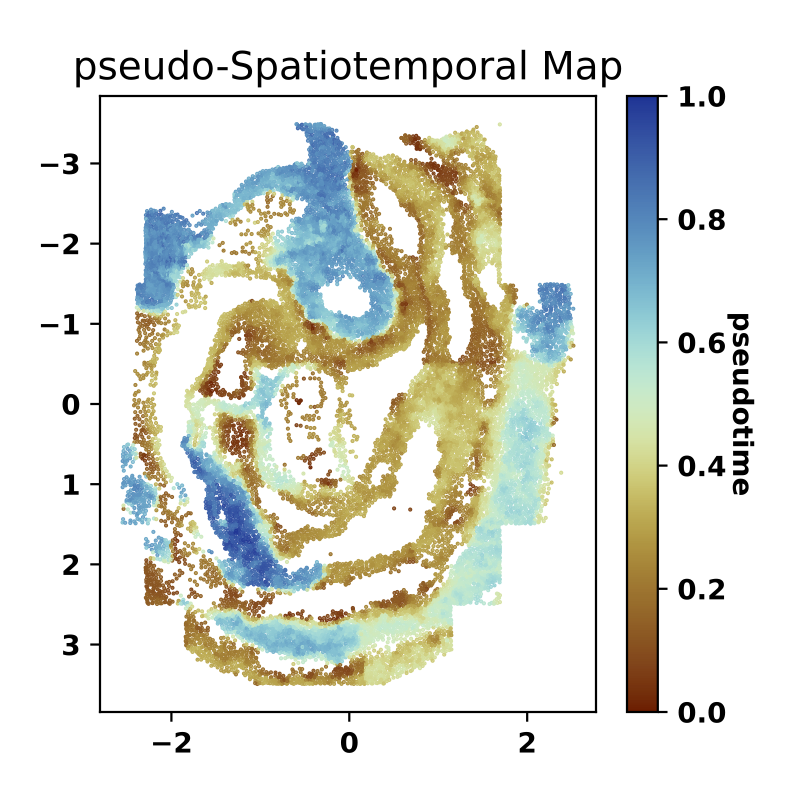
参数:
- pSM_figure_save_filepath:可选,类型:str,用于保存 pSM 可视化图的文件路径。(默认值:“./pseudo-Spatiotemporal-Map.pdf”)
- colormap:可选,类型:str,pSM 的颜色图(默认值:“roma”),完整颜色图选项请参阅科学色图
- scatter_sz:可选,类型:float,标记大小(以磅为单位)。(默认:1.0)
- rsz:可选,类型:浮点数,图形的行大小(以英寸为单位),(默认值:4.0)
- csz:可选,类型:float,图形的列大小(以英寸为单位),(默认:4.0)
- wspace: 可选,类型:float,为子图之间的空间保留的宽度量,表示为平均轴宽度的分数(默认值:0.4)
- hspace:可选,类型:float,为子图之间的空间保留的高度量,表示为平均轴高度的分数(默认值:0.4)
- left:可选,类型:float,图的子图的最左边位置(以分数表示)(默认值:0.125)
- right:可选,类型:float,图的子图的最右边位置(以分数表示)(默认值:0.9)
- bottom:可选,类型:float,图的子图的底部位置(以分数表示)(默认值:0.1)
- top:可选,类型:float,以分数表示的图形子图的顶部位置(默认值:0.9)
小结
SpaceFlow通过使用空间正则化的深度图网络结合表达式相似性和空间信息来生成空间一致的低维嵌入。在嵌入的基础上,我们引入了一种伪时空图,将伪时间概念与细胞的空间位置相结合,以解开细胞的时空格局。SpaceFlow能够产生稳定的区域分割,并识别具有生物学意义的时空模式。综上所述,SpaceFlow提供了一个强大的框架和有效的工具,将先验知识或空间限制纳入ST数据分析,以推断组织中细胞的时空模式。最后大海哥给大家介绍一个云工具!同学们如果觉得自己的代码水平一般,对于很多的参数不知道怎么改,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表,字体大小、标题等也可一键更改。感兴趣的小伙伴去云生信(http://www.biocloudservice.com/home.html)体验一下吧!
