前言
天空一声巨响,大海哥闪亮登场!开始今天的分享之前,小伙伴们竖起耳朵听听大海哥的问题!单细胞基因表达谱的部分突出信息对于我们的分析真的都是有益的吗?有没有小伙伴们思考过这个问题呢?做过单细胞的小伙伴们都知道细胞群体不仅具有空间配置、时间轨迹和细胞间互动等多种生物属性,而且这些属性在数据中交织在一起,形成了复杂的生物信号网络。但是,在实际的研究中,部分突出的信息可能会使我们忽略关键的生物学隐藏属性。比如,在一张典型的 UMAP 降维图中,细胞类型往往会在降维后的主成分中表现的差异最大,即可以在图中将不同的细胞分开,但此时可能会使我们忽略掉一些隐藏的生物学属性。为了更好的使小伙伴们不漏掉重要的信息,所以大海哥要交个大家一个新的python包SiFT。SiFT是一种基于核心的投影方法,专门用于过滤单细胞数据中的生物信号,以揭露潜在的、隐藏在数据背后的生物过程。SiFT的设计目标是广泛且具有适应性,不仅能够从数据中移除不需要的变异,还能揭示隐藏的生物结构。
SiFT算法的概览
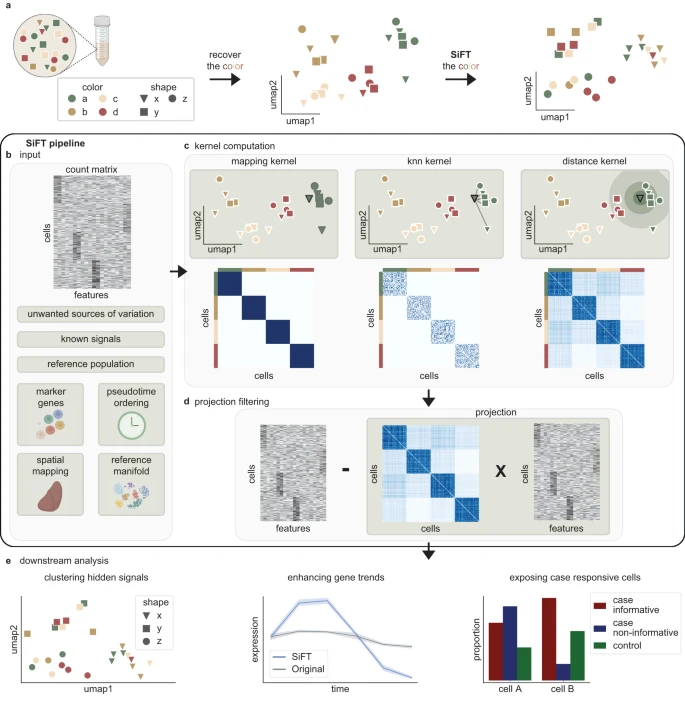
1a的概念性插图说明了SiFT算法应用于每个细胞具有两个属性(形状和颜色)的情况。应用SiFT将暴露形状属性,基于我们对颜色结构的现有知识。图像由BioRender.com创建。1b是输入;SiFT接受一个计数矩阵和预先计算好的细胞映射作为输入。映射可以是连续的或离散的,也可以是单变量的或多维的。图像由BioRender.com创建。1c是细胞-细胞相似性核心计算;根据给定的信号映射计算细胞-细胞相似性核心。列对应不同的核心,映射核心(左),knn核心(中),以及距离核心(右)。顶部行展示了核心捕获的细胞相似性结构,底部行展示了细胞-细胞相似性核心。1d是投影过滤;通过将核心投影到计数矩阵上并从原始计数矩阵中推导出投影来执行过滤。1e是后续分析;过滤后,隐藏结构被暴露并且可以在后续分析中轻松恢复。SiFT允许根据隐藏信号标记(左),增强基因趋势(中),并使用参考对照群体来识别对案例反应的细胞,从而纠正天真的丰度测试,并识别类型B的细胞包含更多疾病状态的信息性细胞群体(右)。
对了,有什么生信分析上的问题大家尽管咨询大海哥!没有时间学习的小伙伴们也不要着急哦!有需要生信分析的小伙伴们也可以找大海哥哦!练了十年生信分析的大海哥对于生信分析知识已经如鱼得水从分析到可视化直到你满意为止!
生信数据处理起来占用内存实在太大了,放过自己的电脑吧!大海哥在这里给大家送上福利了,有需要服务器的小伙伴们,欢迎大家联系大海哥,保证服务器的性价比最高哦!
代码教程
SiFT 的安装
SiFT 基于 python 语言环境,在配置好 Python 环境的前提下,可以在命令行中输入以下代码进行安装:
pip install sift-sc
读入数据并初步可视化
import numpy as np
import scanpy as sc
import sift
读入示例数据,大概700M,示例数据的下载链接如下:
DATA_DIR = “…/drosophila.annotated.h5ad” # set path to anndata
adata = sc.read(DATA_DIR)
adata_ref = adata.copy()
adata
# 此时的adata为AnnData对象
#AnnData object with n_obs × n_vars = 19885 × 3387
# obs: ‘batch’, ‘lncRNA:roX1_cutoff’, ‘lncRNA:roX2_cutoff’, ‘sex’, ‘S_#score’, ‘G2M_score’, ‘phase’, ‘phase_sex’
# uns: ‘batch_colors’, ‘cell_cycle_genes’, ‘log1p’, ‘phase_colors’, ‘ph#ase_sex_colors’, ‘sex_colors’, ‘sex_genes’
# layers: ‘counts’
随后我们执行PCA分析、构建细胞临近图并执行UMAP分析,最后进行可视化
sc.tl.pca(adata_ref)
sc.pp.neighbors(adata_ref)
sc.tl.umap(adata_ref)
##可视化
sc.pl.umap(
adata_ref,
color=[“phase”, “sex”, “phase_sex”, “PCNA”, “lncRNA:roX1”, “Nrt”],
title=[
“cell cycle phase”,
“sex”,
“cc phase & sex”,
“PCNA\n(cc marker)”,
“lncRNA:roX1\n(sex marker)”,
“Nrt\n(biological marker)”,
],
frameon=False,
wspace=0.15,
ncols=3,
)
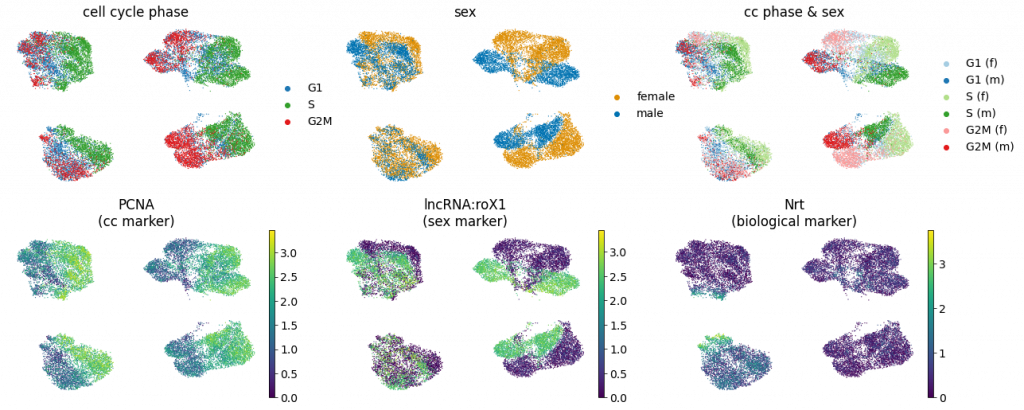
UMAP 嵌入遵循不同的数据校正程序(行)并由不同的协变量着色,代表不需要的变异源和所需的生物信号(列)。行(从上到下)显示未校正的数据(原始数据)、使用 knn 核 (SiFT) 的 SiFT 滤波、Sampy 的 scanpy.pp.regress_out() (regress_out) 和具有连续协变量 (scVI) 的 scVI 潜在空间。列(从左到右)显示了按批次标记、性别标记、细胞周期阶段和 Nrt(参考文献中鉴定的一种新型 Hedgehog 靶基因)着色的细胞。可以看到此时的降维明显是具有细胞周期、性别特异性的。同时,上图还展示了细胞周期、性别相关的一些 marker 的分布,也同样验证了这个结论:
- cell cycle marker: PCNA
- sex marker: lncRNA:roX1
- biological marker: Nrt
执行 SiFT 分析
基于细胞周期、性别相关的干扰基因设置 SiFT 过滤的 adata,随后将干扰基因(性别基因和细胞周期基因)的表达复制到 adata.obsm 中,并从表达矩阵中移除这些基因。
nuisance_genes = np.concatenate((adata.uns[“sex_genes”], adata.uns[“cell_cycle_genes”]))
adata.obsm[“nuisance_genes”] = adata[:, nuisance_genes].X
adata.uns[“nuisance_genes”] = nuisance_genes
gene_subset = [g for g in adata.var_names if g not in nuisance_genes]
adata = adata[:, gene_subset].copy()
设置参数
metric:用于 SiFT 内核的指标。这里我们考虑 k-NN 内核。
n_neighbors:对于 k-NN 内核,我们可以指定使用的近邻数量。
kernel_key:用于定义核的anndata中的键
metric_ = “knn”
n_neighbors_ = 5
kernel_key_ = “nuisance_genes”
##执行SIFT分析
sft = sift.SiFT(
adata=adata,
kernel_key=kernel_key_,
n_neighbors=n_neighbors_,
metric=metric_,
copy=True,
)
我们可以使用 plot_kernel()可视化细胞相似性矩阵:
参数:
- groupby:我们可以对细胞进行分组。
- color_palette:组的颜色。
sft.plot_kernel(
groupby=”phase_sex”, color_palette=adata.uns[“phase_sex_colors”], ncol=3
)
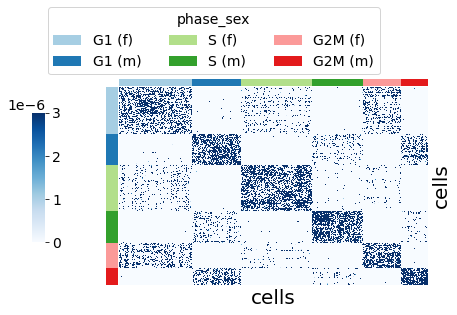
实验数据通常包含不需要的变异源,这些变异源掩盖了感兴趣的生物信号。这些信号可以是离散的(例如,性别标签)或连续的(例如,细胞周期阶段)信号。所需的预处理步骤是去除此类变异源。最佳去除程序应:在保留生物属性的同时有效去除不需要的信号,具有通用性和适应性,并且易于包含在分析管道中。我们应用SiFT来过滤细胞周期和性别相关的变异。
执行 SiFT 的过滤步骤,获得更新的表达矩阵:我们使用filter()函数。
参数:
embedding_key:我们要使用的 for 数据对象。
pseudocount:将 A 添加到过滤的对象中,以确保pseudocount的非负性。
adata = sft.filter(embedding_key=”X”, pseudocount=False)
可视化最终的结果
我们观察对标记基因(细胞周期标记)和(性别标记)的干扰效应的去除。
sc.tl.pca(adata)
sc.pp.neighbors(adata)
sc.tl.umap(adata)
sc.pl.umap(
adata,
color=[“phase”, “sex”, “phase_sex”, “PCNA”, “lncRNA:roX1”, “Nrt”],
title=[
“cell cycle phase”,
“sex”,
“cc phase & sex”,
“PCNA\n(cc marker)”,
“lncRNA:roX1\n(sex marker)”,
“Nrt\n(biological marker)”,
],
frameon=False,
wspace=0.15,
ncols=3,
)
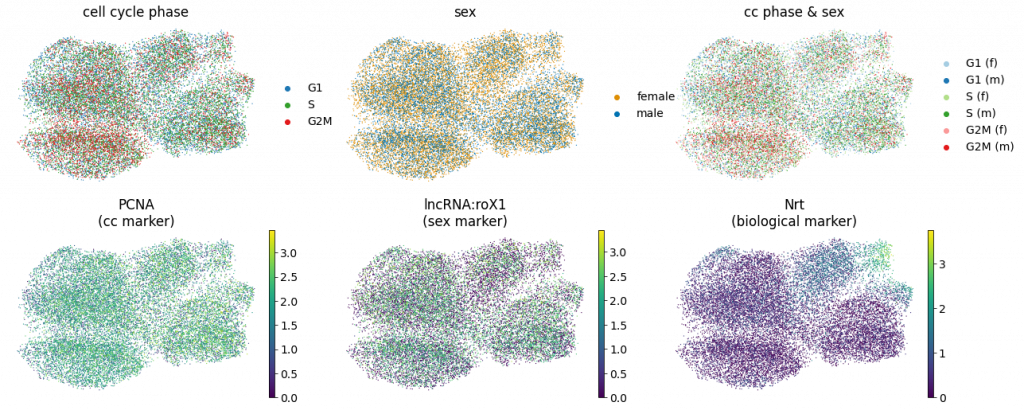
小结
SiFT(Signal FilTering)的研究揭示了一个强大的框架,用于在单细胞数据中有效地过滤和揭露生物过程。通过计算概率细胞-细胞相似性核心,SiFT能够精确地捕捉和过滤出特定的生物信号,从而揭示那些可能被其他信号掩盖的细胞属性。这种方法的优势在于其广泛的适用性和灵活性,不仅能够从数据中移除不需要的变异,还能揭示隐藏的生物结构。SiFT在多个案例中展示了其能力,包括增强肝脏昼夜节律信号、恢复空间分辨肝脏数据中的再生细胞亚群,以及识别与COVID-19疾病相关的细胞、途径和动态。SiFT及类似工具的出现预示着单细胞数据分析领域的新篇章。它们不仅为生物学研究提供了更深层次的洞察,而且为处理和分析日益增长的复杂生物数据集提供了强大的工具。最后大海哥给大家介绍一个云工具!同学们如果觉得自己的代码水平一般,对于很多的参数不知道怎么改,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表,字体大小、标题等也可一键更改。感兴趣的小伙伴去云生信(http://www.biocloudservice.com/home.html)体验一下吧!