
大家好!今天小师妹要向大家介绍一款在生物信息学研究中不可或缺的工具——clusterProfiler包。clusterProfiler是一款功能强大的R包,专门用于基因功能注释和富集分析。通过它,我们可以轻松地对基因列表进行GO和KEGG通路的富集分析,生成详尽的统计结果和丰富的可视化图表,如GO富集网络图。它不仅能帮助我们理解基因在生物学功能上的分布,还能揭示基因调控网络和关键的生物学通路。
生信分析对小师妹来说可不是难题!如果同学们有自己做不了的生信分析,欢迎随时联系我们!
在今天的学习中,小师妹将带领大家一起学习如何安装和加载clusterProfiler包、进行数据预处理、执行进行GO和KEGG富集分析,并利用可视化工具展示分析结果。通过这些学习内容,同学们将能够掌握功能富集分析的基本技能,并且可以将这些方法应用于自己的科研工作中。听起来是不是很有趣呢?那就紧跟小师妹的步伐,让我们一起开启对clusterProfiler包的学习之旅吧!
本次介绍的R包需要较多的硬件资源,在服务器可以更加流畅运行,同学们如果没有自己的服务器欢迎联系我们使用服务器租赁~
clusterProfiler包简介
clusterProfiler包是R语言中用于基因功能注释和富集分析的强大工具,广泛应用于生物信息学领域。它能够对基因列表进行基因本体(GO)和KEGG通路的富集分析,帮助研究者识别显著富集的生物学过程、分子功能和细胞组成。除了基本的富集分析功能,clusterProfiler还支持丰富的可视化工具,能够生成多种类型的图表,如GO条形图、气泡图和富集网络图,使得分析结果更直观易懂。通过简化复杂的基因功能分析流程,clusterProfiler极大提升了数据解读效率。
clusterProfiler包安装
需要R语言版本为4.4,在控制台中输入以下命令:
if (!require(“BiocManager”, quietly = TRUE))
install.packages(“BiocManager”)
BiocManager::install(“clusterProfiler”) # 在BiocManager环境下安装clusterProfiler包
查看是否安装成功
packageVersion(“clusterProfiler”) # 查看clusterProfiler版本

显示为4.12.1版本,则表示已经安装了clusterProfiler包。
除此之外,后续示例还需要使用org.Hs.eg.db包,我们可以提前安装,安装命令如下:
if (!requireNamespace(“BiocManager”, quietly = TRUE))
install.packages(“BiocManager”)
BiocManager::install(“org.Hs.eg.db”)# 在BiocManager环境下安装org.Hs.eg.db包
使用clusterProfiler包进行GO富集分析
载入包和数据
想要进行后续的GO富集分析,我们首先需要载入需要的包和数据,在本文中,我们使用DOSE包提供的genlist数据集作为示例数据,相关代码如下:
library(clusterProfiler) #载入clusterProfiler包
library(org.Hs.eg.db) #载入org.Hs.eg.db包
data(geneList, package=”DOSE”) #从DOSE导入geneList数据
gene <- names(geneList)[abs(geneList) > 2] #筛选出与绝对值大于2的分数相关联的基因
head(gene) # 查看筛选后的Entrez基因ID
结果显示如下图

基因本体(Gene Ontology,GO)分析,主要包括以下三个方面:
生物过程(Biological Process):基因产品参与的生物学事件,如细胞周期、信号传导等。细胞组分(Cellular Component):基因产品所在的细胞部位,如细胞核、膜、细胞质等。分子功能(Molecular Function):基因产品的基本活动,如酶活性、结合活性等。
在本文中我们主要演示使用clusterProfiler包进行细胞组分(Cellular Component)GO富集分析的过程,其余两个方面的GO分析步骤与细胞组分(Cellular Component)大致相同,同学们可以举一反三。
GO分类
首先我们需要使用groupGO()函数,根据特定级别的GO分布对基因进行分类。以下是使用DOSE提供的geneList数据集的示例,代码如下:
# 使用groupGO函数对筛选出的基因进行GO分类
ggo <- groupGO(
gene = gene, # 输入的基因ID向量
OrgDb = org.Hs.eg.db, # 使用的人类基因注释数据库
ont = “CC”, # GO分类本体,”CC”表示细胞组分(Cellular Component)
level = 3, # GO术语层级
readable = TRUE # 将基因ID转换为基因符号
)
head(ggo) #显示前六个GO分类结果

GO富集分析
接着我么可以用clusterProfiler包中的enrichGO()进行GO富集分析,代码如下:
# 执行GO富集分析
ego <- enrichGO(
gene = gene, # 要分析的基因列表
universe= names(geneList), # 背景基因列表,用于比较
OrgDb= org.Hs.eg.db, # 基因注释数据库,这里是人类的基因注释数据库
ont= “CC”, # GO本体类别,这里是细胞组分(Cellular Component)
pAdjustMethod = “BH”, # p值调整方法,这里使用Benjamini-Hochberg方法
pvalueCutoff = 0.01, # p值截断,只有p值小于0.01的结果才会被返回
qvalueCutoff = 0.05, # q值截断,只有q值小于0.05的结果才会被返回
readable = TRUE # 将基因ID转换为可读的基因名称
)
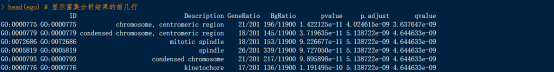
head(ego) # 显示富集分析结果的前几行
结果显示如下图:
我们可以使用goplot()函数可视化enrichGO()函数的GO富集结果,代码如下:
goplot(ego) #可视化富集结果
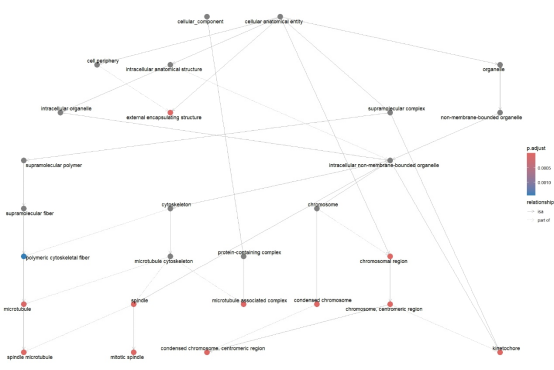
不难看出,图中描述了细胞组件(Cellular Component)类别下的显著GO术语之间的关系网络。图中的节点代表GO术语,节点大小和颜色表示p值的显著性,颜色从红色到蓝色依次表示p值从显著到不显著的渐变。节点之间的线条表示GO术语之间的关系,“isa”表示包含关系,“part of”表示部分关系。图中显示了一些关键的细胞组件,如细胞器(organelle)、染色体(chromosome)、细胞骨架(cytoskeleton)等。通过这些节点和连接线,我们可以看出基因产品在细胞内的位置及其相互关联的结构。例如,“染色体”(chromosome)与“凝缩染色体”(condensed chromosome)之间存在直接关系,而“细胞骨架”(cytoskeleton)与“微管”(microtubule)之间也有直接联系。
载入包和数据
想要进行后续的KEGG富集分析,我们首先需要载入需要的包和数据,在本文中,我们将使用DOSE包提供的genlist数据集作为示例数据,相关代码如下:
library(clusterProfiler) #载入clusterProfiler包
data(geneList, package=”DOSE”) #从DOSE导入geneList数据
gene <- names(geneList)[abs(geneList) > 2] #筛选出与绝对值大于2的分数相关联的基因
gene # 查看筛选后的Entrez基因ID
结果显示如下图

支持KEGG富集分析的生物体
clusterProfiler支持所有在KEGG数据库中具有注释数据的生物体。用户可以直接向organism参数传递kegg_code(生物体名称缩写)。我们所使用的示例数据均为人类基因,所以应该使用缩写“hsa”(Homo sapiens)。
如果你不清楚kegg_code也不用担心,clusterProfiler包提供了search_kegg_organism()函数来帮助搜索支持的生物体。以我们需要搜索大肠杆菌(Escherichia coli)的相关信息为例,相关代码如下:
search_result <- search_kegg_organism(‘ece’, by=’kegg_code’) # 根据 KEGG 代码 ‘ece’ 搜索支持的生物体
search_result # 显示搜索结果
ecoli <- search_kegg_organism(‘Escherichia coli’, by=’scientific_name’) # 根据科学名称 ‘Escherichia coli’ 搜索支持的生物体
dim(ecoli) # 显示搜索结果的维度(行数和列数)
head(ecoli) # 显示搜索结果的前几行

结果如上图,可知kegg_code中包含“ece”的生物体条目为两个,学术名称包含“Escherichia coli”的生物体条目为65个。
KEGG富集分析
KEGG富集通过比较输入的基因列表与KEGG数据库中的通路信息,来识别哪些代谢通路或信号传导通路在基因表达变化中显著富集。通过以上的步骤,我们已经载入了需要的包和示例数据,并且通过search_kegg_organism()函数获得了生物体的kegg_code之后,我们就可以使用enrichKEGG()函数进行KEGG富集分析了,相关代码如下:
kk <- enrichKEGG(gene = gene,
organism = ‘hsa’,
pvalueCutoff = 0.05)
# 使用 enrichKEGG 函数进行 KEGG 富集分析
# gene: 输入的基因列表,通常是显著差异表达基因的 Entrez IDs
# organism: 指定生物体的 KEGG 缩写代码,这里 ‘hsa’ 代表人类 (Homo sapiens)
# pvalueCutoff: 设置 p 值的截断值,用于筛选显著富集的通路,这里设置为 0.05
kk[]# 显示富集分析结果
以上代码运行时间较长,同学们需要耐心等待,结果如下图所示:

可知,KEGG富集结果显示,示例数据中的207个基因富集到了包括细胞周期(Cell cycle)、卵母细胞减数分裂(Oocyte meiosis)、细胞衰老(Cellular senescence)等7个不同的通路。
富集结果可视化
clusterProfiler包有多种方法来可视化富集结果,本文中我们将介绍browseKEGG()函数和pathview::pathview()函数,来对KEGG富集结果进行可视化。
使用browseKEGG()函数的代码如下:
browseKEGG(kk, ‘hsa04110’) #使用browseKEGG()函数可视化“hsa04110”通路
结果如下图:
大家还可以使用pathview包中的pathview()函数来可视化clusterProfiler包中的KEGG富集结果,代码如下:
BiocManager::install(“pathview”) # 在BiocManager环境下安装pathview包
library(“pathview”) # 载入pathview库
hsa04110 <- pathview(
gene.data = geneList, # 输入基因数据,应为一个基因列表或表达数据
pathway.id = “hsa04110”, # 指定KEGG通路ID,这里是细胞周期通路
species = “hsa”, # 指定物种,这里使用人类(hsa)
limit = list(gene=max(abs(geneList)), cpd=1) # 限制显示基因和代谢物的数量
) # 使用pathview函数生成通路可视化
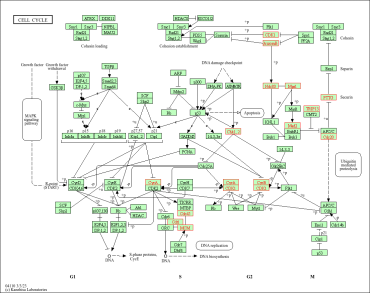
结果显示如下图:

由图可知,KEGG富集结果图详细描述了细胞周期中各个阶段(G1、S、G2、M)涉及的关键基因及其相互作用关系。不同颜色的框表示基因表达的变化情况,红色表示基因上调,绿色表示基因下调,灰色表示基因未显著变化,颜色渐变表示变化的程度。图中还展示了多个与细胞周期调控相关的信号通路和蛋白质复合物,如MAPK信号通路、SCF复合物、APC/C复合物等。通过这些通路和复合物,可以看到基因在细胞周期中的精细调控过程,以及细胞如何通过一系列信号传导和基因表达变化来完成细胞分裂。
以上就是对clusterProfiler包的全面介绍了。通过学习如何安装和使用clusterProfiler,我们掌握了在生物信息学中进行基因本体(GO)富集分析和KEGG通路富集分析的关键步骤和方法。clusterProfiler能够帮助我们识别基因在生物学过程、细胞组分、分子功能中的显著富集,同时揭示与代谢和信号传导相关的重要通路。借助其丰富的可视化工具,如goplot()和pathview()函数,我们能够更直观地解读复杂的富集结果。希望大家继续深入探索生物信息学领域,充分利用clusterProfiler包,为科研贡献力量!
同学们如果觉得自己写代码麻烦,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表。立即访问云生信(http://www.biocloudservice.com/home.html),开启便捷的生信之旅!
