
大家好!今天小师妹要向大家介绍一个在结构生物信息学领域中非常重要的R包——Bio3D包!Bio3D包是专门用于分析生物大分子三维结构的强大工具。随着结构生物学的发展,越来越多的生物大分子结构数据被解密和应用,Bio3D包能够帮助我们深入分析这些结构数据,并通过多种算法揭示生物分子之间的动态关系。它可以处理蛋白质的三维结构,执行包括结构比对、分子动力学轨迹分析、正规模式分析(NMA)等任务,对研究蛋白质的结构功能关系有重要帮助。小师妹可是厉害的生信高手,要是同学们有自己做不了的蛋白质结构分析,欢迎随时联系我!!
在本次学习中,我们不仅将学会如何安装和使用Bio3D包,还将掌握如何提取蛋白质的三维结构数据、执行正规模式分析、并结合二级结构信息展示柔性特征的可视化方法。通过实际示例的演示,我们将深入了解Bio3D包的功能,为研究生物分子结构与功能的关系提供宝贵工具。我相信,通过今天的学习,同学们都能够更加熟练地应用Bio3D包,为结构生物信息学的研究进步贡献自己的一份力量!接下来,同学们跟紧小师妹的步伐,让我们正式开启对Bio3D包的学习之旅吧!
本次介绍的R包在服务器上能更加流畅地运行,如果没有服务器,欢迎联系我们进行服务器租赁~
Bio3D包介绍
Bio3D是一个用于结构生物信息学分析的R语言包,主要用于生物大分子(如蛋白质和核酸)三维结构数据的处理和分析。它能够执行各种结构分析任务,如结构对接、同源建模、分子动力学模拟轨迹分析、结构比对等。Bio3D的核心功能包括从PDB数据库中提取结构数据、计算结构相似性、生成结构比对和聚类分析,以及分析蛋白质动力学特征,如主成分分析(PCA)和正规模式分析(NMA)。该包广泛应用于蛋白质功能研究、分子演化研究以及药物设计等领域,帮助研究者深入理解生物分子结构与功能的关系。
Bio3D包安装
需要R语言版本为4.4,在控制台中输入以下命令:
install.packages(“bio3d”, dependencies=TRUE)
查看是否安装成功:
packageVersion(“Bio3D”) # 查看Bio3D版本

显示为2.4.4版本,则表示已经安装了Bio3D包。
通过Bio3D包的正规模式分析(NMA)来分析蛋白质的三维结构
载入需要的包和数据
首先,我们需要载入本次学习的主角——Bio3D包。
library(bio3d) # 载入bio3d包
在本文中,小师妹将使用pdb数据库中编号为 “1HEL” 的溶菌酶(Lysozyme)作为示例蛋白,通过使用bio3d包的正规模式分析(NMA)来分析其柔性及二维结构特征。
我们可以使用bio3d包中的read.pdb()函数从pdb数据库中下载溶菌酶的PDB文件,代码如下:
pdb <- read.pdb(“1hel”) # 下载溶菌酶的PDB文件
print(pdb) # 显示示例蛋白基础信息

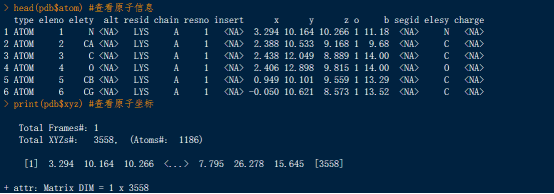
可知溶菌酶是由129个氨基酸残基,1186个原子组成的单链蛋白质。
如果想要详细查看原子构成以及坐标信息,可以使用以下命令:
head(pdb$atom) #查看原子信息
print(pdb$xyz) #查看原子坐标
显示如下图:
进行蛋白质三维结构分析
在本文中,我们将使用bio3d包中的nma()函数,进行正规模式分析(NMA),从而预测示例蛋白质的柔性。代码如下:
modes <- nma(pdb) # 使用bio3d包进行进行蛋白质三维结构分析。
plot(modes) # 使用plot()函数展示分析结果。
显示结果如下图:

由图可知,上方两个图分别显示了特征值(Eigenvalues)和频率(Frequencies)随模式指数(Mode Index)的变化。特征值从左至右递增,说明高模式对应的刚性较大,运动较难发生。频率图与特征值密切相关,也表现出递增趋势,高频模式对应局部振动,低频模式则与全局运动相关。下方的波动图(Fluctuations)展示了蛋白质各残基的振幅波动情况。某些残基区域(如第50、70和120附近)显示出较大的波动,意味着这些区域更具柔性,可能在蛋白质功能中起重要作用。
为了能够清晰的展示蛋白质的柔性和其二级结构之间的关系,我们可以使用plot.bio3d()函数中的参数在波动图(Fluctuations)上添加二级结构信息,代码如下:
plot.bio3d(
modes$fluctuations, # 正常模式分析(NMA)中每个残基的波动数据
sse=pdb, # 提供PDB对象以获取蛋白质的二级结构信息
sheet.col=”orange”, # 将β-折叠的颜色设置为橙色,用于在图中区分二级结构
helix.col=”purple”, # 将α-螺旋的颜色设置为紫色,用于区分二级结构
typ=”l”, # 设置图形类型为线条图(line plot),即用线连接数据点
lwd=3, # 将线条宽度设为3,使线条更加粗壮,图形更清晰
ylab=”Fluctuations from NMA (custom label)” # 设置y轴标签为自定义内容
)
结果显示如下图:

由图可知,x轴表示残基编号,y轴表示波动幅度。图中的黑色曲线显示了各个残基的波动情况,波峰代表特定残基有较大的波动,表明这些区域在蛋白质运动中柔性更高。紫色的横条代表α螺旋区域,橙色横条代表β折叠区域,这些区域的波动通常较小,表明它们结构较为稳定,柔性相对较低。图像分析有助于研究蛋白质的动态特性,分析哪些区域更具柔性或稳定性。
以上就是对于Bio3D包的全部介绍了。通过本文,我们了解了Bio3D包作为蛋白质三维结构分析的强大工具及其应用场景。Bio3D包能够通过Normal Mode Analysis(NMA)预测蛋白质的柔性,揭示其结构动态特性,并通过与二级结构的结合更直观地展示蛋白质的关键区域。希望同学们能够继续深入学习和探索Bio3D包,通过不断的实践和应用,大家一定能在蛋白质结构分析的研究中取得更大的进步和成果。
同学们如果觉得自己写代码麻烦,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表。立即访问云生信(http://www.biocloudservice.com/home.html),开启便捷的生信之旅!
