前言
基于特定marker的单细胞亚群注释是不是已经手到擒来、轻松搞定了?是不是想尝试一下新的单细胞注释方法了?基于表型信息预测单细胞数据中的相关亚群的方法你听说过吗?目前,在大容量数据中整合来自具有广泛临床特征的小样本的单细胞数据仍未得到充分探索。因此,PIPET横空出世!PIPET从批量数据中差异表达的基因生成每种表型的特征载体,然后通过评估单细胞数据与这些载体之间的相似性来识别相关的细胞亚群。随后,可以根据这些亚群分析与表型相关的细胞状态。PIPET从批量数据中差异表达的基因生成每种表型的特征载体,然后通过评估单细胞数据与这些载体之间的相似性来识别相关的细胞亚群。随后,可以根据这些亚群分析与表型相关的细胞状态。单细胞数据带有表型、临床相关信息的小伙伴们开始学起来吧!大海哥就是这么的宠粉,有什么生信分析上的问题大家尽管咨询大海哥!没有时间学习的小伙伴们也不要着急哦!有需要生信分析的小伙伴们也可以找大海哥哦!练了十年生信分析的大海哥对于生信分析知识已经如鱼得水从分析到可视化直到你满意为止!

生信数据处理起来占用内存实在太大了,放过自己的电脑吧!大海哥在这里给大家送上福利了,有需要服务器的小伙伴们,欢迎大家联系大海哥,保证服务器的性价比最高哦!
代码教程
要安装此包,请启动 R(版本 >= 4.3.1)并输入:
if (!require(“devtools”))
install.packages(“devtools”)
devtools::install_github(“ruan2ruan/PIPET”)
如果在安装过程中出现错误,我们建议您先尝试 通过引用 DESCRIPTION 文件中的Imports来安装所有 R 依赖项,然后安装 PIPET。
##加载R包
library(PIPET)
在本教程中,我们的单细胞数据集来自于外周血单个核细胞(PBMC),可从10X基因组免费获得。经过预处理和单元格类型标注的数据可以从SeuratData下载。因此,在加载Seurat对象数据之前,我们还需要加载一些相关的包:
library(tidyverse)
library(patchwork)
library(Seurat)
library(SeuratData)
本文展示了单细胞数据PIPET预测的关键步骤,并提供了一些下游可视化结果。PIPET的输入需要一个单细胞表达矩阵和一组包含表型信息的特征向量。该软件包包含用于AML分类的571个基因面板,以及TCGA-LAML的TPM表达数据和生存数据的预处理,现在我们将展示如何在实际应用中执行PIPET。
#InstallData(“pbmc3k”)
pbmc3k.final <- LoadData(“pbmc3k”, type = “pbmc3k.final”)
SC = UpdateSeuratObject(object = pbmc3k.final)
SC
然后,我们加载了AML分类的特征数据(571个基因识别了三个主要簇c1, c2, c3)。生成了这些特征基因,并证明它们在AML临床相关亚型的分类中具有良好的性能。
markers <- read.table(“../data/AML_Subtypes_signature.txt”,sep = “\t”,check.names = F,stringsAsFactors = F,header = T,row.names = NULL)
table(markers$class)
head(markers,5)
使用PIPET识别细胞亚群
基于上述输入数据,我们可以使用PIPET识别表型相关的细胞亚群。计算可能需要几分钟,这取决于你的数据量和你设置的核心数量。
set.seed(123)
tmp <- PIPET(SC_data=SC, markers=markers, gene_col= “genes”, class_col= “class”,
nCores=4)
我们将PIPET预测的细胞亚群结果与原始单细胞数据合并。
tmp$ID <- rownames(tmp)
metadata <- SC@meta.data
metadata$ID <- rownames(metadata)
meta <- merge(metadata,tmp,by=”ID”)
rownames(meta) <- meta$ID
SC <- AddMetaData(SC, metadata = meta)
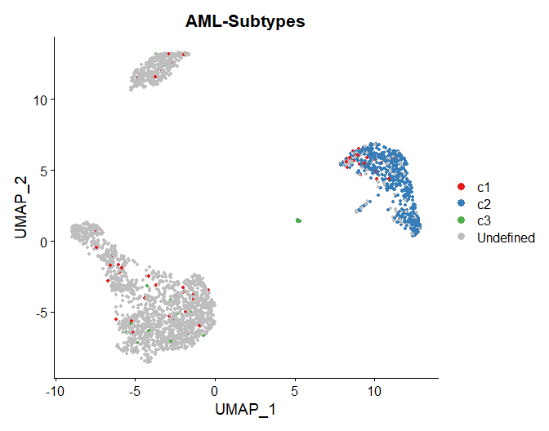
滤除显著相关的细胞亚群(P值小于0.05)后,PIPET鉴定出与AML-c1亚型相关的PIPET_c1细胞66个,与AML-c2亚型相关的PIPET_c2细胞517个,与AML-c3亚型相关的PIPET_c3细胞34个。
SC1 <- subset(x = SC, subset= (Pvalue < 0.05))
table(SC1$prediction)
使用UMAP可视化所有细胞
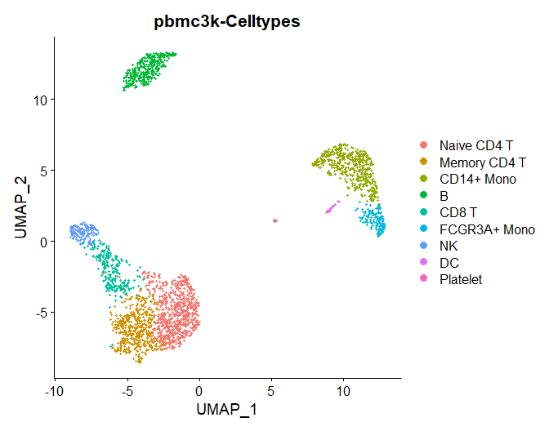
我们首先使用UMAP图可视化了分析中包含的所有细胞。不同的细胞类型通过颜色来区分。
使用UMAP可视化预测的细胞,然后我们只绘制带PIPET注释的细胞。
metadata <- SC@meta.data
temp <- data.frame(ID=SC1$ID,type=SC1$prediction)
meta <- merge(metadata,temp,by=”ID”,all = TRUE)
meta$type <- as.character(meta$type)
meta$type <- ifelse(is.na(meta$type), “Undefined”, meta$type)
rownames(meta) <- meta$ID
meta <- meta[SC$ID,]
SC <- AddMetaData(SC, metadata = meta)
library(RColorBrewer)
colors<-brewer.pal(n = 8, name = “Set1”)
PIPET_DotPlot(SC,colors,group=”type”,title=”AML-Subtypes”)
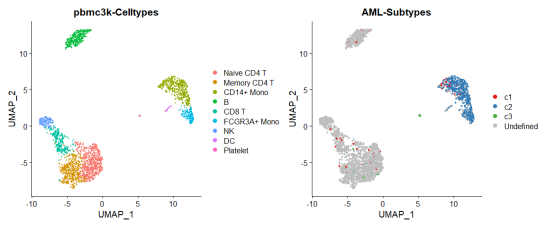
为了更直观的比较,我们同时绘制两个结果。
p1 <- DimPlot(SC, reduction = “umap”, group.by = ‘seurat_annotations’, label = F) +
ggtitle(“pbmc3k-Celltypes”)
p2 <- PIPET_DotPlot(SC,colors,group=”type”,title=”AML-Subtypes”)
p1 | p2
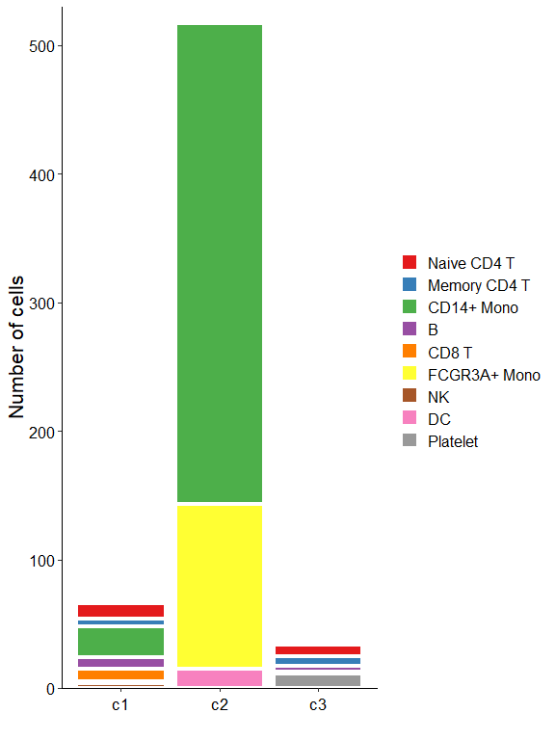
我们可以看到,每个PIPET亚群中的细胞来自不同的细胞类型。PIPET_BarPlot()可以通过堆叠的直方图显示每个细胞子群的主要细胞组成。
table(SC1$seurat_annotations,SC1$prediction)
PIPET_BarPlot(SC1,group=”seurat_annotations”,prediction=”prediction”,ylim=530)
然后我们发现了PIPET_c1, PIPET_c2和PIPET_c3的差异表达特征,并选择顶级标记基因来可视化。
DefaultAssay(SC1) <- “RNA”
Idents(SC1)=”prediction”
SC.markers <- FindAllMarkers(SC1, only.pos = TRUE,
min.pct = 0.25, logfc.threshold = 0.25)
biomarkers <- SC.markers %>%
dplyr::filter(p_val_adj<0.05) %>%
group_by(cluster) %>%
slice_max(n = 20000, order_by = avg_log2FC)
top <- biomarkers %>%
group_by(cluster) %>%
slice_head(n = 4)
top
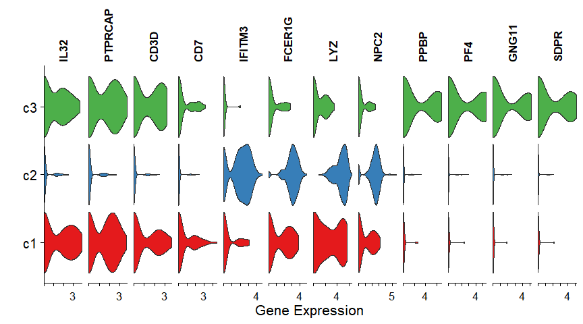
我们使用PIPET_MultiViolin()创建了一个堆叠小提琴图,它可以在同一幅图中显示多个细胞群体或细胞类型的小提琴图,以比较它们之间的基因表达变化。
features <- top$gene
PIPET_MultiViolin(SC1, features, preprocess=FALSE)
加载预处理后的TCGA-LAML体表达矩阵和相应的临床生存数据。TCGA-LAML的FPKM数据和相应的临床数据从Xena公共平台(https://xena.ucsc.edu/public-hubs)下载。我们将FPKM表达数据转换为TPM,并将样本与表达数据和临床数据进行匹配。
data(“TCGA_LAML_TPM”)
TPM <- TCGA_LAML_TPM
data(“TCGA_LAML_Surv”)
surv.info <- TCGA_LAML_Surv
在这个TPM表达矩阵中,每行代表一个基因,每列代表一个样本。后续分析共纳入49038个基因和130个样本。
dim(TPM)
我们只选取生存信息的临床数据,包括样本ID、结局事件(1表示死亡,0表示存活)和生存时间(以天为单位)。
head(surv.info,3)
选取PIPET_c3中有显著差异表达的基因(c3亚型为预后最差的亚型),建立多变量Cox回归模型。
c3 <- biomarkers %>%
filter(cluster==”c3″) %>%
filter(avg_log2FC>=2)
TPM.norm <- log2(TPM + 1)
TPM.norm <- data.frame(t(na.omit(TPM.norm[c3$gene,])))
genes <- colnames(TPM.norm)
tmp <-TPM.norm[rownames(surv.info),]
survival_info_df <- cbind(tmp,surv.info)
library(survival)
multi_COX<-coxph(Surv(futime, fustat) ~ ., data=survival_info_df)
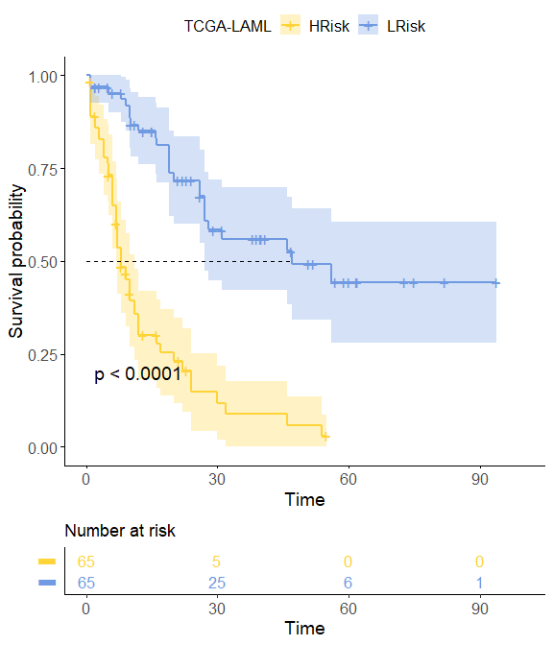
PIPET_KM()根据多变量Cox回归模型得到的系数计算每个样本的风险评分。然后根据风险评分中位数将样本分为高/低风险组。最后,根据风险群体绘制KM生存曲线。如下图所示,高危组患者预后差于低危组患者。
PIPET_KM(data=survival_info_df, genes=genes,coxph=multi_COX, surv_cols=c(“fustat”,”futime”),
xlim=100,legend.title=”TCGA-LAML”, legend.labs = c(“HRisk”, “LRisk”),
pval = TRUE, conf.int = TRUE, palette = “simpsons”)
小结
以上,我们介绍了PIPET包包含的主要功能。它可以帮助您整合来自批量数据的信息,并注释单细胞数据,并进行多组学分析。PIPET包不仅可以用于预测单个细胞亚群中的相关细胞亚群,而且还提供了几种下游分析的可视化功能。最后了大海哥还是要给大家小送一波福利!大海哥给大家介绍一个云工具!同学们如果觉得自己的代码水平一般,对于很多的参数不知道怎么改,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表,字体大小、标题等也可一键更改。感兴趣的小伙伴去云生信(http://www.biocloudservice.com/home.html)体验一下吧!