前言
沉浸在暑期快乐时光里的小伙伴们快收收心吧!千万别忘记大海哥的每日小课堂啊!厌倦了游戏、短视频的小伙伴抓紧时间来看看大海哥又给大家带来哪些新的技能了!单细胞中的细胞通讯都听说过吧,空间转录组中的细胞通讯分析你get到了吗?今天跟着大海哥的步伐来看看是怎么个是!小伙伴们都知道解码细胞间通讯对于理解许多生物过程的机制很重要,但由于多种因素的混合和缺乏直接观察,所一清华大学生物信息学教育部重点实验室雷伟团队开发了一种使用空间转录组数据解码细胞间通讯的方法HoloNet。它通过将配体-受体对、细胞类型空间分布和下游基因表达集成到深度学习模型中来解码细胞间通讯。小伙伴们快来学习吧!生信分析不熟悉的小伙伴们欢迎来滴滴大海哥,大海哥就是这么的宠粉,有什么生信分析上的问题大家尽管咨询大海哥!没有时间学习的小伙伴们也不要着急哦!有需要生信分析的小伙伴们也可以找大海哥哦!练了十年生信分析的大海哥对于生信分析知识已经如鱼得水从分析到可视化直到你满意为止!
生信数据处理起来占用内存实在太大了,放过自己的电脑吧!大海哥在这里给大家送上福利了,有需要服务器的小伙伴们,欢迎大家联系大海哥,保证服务器的性价比最高哦!
代码教程
在本教程中,我们将演示如何使用 HoloNet 来分析和可视化细胞间通信在空间转录组学数据中的应用。
本教程主要遵循以下步骤:
- 加载空间转录组数据。
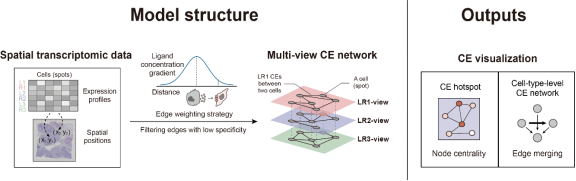
- 在单细胞之间构建多视图通信事件(CE)网络(每个LR对应一个视图)。
- 基于多视图网络的通信可视化。
- 其他分析方法(例如聚类 LR 对)。
#安装
pip install HoloNet
HoloNet 需要三个输入:
- 空间转录组数据(包括基因表达矩阵和空间信息)。
在此版本的 HoloNet 中,我们支持基于 Scanpy 加载的空间转录组数据。
- 细胞类型信息。
来自反卷积方法的细胞类型百分比更倾向于保存在adata.obsm[‘predicted_cell_type’]中。
分类细胞类型标签更愿意保存在adata.obs[‘cell_type’]中。
- 具有成对配体和受体基因的数据库。
pandas 数据必须包含两列:“Ligand_gene_symbol”和“Receptor_gene_symbol”。
#加载所有python库
import HoloNet as hn
import os
import pandas as pd
import numpy as np
import scanpy as sc
import matplotlib.pyplot as plt
import torch
import warnings
warnings.filterwarnings(‘ignore’)
hn.set_figure_params(tex_fonts=False)
sc.settings.figdir = ‘./figures/’
加载示例空间转录组数据
我们为 10x Genomics 网站的用户准备了一个乳腺癌数据集示例。 我们对数据集进行了预处理,包括过滤、归一化和细胞类型标注。 用户可以通过hn.pp.load_brca_visium_10x()加载示例数据集。
adata = hn.pp.load_brca_visium_10x()
本教程中绘图函数的参数主要继承自两个基础绘图函数:
- HoloNet.plotting.feature_plot()
- HoloNet.plotting.cell_type_level_network()
可视化每个点的细胞类型百分比
hn.pl.plot_cell_type_proportion(adata, plot_cell_type=’stroma’)

##细胞类型注释
sc.pl.spatial(adata, color=[‘cell_type’], size=1.4, alpha=0.7,
palette=hn.brca_default_color_celltype)

加载配体受体数据库
我们为用户提供了一个包含成对配体和受体基因的数据库。 加载数据库并过滤 LR 对,需要表达配体和受体基因在一定比例的细胞中。
interaction_db, cofactor_db, complex_db = hn.pp.load_lr_df(human_or_mouse=’human’)
expressed_lr_df = hn.pp.get_expressed_lr_df(interaction_db, complex_db, adata)
expressed_lr_df.shape
我们还允许访问基于 MITAB 规范化命名法的配体-受体对数据库。 我们在这里提供样本,以便用户可以导入符合MITAB的配体受体数据库并替换。
pd.set_option(‘display.max_columns’, None)
interaction_db_MITAB = pd.read_csv(‘cellchatdb_human_interaction_example_with_MITAB.csv’, index_col=0)
interaction_db_MITAB.head()
expressed_lr_df = hn.pp.get_expressed_lr_df(interaction_db_MITAB, complex_db, adata)
构建多视图CE网络
w_best = hn.tl.default_w_visium(adata)
hn.pl.select_w(adata, w_best=w_best)

在此基础上,我们可以构建多视角通信网络。我们计算了多视图通信网络的边缘权重。 随着配体的归属增加,可以为分泌的配体和质膜结合配体设置不同的配体。然后,我们过滤具有低特异性的通信网络的边缘权重。
elements_expr_df_dict = hn.tl.elements_expr_df_calculate(expressed_lr_df, complex_db, cofactor_db, adata)
ce_tensor = hn.tl.compute_ce_tensor(expressed_lr_df, w_best, elements_expr_df_dict, adata)
filtered_ce_tensor = hn.tl.filter_ce_tensor(ce_tensor, adata, expressed_lr_df, elements_expr_df_dict, w_best)
注意:以下骤将消耗大量内存。 如果内存不足,可以选择仅计算配体-受体对较少的通信网络,并调低参数的值,此外,您可以设置或下采样数据集以减少内存消耗。
import random
cut_porp = 2
adata_subset = adata[random.sample(list(adata.obs_names), round(adata.shape[0] / cut_porp))]
elements_expr_df_dict = hn.tl.elements_expr_df_calculate(expressed_lr_df, complex_db, cofactor_db, adata_subset)
ce_tensor = hn.tl.compute_ce_tensor(expressed_lr_df, w_best, elements_expr_df_dict, adata_subset)
filtered_ce_tensor = hn.tl.filter_ce_tensor(ce_tensor, adata_subset, expressed_lr_df,
elements_expr_df_dict, w_best, copy=False)
可视化 CE
基于多视图CE网络,我们提供两种可视化方法:
- 用于可视化细胞中心的CE热点图。提供两种计算方法:
- Degree centrality: out-degree + in-degree,忠实地反映了与每个点相关的CE强度。
- 特征向量中心性:反映通信活跃的核心区域。
- 细胞类型级别的CE网络:
细胞类型间的CE强度
CE 热点图
TGFB1:(TGFBR1+TGFBR2) CE网络中各点的度中心性。TGFB1:(TGFBR1+TGFBR2)通信活跃的反射区域。
hn.pl.ce_hotspot_plot(filtered_ce_tensor, adata,
lr_df=expressed_lr_df, plot_lr=’TGFB1:(TGFBR1+TGFBR2)’)

基于特征向量中心度的热点图。该图比基于度中心性的图更好地检测出了一个清晰的中心。
hn.pl.ce_hotspot_plot(filtered_ce_tensor, adata,
lr_df=expressed_lr_df, plot_lr=’TGFB1:(TGFBR1+TGFBR2)’,
centrality_measure=’eigenvector’)

细胞类型间的CE强度
##加载每个点的细胞类型百分比
cell_type_mat, \
cell_type_names = hn.pr.get_continuous_cell_type_tensor(adata, continuous_cell_type_slot = ‘predicted_cell_type’,)
绘制细胞类型间的CE 网络。 边缘的厚度代表了两种细胞类型之间通讯的强度:TGFB1:(TGFBR1+TGFBR2)。
_ = hn.pl.ce_cell_type_network_plot(filtered_ce_tensor, cell_type_mat, cell_type_names,
lr_df=expressed_lr_df, plot_lr=’TGFB1:(TGFBR1+TGFBR2)’, edge_thres=0.2,
palette=hn.brca_default_color_celltype)

LR 对聚类: 根据每个点的中心性对配体-受体对进行聚类。
cell_cci_centrality = hn.tl.compute_ce_network_eigenvector_centrality(filtered_ce_tensor)
clustered_expressed_LR_df,_ = hn.tl.cluster_lr_based_on_ce(filtered_ce_tensor, adata, expressed_lr_df,
w_best=w_best, cell_cci_centrality=cell_cci_centrality)
用于分层聚类所有配体-受体对的树状图。
hn.pl.lr_clustering_dendrogram(_, expressed_lr_df, [‘TGFB1:(TGFBR1+TGFBR2)’],
dflt_col = ‘#333333’,)

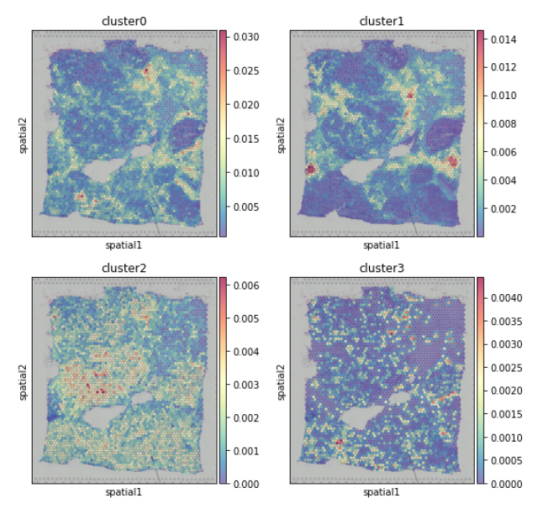
每个配体-受体簇的CE热图(将所有细胞的CE热点叠加在一个簇中)。
hn.pl.lr_cluster_ce_hotspot_plot(lr_df=clustered_expressed_LR_df,
cell_cci_centrality=cell_cci_centrality,
adata=adata)
小结
HoloNet可以帮助用户解决许多生物学问题。例如,与本文中介绍的分析类似,用户可以使用 HoloNet 来分析微环境中其他细胞类型的细胞表达谱如何影响。通过提供治疗前和治疗后的空间数据,用户还可以分析治疗如何影响组织内的完整通信景观,了解意想不到的治疗结果,并开发新的疾病干预措施。HoloNet可以应用于各种空间转录组数据。基于单细胞的空间技术,如SeqFISH和MERFISH,也可以很容易地被用作输入。最后大海哥给大家介绍一个云工具!同学们如果觉得自己的代码水平一般,对于很多的参数不知道怎么改,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表,字体大小、标题等也可一键更改。感兴趣的小伙伴去云生信(http://www.biocloudservice.com/home.html)体验一下吧!