同学们,今天大海哥要向大家介绍一款在生物信息学和结构生物学研究中非常重要的工具——PyMOL!只要我们需要理解蛋白质和核酸的三维结构,揭示它们在细胞内的功能和相互作用,就离不开PyMOL的帮助。作为一款强大的分子结构可视化软件,PyMOL能够以高分辨率展示复杂的生物大分子结构,帮助我们更好地理解这些分子在生物过程中的关键作用。通过PyMOL,我们可以轻松地处理和分析这些结构数据,为我们的科研工作提供坚实的支持。大海哥可是拥有丰富经验的生信高手哦,如果同学们有任何生信分析方面的问题,欢迎随时联系我!
在本次学习中,大海哥将细致地教大家如何安装和使用PyMOL,并通过实际示例的演示,让同学们更深入地了解如何进行分子结构的可视化分析。因此,本次学习对大家来说十分重要,希望同学们认真听讲并积极动手实践。大海哥相信,通过这次学习,同学们不仅能掌握PyMOL的使用技巧,还能更深入地理解生物大分子的结构和功能,为今后的科研工作打下坚实的基础。
本次介绍的工具需要在服务器上才能正常运行,同学们如果没有自己的服务器欢迎联系我们进行服务器租赁~
PyMol的介绍
PyMOL 是一款广泛应用于分子结构可视化的开源软件,特别适用于生物化学和生物物理学研究。它能够以高分辨率显示蛋白质、核酸等生物大分子的三维结构,支持多种文件格式如PDB和CIF。用户可以通过直观的图形界面或灵活的命令行工具进行分子建模和分析,包括旋转、平移和缩放结构,以及显示氢键等生物分子间的相互作用。PyMOL 提供强大的渲染功能,生成高质量的静态图像和动态动画,适用于科研、教学和学术演示。
PyMol安装
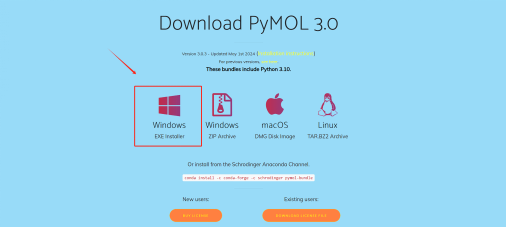
首先,打开你的浏览器,访问PyMOL的官方网站(https://PyMol.org/2/)。在主页上,你会看到“Download”按钮,点击它进入下载页面。在这里,你可以选择适合你操作系统的版本进行下载。PyMOL支持Windows、MacOS和Linux系统,所以不管你用的是哪种电脑,都可以找到对应的安装包。
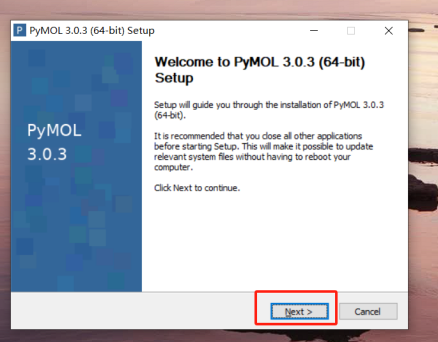
下载完成后,双击安装包开始安装。安装过程很简单,只需要按照提示点击“Next”就可以了。需要注意的是,如果你使用的是Windows系统,在安装过程中可能会提示你安装一些必要的依赖组件,按照提示安装即可。
成功安装之后,PyMol的界面如图所示:
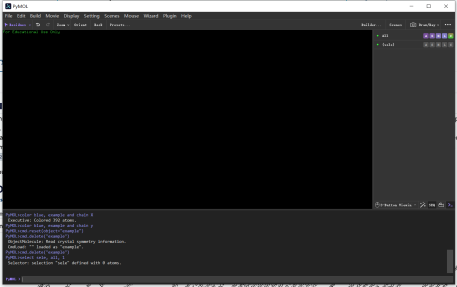
使用PyMol进行分子结构可视化分析
文件载入
在本文中,大海哥将使用转录因子和DNA结合的复合物的三维结构向大家演示如何使用PyMol进行分子结构可视化分析。大海哥已经准备好了转录因子(General transcription factor IIH subunit 4)和DNA分子的示例文件(example.pdb),同学们可以直接使用。首先打开 PyMOL 软件。在界面上方的菜单栏中,选择“File” -> “Open”来加载example.PDB 文件,PyMOL 将自动加载并显示蛋白质的三维结构。
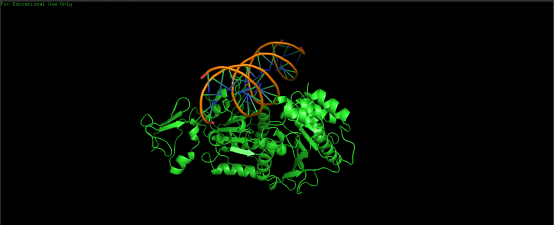
结果显示如上图,同学们可以使用鼠标左键拖动来旋转分子,右键拖动来平移分子,滚动鼠标滚轮来缩放分子的大小。这些操作能够帮助我们更全面地观察和分析蛋白质与 DNA 结构复合体的各个方面。
蛋白质与DNA结合解析
通过以上的步骤,虽然蛋白质和氨基酸结构被清晰显示出来,但是两者之间的结合关系还不够明确。接下来,让我们选中蛋白质,在右边的状态栏中(sele)项目中选择”A”->”find”->”polar contacts”->”to other atoms in object”。
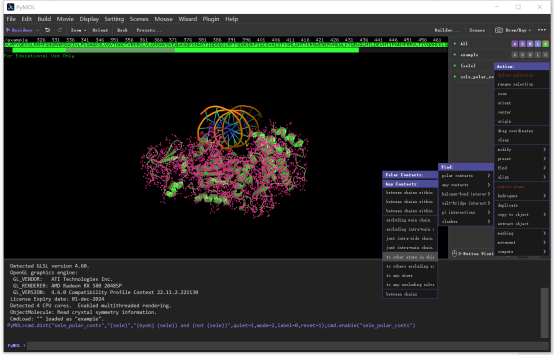
至此就成功显示了蛋白质和DNA相互链接的氢键了,随后我们把蛋白质单独出提取出来,并设置为surface模式显示,代码如下:
extract protein, example and chain V #单独提取蛋白质
cmd.show_as(“surface”,”protein”) #设置蛋白质为surface显示
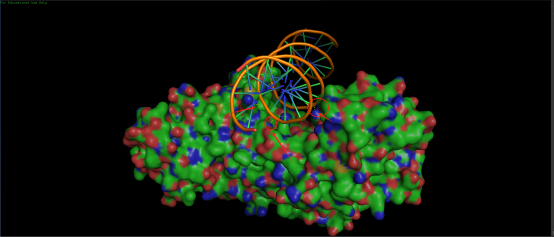
显示如上图,其中黄色虚线即为两者之间形成的氢键。为了让结果更加直观清晰,我们可以调整PyMol的一些参数,代码如下:
# 设置背景颜色为白色
bg_color white
# 设置光线追踪相关参数
set ray_trace_gain, 0.1
set ray_trace_discoeff, 0.1
set ray_trace_mode, 1
set ray_shadows, 1
# 设置光照效果
set ambient, 0.3
set specular, 0.5
set shininess, 75
set spec_direct, 0.3
set spec_direct_power, 200
set reflect, 0.2
# 设置抗锯齿
set antialias, 2
# 调整阴影和透明度的精度
set ray_shadow, 1
set ray_transparency_shadows, 1
set ray_interior_shadows, 1
set ray_trace_depth, 4
# 关闭深度提示和光线追踪雾
set depth_cue, 0
set ray_trace_fog, 0
随后调整好合适的画面角度,我们就可以渲染图像并保存,命令如下:
ray 2000, 2000 #进行渲染
这个过程可能需要消耗一些时间,大家需要耐心等待一下,加载完成之后,依次选择“File”->”Export Image As”->”PNG”。
渲染结果解析:
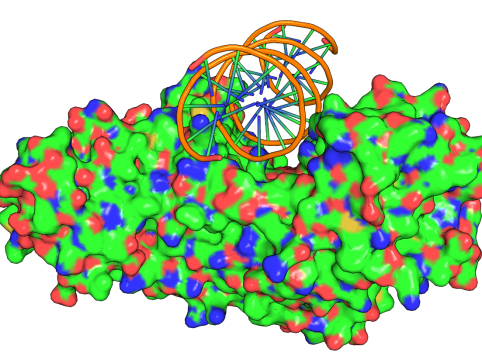
结果如上图所示,蛋白质转录因子(以表面模型表示)与DNA(以双螺旋结构表示)特异性结合,形成稳定的复合物。转录因子通过其特定的结构域识别并结合DNA上的特定序列,通常位于基因的启动子区域或增强子区域。这种结合通常会改变DNA的空间构象,促进或抑制RNA聚合酶的结合和转录起始,最终调控基因的表达。图像中的颜色表示不同氨基酸的分布,突出显示了转录因子与DNA之间的相互作用界面,展示了蛋白质与核酸分子间复杂而精确的结合机制。
以上就是对PyMOL软件的全部介绍了,在本次的学习中,我们探索了PyMOL在分子结构可视化中的重要作用。通过学习如何安装和使用这个强大的工具,我们不仅掌握了分子建模和分析的方法,还进一步理解了生物大分子的结构和功能。希望大家能够充分利用PyMOL,继续探索更多有关分子结构的奥秘,为我们更深入地理解生命的机制做出更多贡献!
同学们如果觉得自己写代码麻烦,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表。立即访问云生信(http://www.biocloudservice.com/home.html),开启便捷的生信之旅!