相信大家都会做富集分析,但是你会把富集分析的结果美观、全面地展示出来吗?一个好的分析结果固然重要,一个好的结果图也同样重要,结果图可以让读者对你的分析结果一目了然,赏心悦目的图片也可以提升你的文章的档次。大家对分析流程都胸有成竹,但是可能会因不知道如何更好地将结果展现出来而苦恼。别担心,今天小花就带大家绘制一种美观的富集分析结果图。要是您有自己做不了的生信分析,可以联系我,如果您的数据量比较大也可以联系小花租赁高性价比的服务器哦。
这是一个普通的富集分析和一张普通的富集条形图:
## 普通的富集分析结果图
## 读进差异分析结果
allDiff = read.csv(“../input/DEGs.csv”, row.names = 1)
## 设置差异阈值为pvalue<0.05 & abs(log2FC)>0.585
allDiff$trend = ifelse(allDiff$pvalue<0.05 & abs(allDiff$logFC)>=0.585,
ifelse(allDiff$logFC>0, “up”, “down”), “stable”)
up_gene = allDiff$gene[which(allDiff$trend==”up”)]
down_gene = allDiff$gene[which(allDiff$trend==”down”)]
## 对上调基因做GO富集分析
library(clusterProfiler) #富集分析主要的包
library(org.Hs.eg.db)#查找物种注释信息
library(ggplot2)#分面绘图所需
library(dplyr)#整理数据
library(ggsci)#配色
library(showtext)
library(scico)
Genes <- bitr(up_gene,
fromType = “SYMBOL”, #输入数据的类型
toType = c(“ENTREZID”), #要转换的数据类型
OrgDb = org.Hs.eg.db)
# 富集分析 ——————————————————————–
GO <- enrichGO(gene = Genes$ENTREZID, #输入基因的”ENTREZID”
OrgDb = org.Hs.eg.db,#注释信息
keyType = “ENTREZID”,
ont = “all”, #可选条目BP/CC/MF
pAdjustMethod = “BH”, #p值的校正方式
pvalueCutoff = 1, #pvalue的阈值
qvalueCutoff = 1, #qvalue的阈值
minGSSize = 5,
maxGSSize = 5000,
readable = TRUE) #是否将entrez id转换为symbol
GO_result <- as.data.frame(GO)
## 显著富集阈值是p.adjust<0.05,count>=2
GO_sig = GO_result[which(GO_result$p.adjust<0.05 & GO_result$Count>=2),]
# 挑选每个分类中前5显著的条目
BP_top5 <- GO_sig %>%
filter(ONTOLOGY == “BP”) %>%
arrange(p.adjust) %>%
head(5)
CC_top5 <- GO_sig %>%
filter(ONTOLOGY == “CC”) %>%
arrange(p.adjust) %>%
head(5)
MF_top5 <- GO_sig %>%
filter(ONTOLOGY == “MF”) %>%
arrange(p.adjust) %>%
head(5)
top5 <- rbind(BP_top5,CC_top5,MF_top5)
top5$ONTOLOGY <- factor(top5$ONTOLOGY, levels = c(“BP”, “CC”,”MF”))
top5$logPvalue <- (-log(top5$p.adjust))
top5$log10P.adjust <- (-log10(top5$p.adjust))
# 美化版本 ——————————————————————–
colnames(top5)
top5$Description <- factor(top5$Description, levels = unique(top5$Description[order(top5$ONTOLOGY)]))
p = ggplot(top5, aes(x = Description, y = log10P.adjust, fill = ONTOLOGY)) +
geom_bar(stat = “identity”, width = 0.7, color = “black”) +
scale_x_discrete(limits = unique(top5$Description[order(top5$ONTOLOGY)])) +
coord_flip() +
scale_y_continuous(expand = c(0,0))+
theme(panel.background = element_rect(fill = “white”))+
theme(axis.text = element_text(size = 38, color=”black”,family = “Arial”),
axis.title = element_text(size=45,family=”Arial”,colour = “black”),
legend.text = element_text(size = 45, family = “Arial”),
legend.title = element_text(size = 45, family = “Arial”),
legend.position = “top”)+
labs(x = “GO term”, y = “-log10 P.adjust”)+
scale_fill_nejm(alpha = 0.8)
showtext_auto()
pdf(“../output/figure1.pdf”,width = 25,height = 15)
print(p)
dev.off()
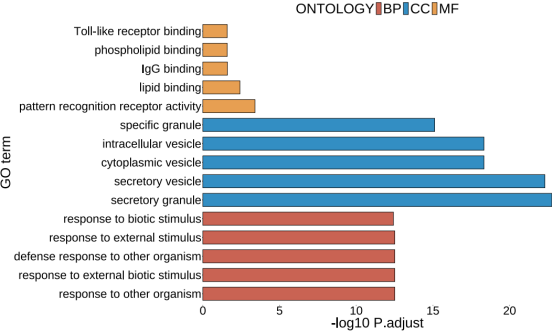
这张富集结果图展示了GO富集分析各个类别富集最显著的前5个条目,它的横坐标是-log10(p.adjust),纵坐标是条目的描述。
有的小伙伴就说了,这个图看着太普通了,就是一个简单的条形图嘛。哎,这个时候我就要介绍一下GOplot这个包了。GOplot,专门用于可视化基因本体论(Gene Ontology, GO)富集分析的结果。它可以绘制条形图,显示每个GO术语的富集分数和显著性;它可以绘制圆形图,显示GO术语的富集分数和显著性;他可以绘制气泡图,显示GO术语的显著性和基因数;它可以绘制和弦图,显示不同GO类别之间的基因重叠。你是否有些心动了呢,下面小花就带大家绘制这些美观的富集分析图。
GOplot需要的数据格式和普通的富集分析结果不太一样,所以我们要先制作准备数据:
## diff是差异分析结果,且只包含了差异基因
diff = allDiff[which(allDiff$trend!=”stable”), -8]
colnames(diff) = c(“ID”, “logFC”, “AveExpr”, “t”, “P.Value”, “adj.P.Val”, “B”)
Genes <- bitr(diff$ID,
fromType = “SYMBOL”,
toType = c(“ENTREZID”),
OrgDb = org.Hs.eg.db)
# 富集分析 ——————————————————————–
GO <- enrichGO(gene = Genes$ENTREZID,
OrgDb = org.Hs.eg.db,
keyType = “ENTREZID”,
ont = “all”,
pAdjustMethod = “BH”,
pvalueCutoff = 1,
qvalueCutoff = 1,
minGSSize = 5,
maxGSSize = 5000,
readable = TRUE)
## 把结果整理成需要的形式
GO_result <- as.data.frame(GO)
GO_result = GO_result[which(GO_result$p.adjust<0.05 & GO_result$Count>=2), c(1, 2, 3, 9, 7)]
colnames(GO_result) = c(“Category”, “ID”, “Term”, “Genes”, “adj_pval”)
GO_result$Genes = apply(GO_result, 1, function(x){
return(gsub(“/”, “, “, x[4]))
})
circ <- circle_dat(GO_result, diff)
head(circ)
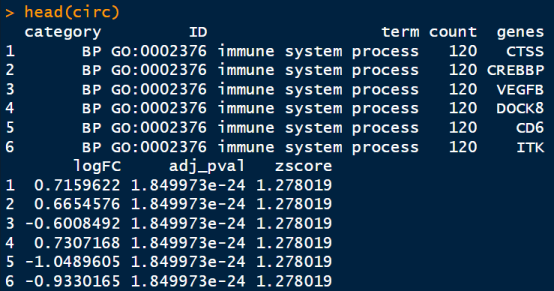
这里的circ就是我们需要的数据了,这里有一列是zscore,它用来表示这个条目中上调基因和下调基因的情况,如果zscore大于0表示这个富集到这个条目的上调基因多,反之则是下调基因多。
这里的up是指富集到这个条目的上调基因的数目,down是指富集到这个条目的下调基因的数目,count是富集到这个条目的所有基因的数目。
首先,我们来展示一下GOplot的条形图:
library(GOplot)
## 展示各个类别最显著的前5个条目
BP_top5 <- GO_result %>%
filter(Category == “BP”) %>%
arrange(adj_pval) %>%
head(5)
CC_top5 <- GO_result %>%
filter(Category == “CC”) %>%
arrange(adj_pval) %>%
head(5)
MF_top5 <- GO_result %>%
filter(Category == “MF”) %>%
arrange(adj_pval) %>%
head(5)
## 条形图
pdf(“../output/figure2.pdf”)
GOBar(subset(circ, ID %in% c(BP_top5$ID, CC_top5$ID, MF_top5$ID)), ## 展示的条目数据
display = “multiple”, ## 分类进行分面
zsc.col = c(‘red’, ‘black’, ‘blue’)) ## 设置z-score色阶,依次为c(high, midpoint, low)
dev.off()
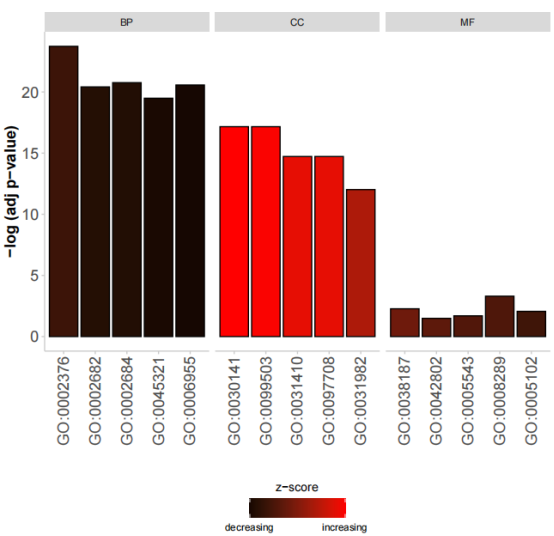
这里其实也是条形图,但是多了颜色,从颜色中我们可以知道这个条目整体是上调的还是下调的。但是因为这几个条目的zscore都是正的,所以设置的色阶中蓝色没有显现出来。
然后我们再来看一下气泡图:
## 气泡图
pdf(“../output/figure3.pdf”)
GOBubble(circ, colour = c(‘orange’, ‘forestgreen’, ‘purple’), ## 设置每个类型的颜色
display = ‘multiple’, labels = 10 ## 显示y>10的标签
)
dev.off()
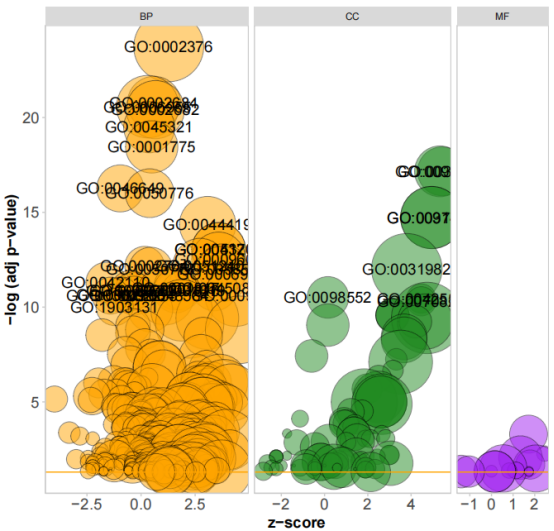
这里横坐标就是zscore,但是这样看着太乱了,这是可以把相似性比较高的条目删掉:
## 很明显可以看到条目太多了,显示会挤在一起
## 删除基因重叠大于或等于 0.75的 terms(具体不知道按什么原则删的)
reduced_circ <- reduce_overlap(circ, overlap = 0.75)
pdf(“../output/figure4.pdf”)
GOBubble(reduced_circ, colour = c(‘orange’, ‘forestgreen’, ‘purple’), ## 设置每个类型的颜色
display = ‘multiple’, labels = 10 ## 显示y>10的标签
)
dev.off()
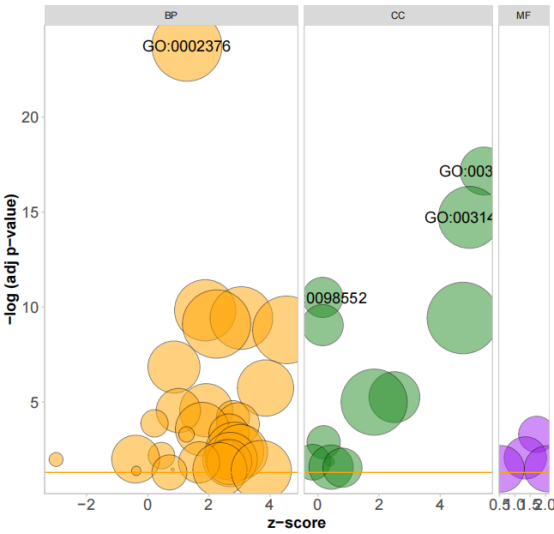
这样看着就清爽很多了。然后我们再来看一下圈图:
## # 可视化感兴趣的条目
IDs <- c(BP_top5$ID[1:3], CC_top5$ID[1:3], MF_top5$ID[1:3])
pdf(“../output/figure5.pdf”, width = 13, height = 7)
GOCircle(circ, nsub = IDs)
dev.off()
# 可视化前5个条目
pdf(“../output/figure6.pdf”, width = 10, height = 5)
GOCircle(circ, nsub = 5)
dev.off()
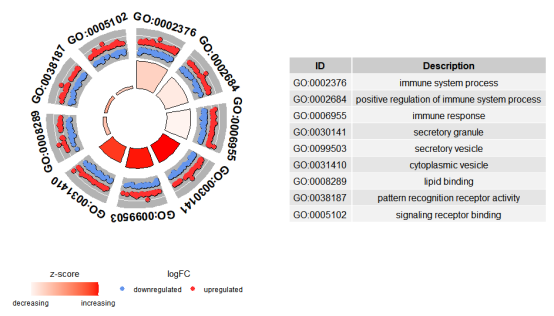
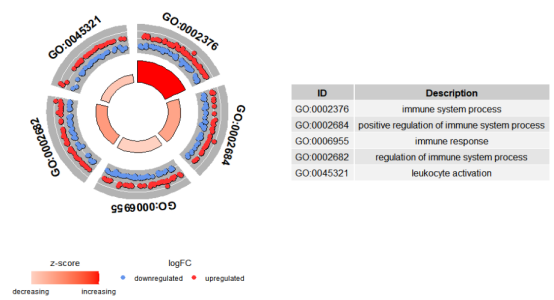
最外圈显示了富集基因的上调和下调情况,内圈颜色表示zscore的大小,内圈高低表示富集结果的显著性。我们再来看一下弦图:
## 弦图
chord <- chord_dat(data = subset(circ, ID %in% c(BP_top5$ID[1:3], CC_top5$ID[1:3], MF_top5$ID[1:3])),
genes = diff[,c(1, 2)])
pdf(“../output/figure7.pdf”)
GOChord(chord, space = 0.02, gene.order = ‘logFC’, gene.space = 0.25, gene.size = 3,
limit = c(3, 30)) ## 展示基因至少富集到三个条目,展示条目至少有30个基因富集
dev.off()
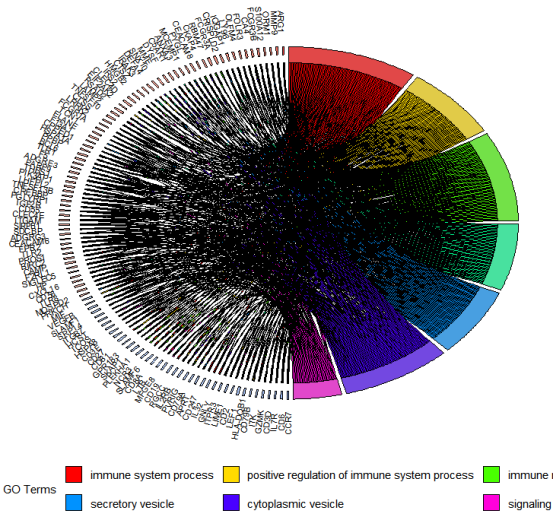
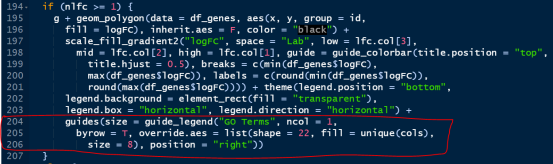
很难受的一点就是无法调整图例的排列和位置,导致很多图例看不到,而且函数没有提供参数来修改。这时候我们可以通过修改源代码来解决。
trace(GOChord, edit = TRUE)
pdf(“../output/figure8.pdf”, width = 10, height = 5)
GOChord(chord, space = 0.02, gene.order = ‘logFC’, gene.space = 0.25, gene.size = 3,
limit = c(3, 30)) ## 展示基因至少富集到三个条目,展示条目至少有30个基因富集
dev.off()
在所示位置添加这几行代码即可:设置GO Terms图例位置在右边,且图例的列是一列。代码已经为大家准备好了,大家可以在code/GO_Chord.R复制粘贴即可。
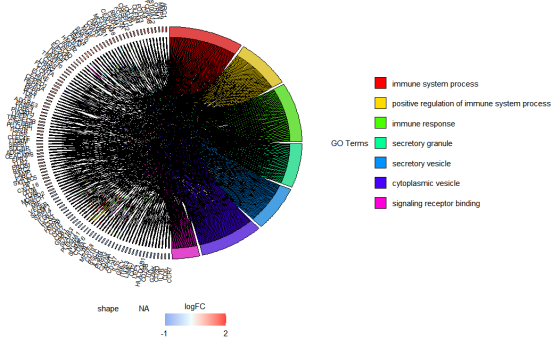
这样弦图就显示完全了,弦图展示了所选基因和条目之间的关系,以及这些基因的logFC。
今天的富集分析结果画图方法就分享到这里,希望对大家能有所帮助,如果大家发现文中有不对的地方或有其他疑问,欢迎留言分享。如果各位觉得自己运行代码太麻烦,欢迎用我们的云生信小工具(http://www.biocloudservice.com/home.html),只要输入合适的数据就可以直接出想要的图呢。