嘿朋友们,小果向导又来啦!今天,我们要一起挑战的是生物过程中的调控网络,这可是个大有可为的任务哦!为了更好地解决这个问题,我们将借助当前非常热门的人工智能技术。让我们一起揭开这个神秘领域的面纱,开始我们的探索之旅吧!
生物过程,比如细胞/组织的分化,涉及到许多转录因子和信号输入的调控网络。这些网络的复杂性以及它们在控制下游基因网络方面的作用,至今仍存在很多未解之谜。但别担心,有了LedPred这个神奇的包,我们就可以轻松预测新的顺式调节模块啦!通过支持向量机(SVM)学习转录因子结合位点(TFBS)的模式、二代测序数据信号,以及预先计算的数值,最终我们就能成功预测出新的顺式调节模块(CRMs)啦!这个R包操作占用内存比较大,建议使用服务器哦,欢迎联系小果租赁性价比高的服务器~
接下来,小果将引领大家逐步掌握操作流程,从使用已知的调控元素开始,到预测新的顺式调控模块,我们将一同揭示生物过程中调控网络的奥秘。如果你在旅途中遇到任何困难,别担心,小果随时待命,记得联系小果为你解答疑惑~
我们先安装并加载LedPred包:
if (!require(“BiocManager”, quietly = TRUE))
install.packages(“BiocManager”)
BiocManager::install(“LedPred”)
library(LedPred)
一、构建训练集
在LedPred包中,mapFeaturesToCRMs函数能够帮助我们从bed CRM 基因组坐标等文件中处理数据得到训练集,感兴趣的小伙伴可以自行尝试。在这里,可以直接调用已经处理好的数据~
crm.features <- read.table(“crm.features.txt”, header = TRUE, sep = “\t”)
这批数据是通过矩阵扫描和评分果蝇的基因组序列来生成的,主要关注的是参与心脏分化的区域。我们使用一个特征矩阵作为训练集,每个基因组区域一行,每个特征一列,还有一个表示正负序列的二进制响应列。
二、模型最优参数选择
使用机器学习方法,结合位置特异性评分矩阵(PSSMs)的信息,可以构建一个能够区分正负样本的模型。支持向量机(SVM)模型可以采用线性核或径向核。线性核假设存在线性超平面可区分正负样本,而径向核则将描述性特征空间映射到更高维度的空间,适用于无法通过线性超平面区分正负样本的情况。
在优化过程中,引入代价参数C和γ参数有助于避免过拟合,增加模型的通用性。为了找到最佳的参数组合,可以使用mcTune函数来测试不同的C和γ参数,通过交叉验证计算预测误差,找到最佳参数。首先可以定义 cost.vector 和 gamma.vector,分别包含了数值 1, 3, 10, 和 30。这些值被用作 mcTune 函数调优参数的候选值。
gamma.vector <- c(1,3,10,30)
接下来调用mcTune 函数,传入数据集data = crm.features及刚刚定义的调优参数候选值,定义file.prefix可以自动保存调参的可视化图。注意哦,若没有指定kernel时,默认使用的是径向核,因此我们也可以修改参数kernel为’linear’,也就是线性核。
#c.g.obj <- mcTune(data = crm.features, ranges = list(cost=cost.vector, gamma=gamma.vector), file.prefix = “vignette_2”)
c.g.obj <- mcTune(data = crm.features, ranges = list(cost=cost.vector, gamma=gamma.vector), kernel=’linear’, file.prefix = “vignette_2”)
mcTune 函数返回一个名为 c.g.obj 的对象,其中包含了模型调优的结果。使用 names(c.g.obj) 函数可以查看 c.g.obj 对象中包含的内容的名称。

接下来从 c.g.obj 对象中提取我们想要的最佳的 cost 和 gamma 参数值。
cost <- c.g.obj$e1071.tune.obj$best.parameters$cost
gamma <- c.g.obj$e1071.tune.obj$best.parameters$gamma
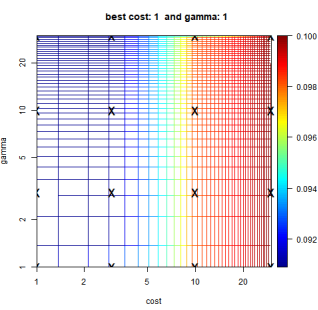
让我们打开看看mcTune 函数自动保存的调参可视化图吧!图中显示了不同 cost 和 gamma 值的网格搜索结果,颜色表示某种性能指标(例如误差率或准确率)。在图中,cost 和 gamma 的最佳组合(即性能指标最优的点)位于 cost = 1 和 gamma = 1 的位置。
三、特征重要性排序和特征选择
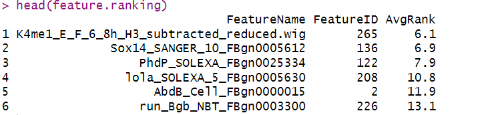
利用递归特征消除(RFE),可以计算基于”重要性”降序排列的特征列表。在这个过程中,SVM将在特征矩阵的子集上进行训练,然后根据分类器给出的权重对特征进行排序,每个特征将被分配一个平均排名。使用rankFeatures函数执行RFE,可是实现特征的重要性排序。
feature.ranking <- rankFeatures(data=crm.features, cost=cost, gamma=gamma, kernel=’linear’, file.prefix = “vignette”)
head(feature.ranking)
接下来,我们就可以使用tuneFeatureNb函数根据特征的重要性进行降序排列,并在特征矩阵上计算kappa度量。它从一个特征开始,逐步增加包含在矩阵中的特征数量(通过`nb.step`参数),直到所有特征都被包含。
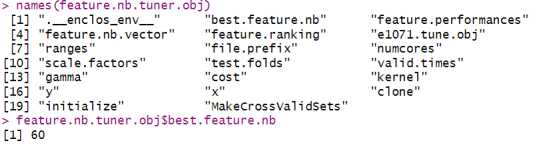
feature.nb.tuner.obj <- tuneFeatureNb(data=crm.features, feature.ranking=feature.ranking, kernel=’linear’, cost=cost, gamma=gamma, file.prefix = “vignette”)
函数返回的对象总结了模型中包含的不同数量的特征的kappa系数,我们可以保留对应于最高kappa的特征数量,在这里为前60个特征。
names(feature.nb.tuner.obj)
feature.nb.tuner.obj$best.feature.nb
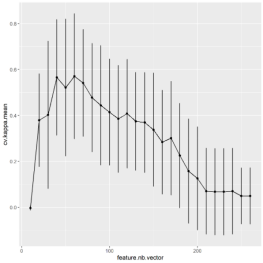
分析tuneFeatureNb函数自动生成的图,我们可以发现:选择特定数量的特征可以获得最佳的模型性能,在这里是选择前60个特征时模型表现最佳。
四、模型的构建
在确定了最优参数和特征后,我们就可以创建出最佳模型啦! createModel 函数可以使用调整后的 SVM 参数、排序的特征和最佳特征数量,并返回最佳的 SVM 模型。
feature.nb.tuner.obj <- tuneFeatureNb(data=crm.features, feature.ranking=feature.ranking, kernel=’linear’, cost=cost, gamma=gamma, file.prefix = “vignette”)
names(feature.nb.tuner.obj)
feature.nb.tuner.obj$best.feature.nb
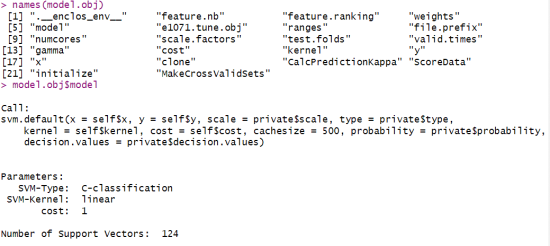
以上输出提供了关于所创建的 SVM 模型的重要信息,包括了模型参数的设置、核函数类型、超参数值和支持向量的数量等,有助于我们理解模型的结构和性能特点。
另外,我们还可以输出特征在这个模型当中的权重哦,通过观察特征权重的正负号,可以判断该特征在正类别和负类别中的重要性,以及该特征是被过度表示还是不足表示。
feature.weights <- as.data.frame(t(t(model.obj$model$coefs) %*% model.obj$model$SV))
head(feature.weights)
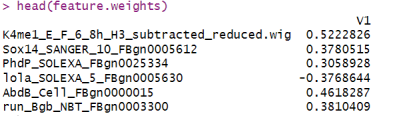
特征权重的正负号可以告诉我们特征对模型的预测结果具有正向影响(正权重)还是负向影响(负权重)。展示特征权重的前几行内容有助于我们了解模型对特征的重要性排序以及特征在预测中的作用。
evaluateModelPerformance函数可以返回在交叉验证轮次中计算的预测概率,并绘制性能结果。
probs.labels.list <- evaluateModelPerformance(data=crm.features, feature.ranking=feature.ranking, feature.nb=feature.nb, cost=cost, gamma=gamma, file.prefix = “vignette”)
这个函数可以生成下面4张模型评估图:Precision 和 Recall 随着阈值变化的曲线、Precision 和 Recall 权衡关系图、ROCR 曲线(其中包含曲线下面积AUC)
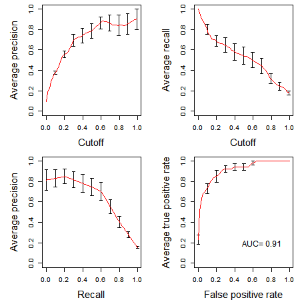
通过分析图中的曲线形态、指标数值以及 AUC 值,可以得出对于模型性能的结论和评估。例如,该模型的Precision会随着阈值的升高而升高,但是 Recall 曲线却随着阈值升高而下降;当 Precision 和 Recall 曲线距离右上角不是很近,表示模型在不能很好地兼顾 Precision 和 Recall;AUC 值为0.91,比较接近1,说明模型具有很好的分类能力。综合各个性能指标的表现可以提供全面的模型评估和决策依据。
接下来,我们终于可以使用构建好的模型进行预测啦!我们可以使用coreData 函数对未知矩阵的序列进行评分。
prediction.crm.features <- read.table(“prediction.crm.features.txt”, header = TRUE, sep = “\t”)
pred.test <- scoreData(data=prediction.crm.features, model=model.obj, score.file=”vignette_prediction.tab”)
head(pred.test)
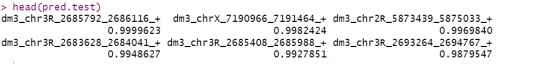
结果显示,模型对测试集中的数据预测值都非常接近1,这和真实值相符,这说明我们构建模型的泛化能力还不错哦!
- 一键建模的神奇函数
最后,小果要给大姐强烈推荐一个神奇函数——LedPred,只要将数据喂给这个函数,它就可以自动完成模型训练和调优,直接省去了5个步骤,一键构建得到最佳模型!
ledpred.obj=LedPred(data=crm.features, cl=1, ranges = list(cost=cost.vector, gamma=gamma.vector), kernel=”linear”, file.prefix=”vignette2″)
pred.test <- scoreData(data=prediction.crm.features, model=ledpred.obj$model.obj, score.file=”vignette_prediction.tab”)
head(pred.test)

怎么样,AI的世界是不是很神奇?好啦,小果已经带领大家一起完成了LedPred包的初步探索,你有没有对AI在生物调控网络上的应用有了感性的认识呢?现在,你可以开始自己动手,利用LedPred包来构建属于你自己的模型啦!
无论你是在优化代码,还是在云端进行便捷的分析,云生信神器都能为你提供强大的支持。欢迎试试我们的云生信神器,只需一键上传数据,想要的图就能轻松get~