
大家好,小果来啦,很高兴又和大家见面了!今天我们要学习的是一个非常有用的R包——motifStack。motifStack包专门用于绘制和可视化DNA、RNA和蛋白质序列的基序,通过它我们可以生成高质量的序列logo图,帮助我们直观地展示序列中各个位置的碱基或氨基酸的保守性和频率分布。没有任何生信分析能难倒生信高手小果,要是同学们有自己做不了的生信分析,欢迎随时联系我!!!
在今天的课程中,我将详细讲解如何安装motifStack包,并演示如何使用它来绘制丰富多彩的DNA序列logo。我们会从包的基本安装开始,逐步介绍如何载入数据、绘制序列logo,并展示如何调整绘图参数以及添加注释标记,使得图形更加美观和信息丰富。相信通过今天的学习,大家一定能够掌握motifStack包的基本用法,并在自己的研究中灵活应用。那就让我们正式开启motifStack包的学习之旅吧!
本次介绍的R包需要较多的硬件资源,在服务器可以更加流畅运行,同学们如果没有自己的服务器欢迎联系我们进行服务器租赁~
motifStack介绍
motifStack是一个专门用于绘制和可视化DNA、RNA和蛋白质序列基序的R包。它能够生成高质量的序列logo图,这些图可以帮助同学们直观地展示序列中各个位置的碱基或氨基酸的保守性和频率分布。motifStack包支持多种配色方案和字体,用户可以根据需要自定义图形的外观。此外,它还提供了添加注释标记的功能,使得序列logo图更具信息性和可读性。通过motifStack包,研究人员能够更加方便地进行序列分析和结果展示,提升数据可视化的效果和研究的直观性。
motifStack包的安装
需要R语言版本为4.3,在控制台中输入以下命令:
if (!require(“BiocManager”, quietly = TRUE))
install.packages(“BiocManager “)
BiocManager::install(“motifStack”) # 在BiocManager环境下安装motifStack
查看是否安装成功
packageVersion(“motifStack”) # 查看motifStack版本
显示为1.46.0版本,则表示已经安装了motifStack包。
成功安装motifStack包后,我们接下来将演示如何使用motifStack包来绘制丰富多彩的序列LOGO(sequence logo),小果会讲的很仔细,同学们可要认真听讲哦。
使用motifStack包绘制DNA序列logo示例
包和数据载入:
进行DNA序列logo绘制,我们首先需要载入需要用到的包和数据,本篇文章演示为了让同学们更容易理解,将只采用motifStack包自带的数据集,同学们如果需要完成自己数据DNA序列logo绘制,进行对应的替换即可,相关命令如下:
# 导入motifStack包,该库用于操作和可视化基序(motif)
library(motifStack)
# 读取包含位置权重矩阵(position count matrix,PCM)的文本文件
# 使用find.package函数找到motifStack包的安装路径,并构建文件路径
pcm <- read.table(file.path(find.package(“motifStack”),
“extdata”, “bin_SOLEXA.pcm”))
# 去除数据框的前两列,只保留第三列及其后的列
pcm <- pcm[,3:ncol(pcm)]
# 设置数据框的行名,代表碱基A、C、G、T
rownames(pcm) <- c(“A”,”C”,”G”,”T”)
# 查看处理之后的pcm数据框
pcm
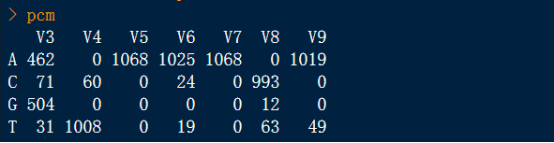
结果显示如上图,可知共有1068个序列的七个位点信息,其中列为序列中的DNA位点,行为碱基,单元格为该碱基在对应位点出现的次数。接下来我们可以构建一个pcm(position count matrix,位置权重矩阵)对象来绘制对应序列的logo,相关命令如下:
# 创建一个新的pcm对象,使用as.matrix将数据框转换为矩阵
# 将矩阵存储在motif对象的mat属性中,并设置motif对象的名称为”bin_SOLEXA”
motif <- new(“pcm”, mat=as.matrix(pcm), name=”bin_SOLEXA”)
# 绘制motif对象
plot(motif)
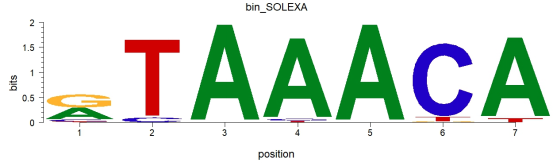
显示如上图,即成功绘制了示例数据DNA的序列logo,从图中我们可知2-7号位点相对保守,可能是与DNA功能相关的一些重要位点。接下来我们还可以对绘制序列logo的参数进行调整,让图像更加美观,满足不同研究的需求。
绘制参数调整
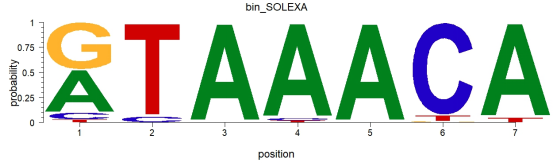
我们可以对plot的参数进行相应的调整,来获得我们想要的效果,例如让y轴标为碱基的概率分布,代码如下:
#绘制碱基概率分布的DNA序列logo
plot(motif, ic.scale=FALSE, ylab=”probability”)
改变x轴为自定义刻度,代码如下:
#绘制自动x轴刻度的DNA序列logo
plot(motif, xaxis=paste0(“pos”, seq.int(7)+10))
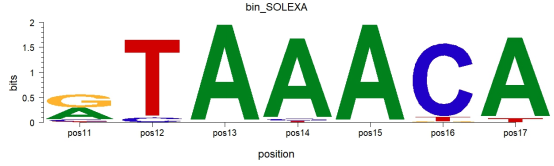
自定义配色方案和字体,代码如下:
#绘制指定配色方案和字体的DNA序列logo
motif@color <- colorset(colorScheme=’basepairing’)
plot(motif,font=”serif”)
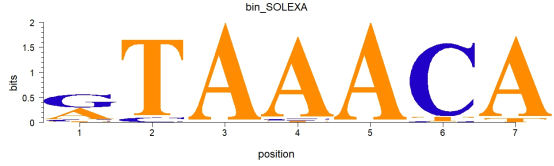
对于DNA 和 RNA可用的配色方案有’auto’, ‘basepairing’, or ‘blindnessSafe’,可用的字体有’sans’,’serif’,’mono’,’Helvetica’ 等绘图常用字体。
添加注释标记
想要让DNA序列logo更加直观易懂,除了对于plot()函数的基础参数调整外,我们还可以直接在logo图上添加marker标记,相关代码如下:
# 创建一个矩形标记对象,标记从第6到第7位,设置为虚线边框,橙色,无填充
markerRect <- new(“marker”, type=”rect”, start=6, stop=7, gp=gpar(lty=2, fill=NA, col=”orange”))
# 创建一个线条标记对象,标记从第2到第7位,设置为红色,线宽为2
markerLine <- new(“marker”, type=”line”, start=2, stop=7, gp=gpar(lwd=2, col=”red”))
# 创建一个文本标记对象,标记第1和第5位,设置为红色,字体大小为2,文本标签为“*”和“core”
markerText <- new(“marker”, type=”text”, start=c(1, 5),
label=c(“*”, “core”), gp=gpar(cex=2, col=”red”))
# 构建一个新的pcm对象,并且添加markerRect, markerLine, markerText三个注释。
motif <- new(“pcm”, mat=as.matrix(pcm), name=”bin_SOLEXA”,
markers=c(markerRect, markerLine, markerText))
#绘制motif对象
plot(motif)
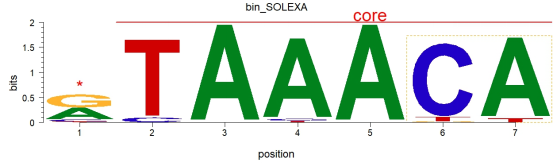
在数据可视化中,尤其是生物信息学中的序列logo绘制时,添加注释标记能够极大地提升图形的直观性和信息量。如图所示,代码分别创建了一个矩形标记对象,标记从第6到第7位,设置为虚线边框,矩形标记常用于强调某些关键区域,比如在DNA序列中标注重要的保守区域、功能域或结构域。一个线条标记对象,标记从第2到第7位,设置为红色,线宽为2,线条标记可以用来表示某些功能元件的范围,比如基因调控元件、蛋白质结合位点等。一个文本标记对象,标记第1和第5位,设置为红色,字体大小为2,文本标签为“*”和“core”,文本标记可以用于标注关键位点,比如突变位点、结合位点等,提供详细的注释信息。通过将矩形标记、线条标记和文本标记添加到绘制的DNA 序列logo上,可以显著提升序列logo的直观性和信息丰富度。这些标记不仅能够强调重要区域和位点,还能通过详细的注释使得图表更加易于理解和解释,对于同学们进行科学研究和成果展示有着重要的意义。
以上,就是对motifStack包的全部介绍了。小果希望同学们通过本文的学习,能够掌握motifStack包的基本用法,并能在自己的研究中灵活应用,继续探索和学习更高级的功能,提升自己的数据可视化能力。如果需要更多帮助或硬件支持,欢迎联系我们。祝同学们学习愉快!
同学们如果觉得自己写代码麻烦,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表。立即访问云生信(http://www.biocloudservice.com/home.html),开启便捷的生信之旅!