大家好啊,相信大家已经学会如何从头处理芯片原始数据了,在公共数据库中我们还经常会遇到转录组数据,转录组数据是无法用R包下载的,因为转录组数据的Series Matrix File(s)没有基因表达量数据,只有临床数据,所以每次处理转录组数据我们都要在Supplementary file下载文件然后处理,但是很不巧的是很少有作者会上传他们整合好的数据,大部分作者都会上传未整合的数据,这时候我们需要从头开始整合数据。下面我们就以GSE220042为例来教大家如何处理并分析RNA-seq的数据。要是您有自己做不了的生信分析,可以联系我。
首先下载这里的补充文件,
这是样本量比较小的数据,有的样本量比较大的数据可能会有好几个G,我们可能需要使用服务器来处理,如果您的数据量比较大可以联系小花租赁高性价比的服务器哦。因为服务器一般用命令行来下载数据,所以这时候需要稍微操作一下,建议使用axel多线程地下载数据,axel可以通过conda安装:
axel -n 20 ftp://ftp.ncbi.nlm.nih.gov/geo/series/GSE号前几位nnn/GSE号/suppl/文件名
以我们的示例数据为例:
axel -n 20 ftp://ftp.ncbi.nlm.nih.gov/geo/series/GSE220nnn/GSE220042/suppl/GSE220042_RAW.tar
下面我们就来看一下如何处理这个数据吧。首先将数据下载下来并解压,
然后用我提供的这个代码进行数据合并:
## 获取样本信息
library(GEOquery)
GSE_id = “GSE220042”
gset <- getGEO(GSE_id, destdir=”../output/”,
AnnotGPL = T, ## 注释文件
getGPL = T) ## 平台文件
pd = pData(gset[[1]])
## 读取原始数据
#### 先看一下我们想要哪些列
library(data.table)
files = list.files(paste0(“../input/”, GSE_id, “_RAW/”), full.names = TRUE)
samples = str_extract(files, “(GSM\\d+)_”, group = 1)
tmp = fread(files[1])
head(tmp)
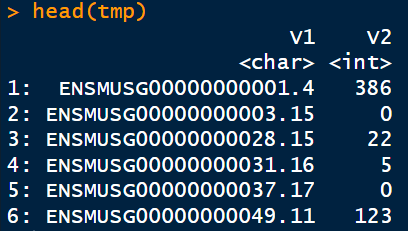
这个数据只有一个V1:ensembl_id和V2:count。下面我们来合并这些数据:
all_data = list()
for(i in 1:length(files)){
all_data[[i]] = as.data.frame(fread(files[i]))
rownames(all_data[[i]]) = all_data[[i]]$V1
all_data[[i]] = all_data[[i]][, -1, drop = FALSE]
colnames(all_data[[i]])[1] = samples[i]
}
data_count = Reduce(cbind, all_data)
head(data_count)
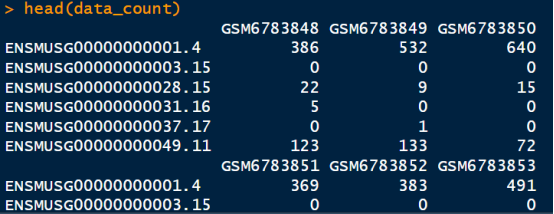
然后开始对基因进行注释,并且相同基因取均值:
## 注释基因名
library(clusterProfiler)
library(org.Mm.eg.db)
library(stringr)
ori_id = data.frame(id = rownames(data_count))
ori_id$ori = str_extract(ori_id$id, “(.*?)\\.”, group = 1)
geneName = bitr(ori_id$ori,
fromType = ‘ENSEMBL’,
toType = ‘SYMBOL’,
OrgDb = ‘org.Mm.eg.db’)
geneName = merge(ori_id, geneName, by.x = “ori”, by.y = “ENSEMBL”)
data_count$id = rownames(data_count)
data_count = merge(data_count, geneName, by.x = “id”, by.y = “id”)
data_count_unique = aggregate(data_count[,c(2:13)], by = list(data_count$SYMBOL), mean)
rownames(data_count_unique) = data_count_unique$gene
data_count_unique = data_count_unique[,-1]
## 只要均值大于1的基因
data_count_unique = data_count_unique[which(rowMeans(data_count_unique)>1),]
write.csv(data_count_unique, “../output/data_GSE220042_count.csv”)
然后进行差异分析:
## 差异分析
brefInfo = data.frame(sample = rownames(pd), type1 = pd$`genotype:ch1`)
brefInfo$group = ifelse(brefInfo$type1==”WT”, “control”, “disease”)
write.csv(brefInfo, “../output/brefInfo_GSE220042.csv”)
library(DESeq2)
## 因为前面取了均值,所以会有小数,这里用round只保留整数部分
brefInfo$group = factor(brefInfo$group, levels = c(“control”, “disease”)) ## 感兴趣的类别放后面
dds <- DESeqDataSetFromMatrix(countData = round(data_count_unique), colData = brefInfo, design = ~ group)
dds1 <- DESeq(dds, fitType = ‘mean’, minReplicatesForReplace = 7, parallel = FALSE)
res <- results(dds1)
summary(res)
res1 <- data.frame(res, stringsAsFactors = FALSE, check.names = FALSE)
res1 <- res1[order(res1$log2FoldChange, res1$pvalue, decreasing = c(TRUE, FALSE)), ]
## 设置差异阈值pvalue<0.05且abs(log2FC)>1
res1$regulation = ifelse(res1$pvalue<0.05 & abs(res1$log2FoldChange)>1,
ifelse(res1$log2FoldChange>0, “up”, “down”), “stable”)
head(res1)
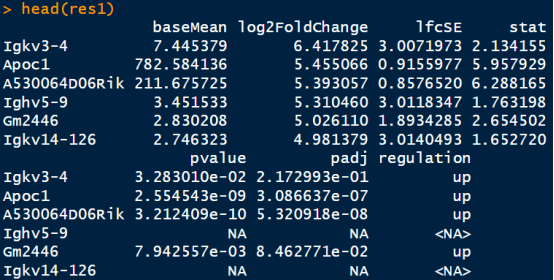
但是有时候结果里会有一些NA,可能是因为这些原因:
- 所有样本计数为零:如果在一行中,所有样本的计数都为零,则基础平均值(baseMean)列将为零,log2折叠变化估计、p值和调整后的p值都将被设置为NA。
- 低平均归一化计数:如果一行的平均归一化计数较低,它可能会被DESeq2自动独立过滤掉,这种情况下,只有调整后的p值将被设置为NA。
- 过滤器移除:在DESeq2的预过滤步骤中,可能会移除表达量极低的基因,这也可能导致p值为NA。
- 分散估计问题:在估计基因表达的分散度时,如果无法准确估计,也可能导致p值为NA。
- 样本大小不足:在某些情况下,如果样本量太小,可能无法进行有效的统计推断,这也可能导致p值为NA。
做完差异分析我们还有后续分析,一般需要提取出DESeq结果中的标准化数据,DESeq2在标准化转录组数据时,采用的是一种称为“size factor”的方法。这种方法的目的是消除样本间由于测序深度不同导致的系统偏差。DESeq2会计算每个样本的size factor,并用它来调整每个样本中基因的计数值,从而使得不同样本之间的计数值可比。这种方法假设大多数基因的表达在所有样本中是不变的,因此可以通过比较所有样本中基因表达的中位数来估计测序深度的变化。这样,即使不同样本的测序量不同,通过size factor标准化后,也可以在样本间进行公平的比较。DESeq2的这种标准化方法是专门为计数数据设计的,它考虑了计数数据的离散性质,并且能够适应不同的测序深度和样本复杂性,这在转录组学数据分析中非常有用。
## 提取标准化后的数据
data_norm = counts(dds1, normalized=TRUE)
boxplot(data_norm)
boxplot(log2(data_norm+1))
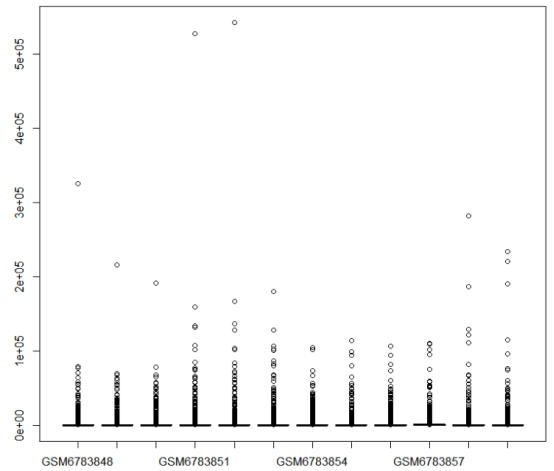
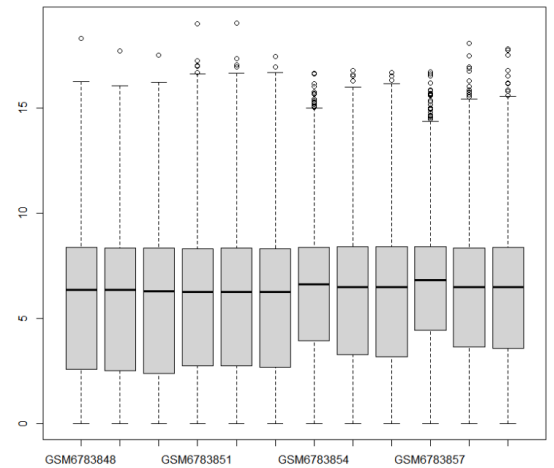
根据你的需求取标准化后的数据或者log2(标准化数据)做后续分析就可以了。
好了,今天的知识就分享到这里了,希望对大家能有所帮助,如果大家有其他疑问或者发现文中有不对的地方,欢迎留言分享。如果各位觉得自己运行代码太麻烦,欢迎用我们的云生信小工具(http://www.biocloudservice.com/home.html),只要输入合适的数据就可以直接出想要的图呢。