持续安利免疫浸润分析工具的小编又来啦~前边几期给大家分享了很多的工具,有同学留言问小编cibersort的教程呢?呐~这不就来了,重要的总要在后边登场。各位同学请放心,cibersort的教程,虽晚必到。
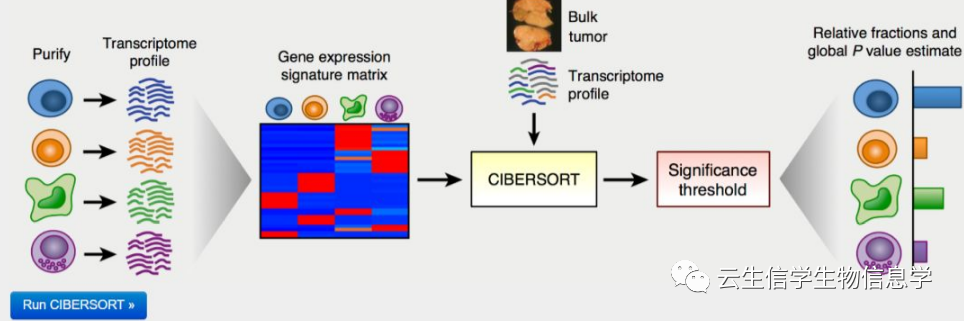
cibersort是目前引用次数最多的免疫细胞浸润估计分析工具,2015年首次发表于Nature method。CIBERSORT是基于线性支持向量回归(linear support vector regression)原理对人类免疫细胞亚型的表达矩阵进行去卷积的工具。用于芯片表达矩阵和测序表达矩阵,对未知混合物和含有相近的细胞类型的表达矩阵的去卷积分析优于其他方法 (LLSR,LLSR,PERT,RLR,MMAD,DSA) 。该方法基于已知参考数据集,默认提供22种免疫细胞亚型的基因表达特征集:LM22。
LM22.txt获取方法: 在Cibersort论文中下载Supplementry table 1 https://www.nature.com/articles/nmeth.3337#MOESM207 CIBERSORT同时提供R包和在线网页工具,可以对用户的表达矩阵进行非负矩阵分解后计算不同细胞类型的比例。
CIBERSORT网页版链接为:https://cibersortx.stanford.edu/ 需要说明的是,使用网页版需要先进行账号注册。
CIBERSORT需要两个准备文件,芯片或者测序表达矩阵以及参考数据集。
表达矩阵格式第一列是基因名,第一行是样品名,不能有重复基因名,第一列列名不能空白。矩阵中不能存在空白或NA值,不要对表达量取Log2。
网页版有LM22的参考数据集,只需要提供表达矩阵文件就可以了。
具体操作如下: 点击运行,等待结果即可。
Cibersort R 代码操作: 在R中新建R Script,复制以下网址中代码,保存为“Cibersort.R” https://rdrr.io/github/singha53/amritr/src/R/supportFunc_cibersort.R (4)以上三个文件需保存在同一文件夹,运行Cibersort的代码如下: 在同一文件夹下可以得到运算结果(”CIBERSORT-Results.txt”) 注意Cibersort结果的默认文件名为CIBERSORT-Results.txt,在同一文件夹下进行第二次运算会覆盖第一次得到的文件,建议在每一次运算之后对文件重命名。
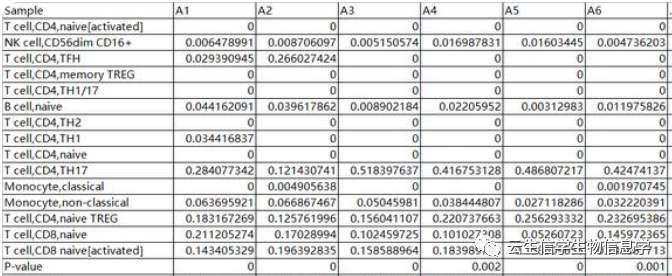
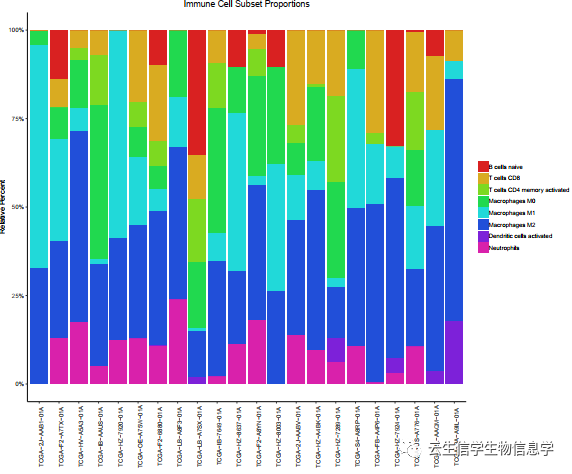
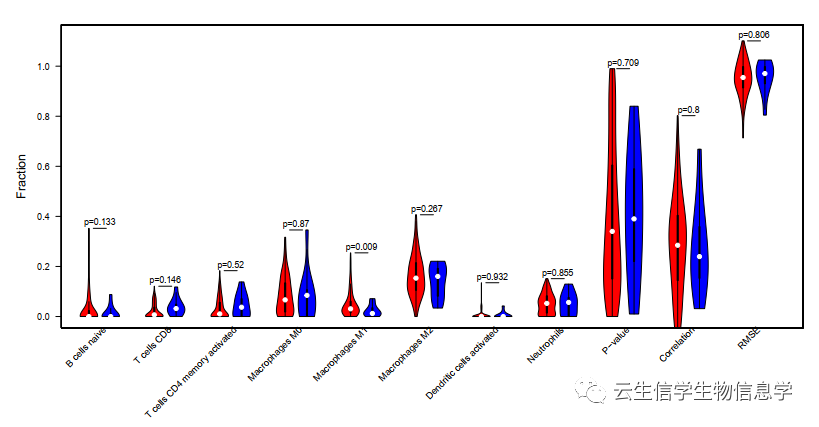
运行结果示例: 基于结果表格,可以进行多种可视化展示,比如箱线图、柱状图等。 cibersort作为目前被引用次数最多的免疫浸润工具是不是也不难,如果大家感兴趣的话,可以做起来啦~~


setwd("三个文件的文件夹")source('Cibersort.R')result <- CIBERSORT('LM22.txt','DATA.txt', perm = 1000, QN = T)