
桑基图(Sankey diagram),即桑基能量分流图,也叫桑基能量平衡图。它是一种特定类型的流程图,图中延伸的分支的宽度对应数据流量的大小,比较适用于用户流量等数据的可视化分析。因1898年Matthew Henry Phineas Riall Sankey绘制的“蒸汽机的能源效率图”而闻名,此后便以其名字命名为“桑基图”。
感兴趣的小伙伴可以去看看Charles Minard在1869年所作的拿破仑东征俄国的信息图,用到的就是桑葚图展示的哦。

那么桑基图要怎么看?小果在这里为大家梳理一下!
(1)看线条的走向(2)看粗细的变化(3)看节点间的比较
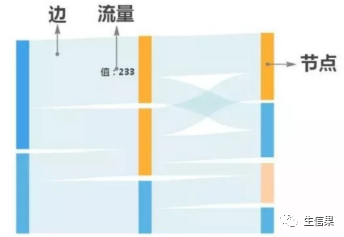
桑基图主要由边、流量和节点组成,其中边代表了流动的数据,流量代表了数据流动的具体数值,节点代表了不同的分类,边的宽度与流量成比例,边越宽,数值越大。
桑基图可以运用的场景主要有:
场景一:
miRNA和靶基因的关系。
场景二:
一个group包含多个基因,同一个基因的不同突变可能属于多个group。像例图一样,展示高频突变基因所处的分组。
场景三:
人群按性别、年龄、家族史等特征分组,展示不同分组得癌症的规律。

下面是具体的代码实战部分:
###读取数据,查看数据
df <- read.table(“sankey.txt”,sep = “t”,row.names = 1,header = T)
head(df)
###install.packages(“ggalluvial”)
###加载R包
library(ggalluvial)
#定义颜色,后面从这里选颜色
mycol <- rep(c(“#223D6C”,”#D20A13″,”#FFD121″,”#088247″,”#11AA4D”,”#58CDD9″,”#7A142C”,”#5D90BA”,”#029149″,”#431A3D”,”#91612D”,”#6E568C”,”#E0367A”,”#D8D155″,”#64495D”,”#7CC767″),3)
#格式转换
UCB_lodes <- to_lodes_form(df[,1:ncol(df)],
axes = 1:ncol(df),
id = “Cohort”)
dim(UCB_lodes)
head(UCB_lodes)
pdf(“1sankey.pdf”)
ggplot(UCB_lodes,
aes(x = x, stratum = stratum, alluvium = Cohort,
fill = stratum, label = stratum)) +
scale_x_discrete(expand = c(0, 0)) +
geom_flow(width = 1/8) + #线跟方块间空隙的宽窄
geom_stratum(alpha = .9,width = 1/10) + #方块的透明度、宽度
geom_text(stat = “stratum”, size = 3,color=”black”) + #文字大小、颜色
#不喜欢默认的配色方案,用前面自己写的配色方案
scale_fill_manual(values = mycol) +
xlab(“”) + ylab(“”) +
theme_bw() + #去除背景色
theme(panel.grid =element_blank()) + #去除网格线
theme(panel.border = element_blank()) + #去除外层边框
theme(axis.line = element_blank(),axis.ticks = element_blank(),axis.text = element_blank()) + #去掉坐标轴
ggtitle(“”)+
guides(fill = FALSE)
dev.off()
###还可以只选其中一部分列来画,此处只画第一列和第三列
UCB_lodes <- to_lodes_form(df[,c(1,3)],
axes = 1:2,
id = “Cohort”)
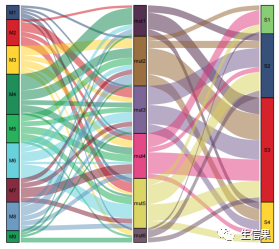
结果图展示
文末附代码和数据,关注小果,悄悄变强哦。

推荐阅读
-
生信分析中不可忽视的GTEx数据库
-
生信分析中的GSEA图谱看不懂?看这里
-
小果教你用blast批量比对蛋白核酸,不费劲
-
简单又实用的转录因子预测数据库ChEA3
-
教你解读WGCNA分析中的POWER图谱
原文始发于微信公众号(云生信学生物信息学):桑基图(Sankey diagram)