¥69.90
购买后会显示你购买的服务器的账号和密码。并且发送服务器的账号和密码到你的邮箱作为备份。格式为xxxx:zzzz (以“:”分割,前边是账号,后边是密码),例如 xiaoyun:998899 那么你的服务器的账号和密码分别是xiaoyun和998899。
有了账号和密码后,不会登录服务器的参考链接:http://www.biocloudservice.com/wordpress/?p=292
有问题咨询客服,客服微信:18502195490
描述
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
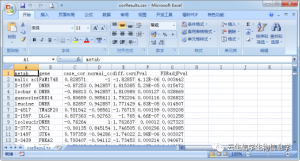
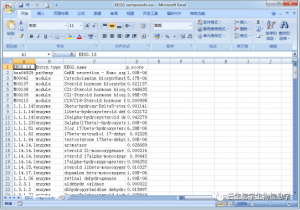
代谢物网络节点在KEGG数据库中的具体含义,KEGG.id:KEGG数据库中对应的ID号,Entry.type:类别,KEGG.name:名称,p.score:p值
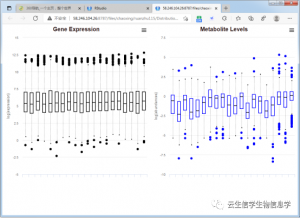
转录组、代谢组表达柱形图 横坐标为样本,纵坐标为表达量,左图为基因样本,右图为代谢组样本
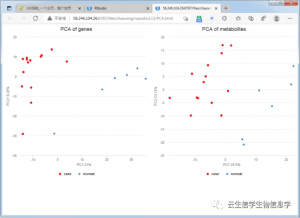
代谢物和基因表达数据主成分分析结果 左图为基因数据,右图为代谢组数据,红色圆形为xx样本,蓝色方形为xx样本
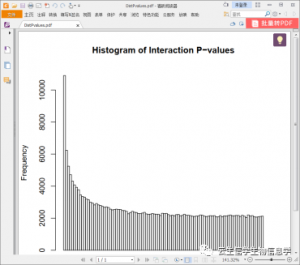
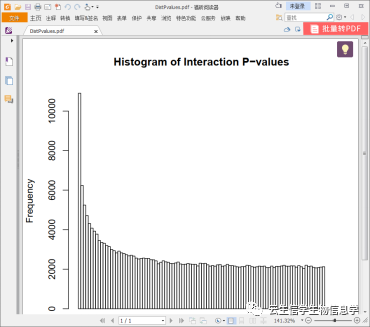
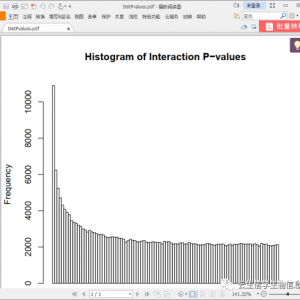
所有基因和代谢物关联性p值分布图 横坐标为关联性p值,纵坐标为该p值出现频率