¥69.90
购买后会显示你购买的服务器的账号和密码。并且发送服务器的账号和密码到你的邮箱作为备份。格式为xxxx:zzzz (以“:”分割,前边是账号,后边是密码),例如 xiaoyun:998899 那么你的服务器的账号和密码分别是xiaoyun和998899。
有了账号和密码后,不会登录服务器的参考链接:http://www.biocloudservice.com/wordpress/?p=292
有问题咨询客服,客服微信:18502195490
描述
PARAMETERS:
–path_file The KEGG pathway abundance table by PICRUSt2, input tsv table format.
–map_file the sample classification labels ,the first column is sample name which is consistent with path_file row in order, the second column is the classification labels with named “group“, input txt table format.
–case the name of the case, Default value is Tumor ,string.
–control the name of the control, Default value is Normal ,string.
–adj.p the DEpath threshold padj, Default value 0.05,string.
–logfc the DEpath threshold logFC, Default value 1,string.

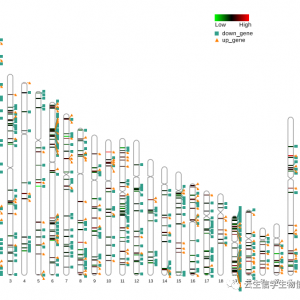
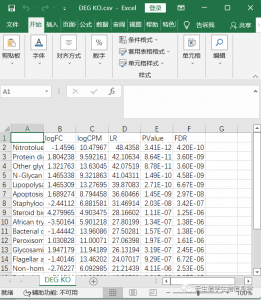
1. DEG KO.xls
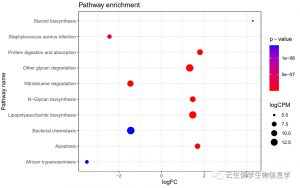
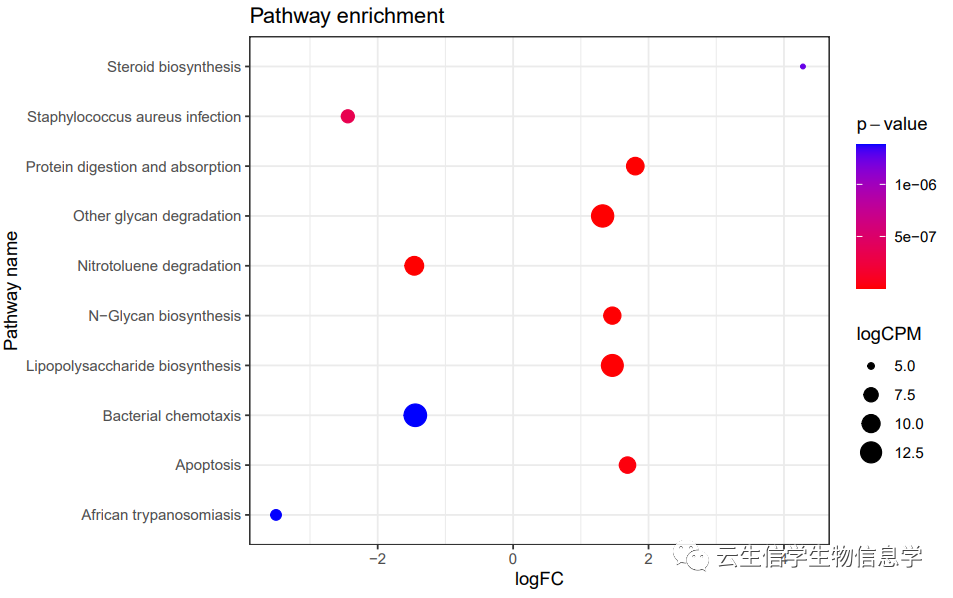
2. pathway top10.pdf