¥69.90
购买后会显示你购买的服务器的账号和密码。并且发送服务器的账号和密码到你的邮箱作为备份。格式为xxxx:zzzz (以“:”分割,前边是账号,后边是密码),例如 xiaoyun:998899 那么你的服务器的账号和密码分别是xiaoyun和998899。
有了账号和密码后,不会登录服务器的参考链接:http://www.biocloudservice.com/wordpress/?p=292
有问题咨询客服,客服微信:18502195490
描述
只需要基因表达谱、样本分组文件及通路富集结果,软件将自行计算出差异表达基因及相应的p.value值,同时挖掘出实验条件或疾病状态下显著偏差的通路以及对通路的显著偏差做出最大贡献的差异表达基因集合。
USAGE:
USAGE:
pathway_score.r –exp=–group=–lfc=–KEGG_pathway=
PARAMETERS:
–exp the matrix of gene expression ,input csv format.
–group the group of sampple ,input txt format,“group“ column must exsist.
–KEGG_pathway the KEGG pathway that DEGs were enriched,input txt format.
–lfc the cutoff of logFC.default:1
操作步骤:
1、打开命令行界面,输入“Rscript pathway_score.r”调阅帮助文档,确定该程序所需的输入文件。
2、用户根据帮助文档中的参数说明内容,对参数进行设置。这里,必须输入参数有3个,分别是–exp,表示基因表达矩阵文件,这里必须保存为csv格式;–group表示样本分组文件,输入格式为txt文本文件,并且必须包含group列且顺序一定要和基因表达矩阵列对应,相应的分组这里必须设置为“Tumor”,“Normal”,如需修改,需进入程序源代码进行相应修改。–KEGG_pathway表示通路富集结果文件,这里默认的是用clusterProfiler跑出来的结果,要用到p.adjust列。
diff.xls(所有基因差异分析结果)
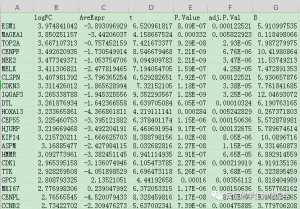
p.value:显著性p值
adj.P.Val:经过BH校正后P值
B:该基因差异表达的概率,越大,差异表达概率越大
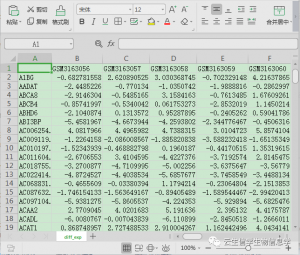
diff_exp.xls(按照所设阈值得到的差异基因表达矩阵文件)
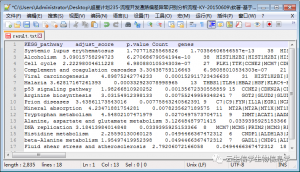
result.txt(通路偏差得分结果)
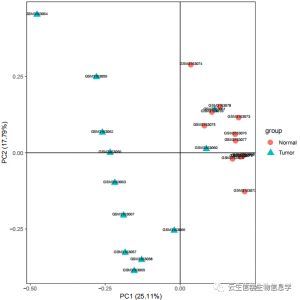
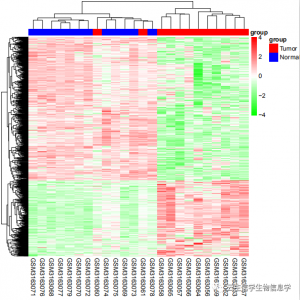
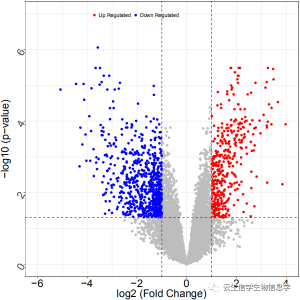
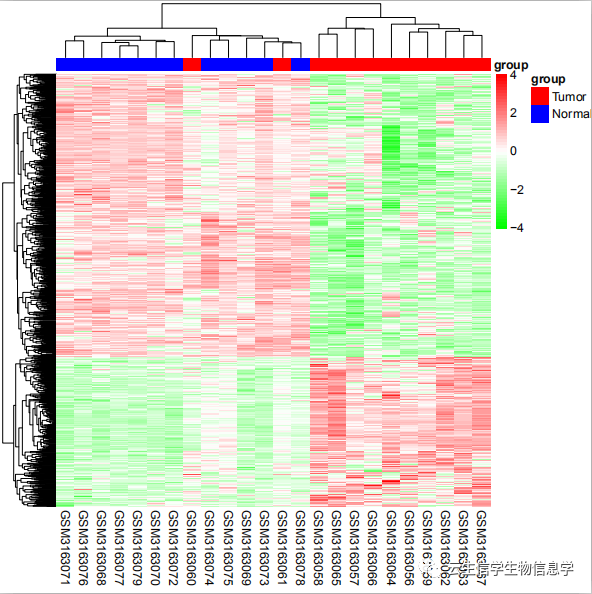
差异基因火山图(蓝色表示下调基因,红色表示上调基因,灰色表示差异不显著基因)
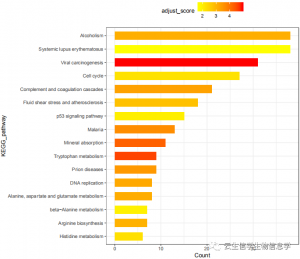
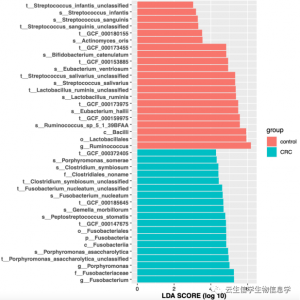
通路偏差得分富集结果图,条形长度表示最大贡献基因个数多少,纵坐标表示通路名称,颜色越深,表示偏离正常水平越明显