¥69.90
购买后会显示你购买的服务器的账号和密码。并且发送服务器的账号和密码到你的邮箱作为备份。格式为xxxx:zzzz (以“:”分割,前边是账号,后边是密码),例如 xiaoyun:998899 那么你的服务器的账号和密码分别是xiaoyun和998899。
有了账号和密码后,不会登录服务器的参考链接:http://www.biocloudservice.com/wordpress/?p=292
有问题咨询客服,客服微信:18502195490
描述
USAGE:
diffcorr.r –exp=–number_control=–gene_gene_cor=–module_module_cor=
PARAMETERS:
–exp exprmatrix input txt format.
–number_control the number of control sample.
–gene_gene_cor the Correlation coefficient between gene and gene,will be used for clustering,default:0.6.
–module_module_cor the Correlation coefficient between module in control and module in case,default:0.8.
操作步骤:
1、打开命令行界面,输入“Rscript diffcorr.r”调阅帮助文档,确定该程序所需的输入文件。
2、用户根据帮助文档中的参数说明内容,对参数进行设置。这里,必须输入参数有2个,分别是–exp,表示基因表达矩阵文件,这里必须保存为txt格式,并且属于同一个处理组的样本列必须挨着,这里对照组在前,处理组在后;–number_control表示对照组样本的个数。
可选参数有2个,分别为–gene_gene_cor,表示对各个表型下基于基因相关性进行聚类的阈值,默认为基因之间相关性大于0.6被聚为一个模块。–module_module_cor,表示对各个表型的模块进行相关性分析的阈值,这里默认为挖掘相关性大于0.8的两个模块。
3、完成参数提交后,按下回车键,整个程序即正式开始进入执行。每步执行内容都会给出提示。程序执行完毕后,界面会显示”Program execution is completed“结束语。
1、control_nerwork.pdf(对照组模块共表达网络)
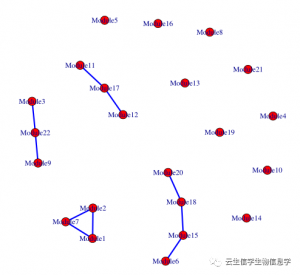
2、case_nerwork.pdf(处理组模块共表达网络)

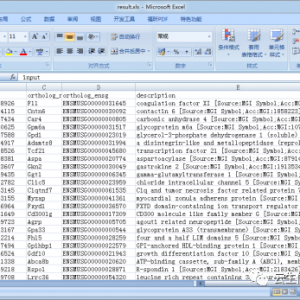
3、module_list_control.txt和module_list_case.txt(按照所设阈值得到的各个表型下的共表达模块及对应基因)
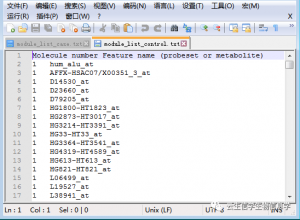
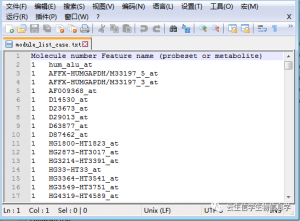
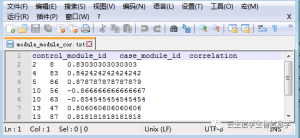
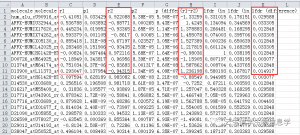
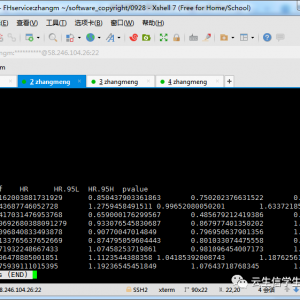
注:r1表示对照组中两个基因的相关系数
p (difference)表示相关性差异显著性p.value值
(r1–r2)表示相关性差异程度
lfdr (in cond. 1)表示校正后的对照组中两个基因的相关显著性FDR值
lfdr (in cond. 2)表示校正后的对照组中两个基因的相关显著性FDR值
lfdr (difference)表示校正后的相关性差异显著性FDR值
这里默认将FDR<0.05的差异共表达关系对导出